El modelo de los datos es el siguiente:
Variables independientes:
Black -> Black Mother -> Madre de color (1 si, 0 no)
Boy -> Baby Boy -> Bebé (1 niño, 0 niña)
CigsPerDay -> Cigarettes Per Day -> Número de Cigarrillos por día
Married -> Married Mother -> Madre casada (1 si, 0 no)
MomAge -> Mother's Age -> Edad de la madre (equivale el 0 a tener 25 años)
MomEdLevel -> Mother's Education -> Level Nivel de educación de la madre (de 0 a 3, siendo 0 nada)
MomSmoke -> Smoking Mother -> Madre fumadora (1 si, 0 no)
MomWtGain -> Mother's Preg Wght Gain -> Embarazo de la madre pérdida/aumento de peso
Visit -> Prenatal Visit -> Visita prenatal (de 0 a 3, siendo el 0 ninguna visita)
Variable dependiente u Objetivo:
weight -> Baby Weight -> Peso del Bebe
Para cargar los datos se pueden usar tanto los datos de suminsitrados por la practica como los de la libreria sashelp.bweight, ya que son los mismos.
data bwg;
set sashelp.bweight;
run;
Si hacemos un estudio de frecuencias de la variable objetivo (peso del bebe al nacer) vemos que no es una variable de clase sino una numérica y continua.
proc freq data=bwg;
tables weight;
run;
Con lo que analizamos la media, así como la gráfica y los test estadisticos para demostrar la normalidad.
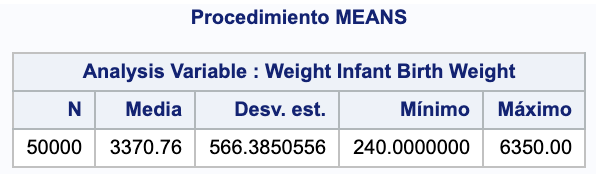
proc means data=bwg;
var weight;
run;
Vemos que tenemos 50000 observaciones con 10 variables.
/* NOTE: There were 50000 observations read from the data set SASHELP.BWEIGHT.
NOTE: The data set WORK.BWG has 50000 observations and 10 variables.
Analysis Variable : Weight Infant Birth Weight
N Media Desv. est. Mínimo Máximo
50000 3370.76 566.3850556 240.00 6350.00
*/;
proc gchart data=bwg;
vbar weight;
run;
/*Analisis Normalidad de la variable objetivo*/;
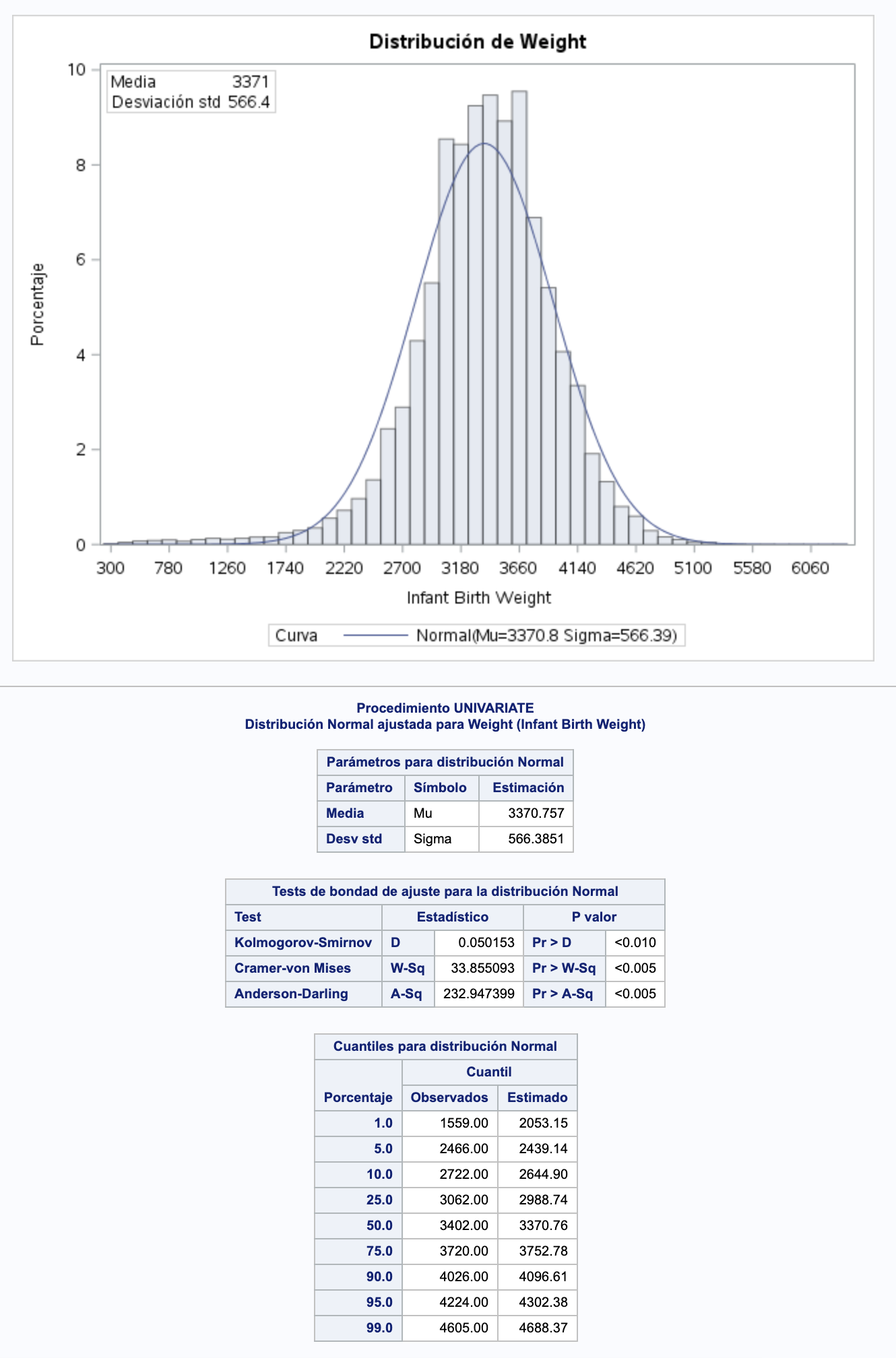
proc univariate data=bwg normal plot;
var weight;
HISTOGRAM /NORMAL(COLOR=MAROON W=4) CFILL = BLUE CFRAME = LIGR;
INSET MEAN STD /CFILL=BLANK FORMAT=5.2;
run;
La variable objetivo weight tiene un número alto de outliers en la parte inferior de los valores. Empiezo el estudio con todos los valores, pero lo tendré en cuenta en el caso de que sea necesario, para eliminarlos si llegase el caso.
Empezamos con 50000 observaciones y 10 variables. Podriamos eliminar duplicados con:
proc sort data=bwg out=B dupout=C nodupkey; By _all_ ; run;
Pero considero que no son datos duplicados, sino dos observaciones distintas de eventos distintos con exactamente los mismos valores en todas las variables. Puede parecer mucha cincidencia, pero si consideramos que gran parte de las variables son variables categoricas con poca variabilidad en sus posibilidades, basta con que se coincidan el peso del bebe y la ganancia del peso de la madre. Si estudiamos el conjunto C vemos que los valores del conjunto C (datos eliminados) son muy cercanos a la mediana, y creo que puedo dar por valido este razonamiento.
Si después de hablar con el suministrador de los datos este nos indica que SI hay variables duplicadas, podriamos seguir el estudio con el conjunto B.
Pero por el momento usamos el conjunto original.
La variable MomSmoke es 0 si la madre no fuma y 1 si lo hace. Además tenemos la variable numero de cigarrilos al dia. Antes de nada hacemos una comprobacion básica de si hay error en los datos y se ha indicado que una mujer fuma pero luego toma 0 cigarrillos al día o al contrario, la observacion dice que no fuma pero luego consume mas de 0 cigarrilos al día:
set bwg;
if cigsperDay = 0 and MomSmoke = 1 then alerta=1;
else
if cigsperDay> 0 and MomSmoke = 0 then alerta=2;
else alerta=0;
run;
proc freq data=bwgDummy;
run;
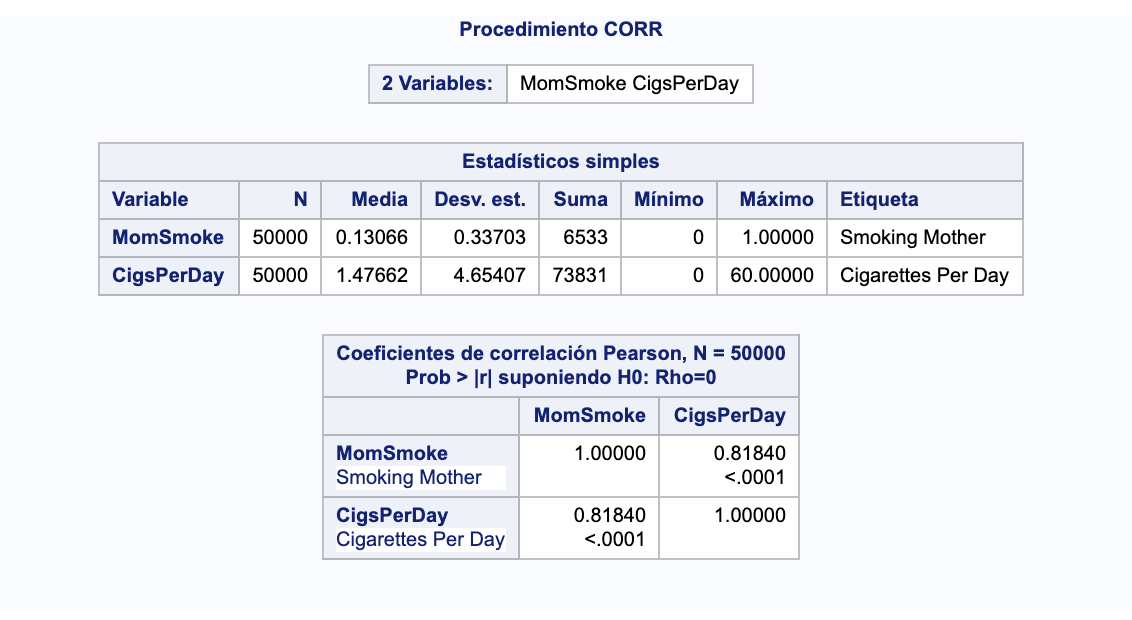
La simple lógica nos dice que estas variables tienen que estar relacionadas, pero aun así realizamos un estudio de correlación entre ambas:
proc corr data=bwg;
var MomSmoke cigsperday;
run;
Podemos eliminar una de las dos del conjunto de datos a estudiar. Ya que CigsPerDayda más datos que MomSmoke, eliminamos MomSmoke.
Además, la variable MomAge está referida a 25 años, para que el estudio sea más sencillo se cambia la variable a su valor real
data bwgC (drop = momAge momSmoke);
set bwg;
realMomAge = momAge + 25;
run;
Con:
ods graphics on;
proc ttest data=bwgc;
var _numeric_;
run;
ods graphics off;
realizamos un TTest de todas las variables numericas ( PDF con resultados de TTest )
Paso a estudiar cada variable:
proc freq data=bwgc;
table black;
run;
| Black | Frecuencia | Porcentaje | Frecuencia acumulada | Porcentaje acumulado |
|---|---|---|---|---|
| 0 | 41858 | 83.72 | 41858 | 83.72 |
| 1 | 8142 | 16.28 | 50000 | 100.00 |
No faltan valores (no hay missings) y se puede usar para el modelo. Es una variable categorica, y no puede ser ajustada a una normal.
proc freq data=bwgc;
table married;
run;
| Married | Frecuencia | Porcentaje | Frecuencia acumulada | Porcentaje acumulado |
|---|---|---|---|---|
| 0 | 14369 | 28.74 | 14369 | 28.74 |
| 1 | 35631 | 71.26 | 50000 | 100.00 |
Este caso es similar al anterior. Es una variable dicotomica.
proc freq data=bwgc;
table boy;
run;
| Boy | Frecuencia | Porcentaje | Frecuencia acumulada | Porcentaje acumulado |
|---|---|---|---|---|
| 0 | 24208 | 48.42 | 24208 | 48.42 |
| 1 | 25792 | 51.58 | 50000 | 100.00 |
No hay ningun missing, es una variable dicotomica y al 50% aprox.
proc freq data=bwgc;
table CigsPerDay;
run;
| CigsPerDay | Frecuencia | Porcentaje | Frecuencia acumulada | Porcentaje acumulado |
|---|---|---|---|---|
| 0 | 43467 | 86.93 | 43467 | 86.93 |
| 1 | 206 | 0.41 | 43673 | 87.35 |
| 2 | 309 | 0.62 | 43982 | 87.96 |
| 3 | 387 | 0.77 | 44369 | 88.74 |
| ... | .... | ... | ... | ... |
| 50 | 4 | 0.01 | 49998 | 100.00 |
| 60 | 2 | 0.00 | 50000 | 100.00 |
De nuevo no faltan valores, pero vemos que el casi el 87% de las observaciones son de no fumadoras, y sólo el 13% restante es de fumadoras. Lo podemos usar para el modelo, pero al ser tan pocos relativamente, no sé seguro cuanto puede afectar al mismo. Por sentido común el hecho de que fume la madre debería afectar al peso del bebe, pero hay que estudiarlo.
proc freq data=bwgc;
table MomAge;
run;
| MomWtGain | Frecuencia | Porcentaje | Frecuencia acumulada | Porcentaje acumulado |
|---|---|---|---|---|
| -30 | 598 | 1.20 | 598 | 1.20 |
| -29 | 56 | 0.11 | 654 | 1.31 |
| ... | .... | ... | ... | ... |
| 64 | 1 | 0.00 | 49977 | 99.95 |
| 66 | 1 | 0.00 | 49978 | 99.96 |
| 68 | 22 | 0.04 | 50000 | 100.00 |
No faltan ningun valor, lo podemos usar para el modelo. Pero presenta una distribución extraña, habiendo muchos más valores concentrados en multiplos de 5. Probablemente sea un error a la toma de los datos en el sentido de que se han redondeado. Por ello voy a generar otro conjunto de datos con los valores en grupos de 5 libras y realizaré el estudio con ambos modelos.
proc freq data=bwg;
table Visit;
run;
| Visit | Frecuencia | Porcentaje | Frecuencia acumulada | Porcentaje acumulado |
|---|---|---|---|---|
| 0 | 403 | 0.81 | 403 | 0.81 |
| 1 | 6339 | 12.68 | 6742 | 13.48 |
| 2 | 1114 | 2.23 | 7856 | 15.71 |
| 3 | 42144 | 84.29 | 50000 | 100.00 |
No faltan valores y se puede usar para el modelo. Es una variable con 4 categorias distintas.
proc freq data=bwg;
table MomEdLevel;
run;
| MomEdLevel | Frecuencia | Porcentaje | Frecuencia acumulada | Porcentaje acumulado |
|---|---|---|---|---|
| 0 | 17449 | 34.90 | 17449 | 34.90 |
| 1 | 12129 | 24.26 | 29578 | 59.16 |
| 2 | 12449 | 24.90 | 42027 | 84.05 |
| 3 | 7973 | 15.95 | 50000 | 100.00 |
No faltan valores y se puede usar para el modelo. Es una variable con 4 categorias distintas.
proc freq data=bwg;
table MomAge;
run;
| MomAge | Frecuencia | Porcentaje | Frecuencia acumulada | Porcentaje acumulado |
|---|---|---|---|---|
| 16 | 1824 | 3.65 | 1824 | 3.65 |
| 17 | 2420 | 4.84 | 4244 | 8.49 |
| 18 | 2588 | 5.18 | 6832 | 13.66 |
| 19 | 2521 | 5.04 | 9353 | 18.71 |
| 20 | 2590 | 5.18 | 11943 | 23.89 |
| ... | .... | ... | ... | ... |
| 42 | 44 | 0.09 | 49974 | 99.95 |
| 43 | 26 | 0.05 | 50000 | 100.00 |
No faltan ningun valor, lo podemos usar para el modelo. La distribución no es normal en su cola de ascenso, se aproxima bastante en su cola de descenso. Hay que tener en cuenta que la edad fertil de la mujer es muy dependiente de su biologia, y aunque en edades tempranas es más probable que se quede embarazada (si no pone medios en contra), mientras que según pasa el tiempo influyen otros valores.
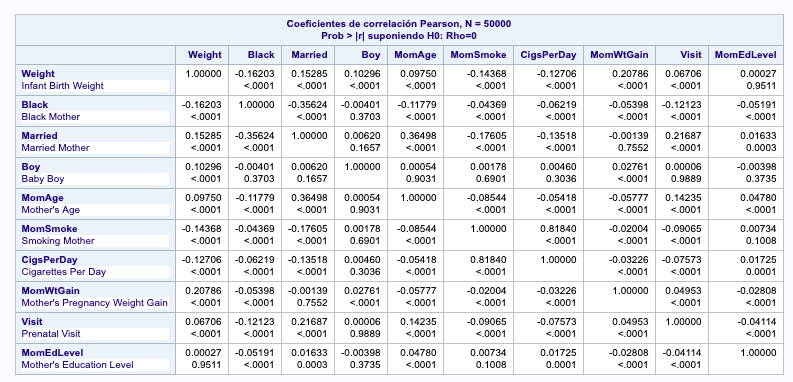
proc corr data=bwg;
run;
/* No hay ninguna variable correlada */;
No existen variables con un valor alto de correlación, con lo que damos este paso por concluido.
A partir de este momento tenemos dos datasets limpios a priori bwgC y bwgW, el primero con la edad de la madre corregido y el segundo con la ganancia de peso agrupada en cada 5 kilos para evitar los errores en la toma de datos (explicado previamente.
Con estos datasets vamos a intentar generar un modelo.
Para ello usamos el GLMSELECT, usando todas las variables como input y todas sus posibles combinaciones para que el procedimiento elija las más significativas
proc glmselect data=bwgC;
class Black Boy Married MomEdLevel Visit;
model weight = Black Boy Married MomEdLevel Visit RealMomAge CigsPerDay MomWtGain
Black*Boy Black*Married Black*MomEdLevel Black*Visit Black*RealMomAge Black*CigsPerDay Black*MomWtGain
Boy*Married Boy*MomEdLevel Boy*Visit Boy*RealMomAge Boy*CigsPerDay Boy*MomWtGain
Married*MomEdLevel Married*Visit Married*RealMomAge Married*CigsPerDay Married*MomWtGain
MomEdLevel*Visit MomEdLevel*RealMomAge MomEdLevel*CigsPerDay MomEdLevel*MomWtGain
Visit*RealMomAge Visit*CigsPerDay Visit*MomWtGain
CigsPerDay*MomWtGain
/selection=stepwise;
run;
Hacemos esto para ambos modelos bwgC y bwgW
y luego estudiamos los efectos
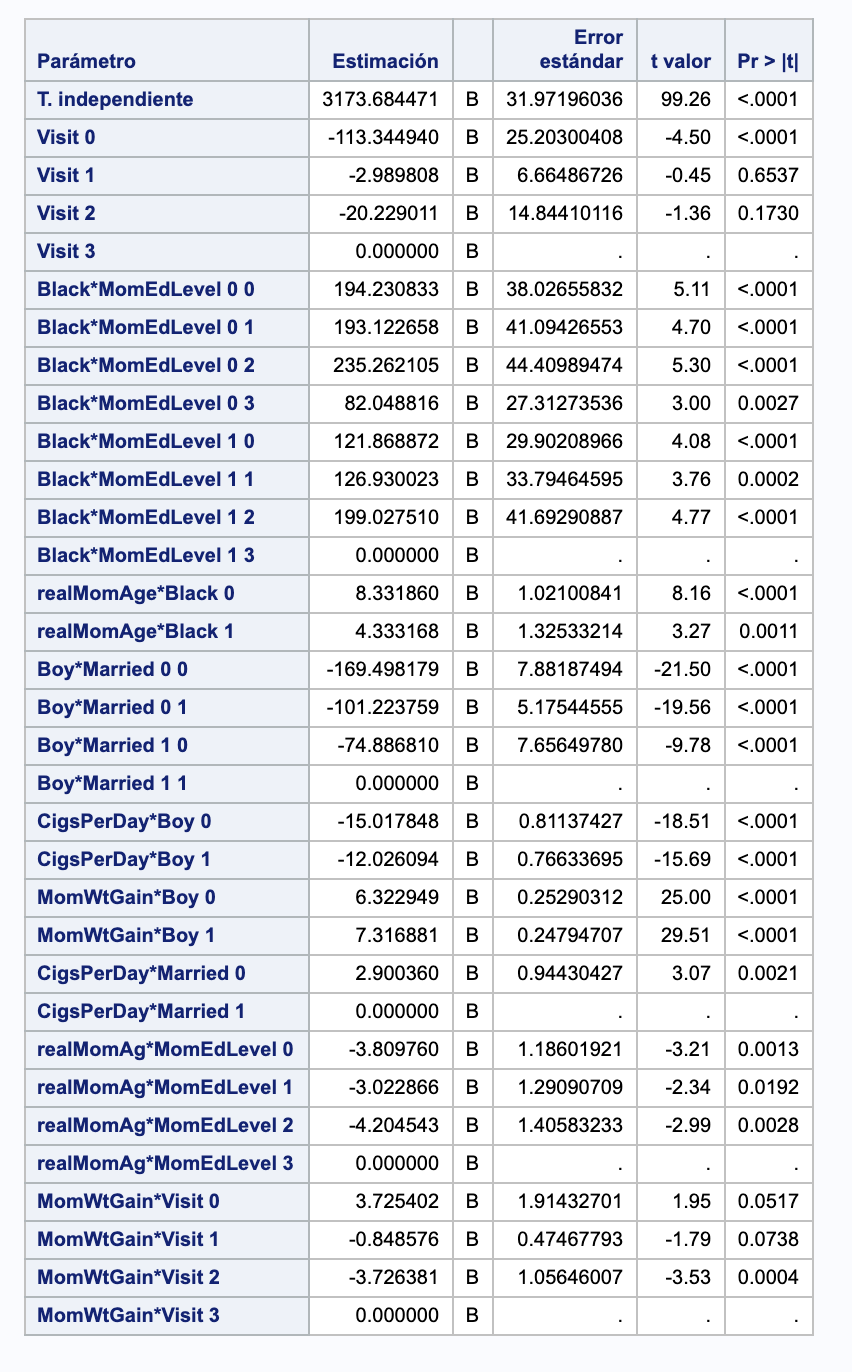
proc glm data=bwgC;
class Black Boy Married MomEdLevel Visit;
model weight = Boy MomEdLevel Black*Married realMomAge*Black CigsPerDay*Boy MomWtGain*Boy MomWtGain*Visit /solution e;
run;
Eliminando interacciones hasta que no vemos ninguna con un error tipo III con valores de P valor superior a 0.05, y saco el coeficiente de correlación.
Además, me surge la duda de si la variable visit puede considerarse una variable de clase o no. A priori, considero que no, que es una variable puramente numerica, pero realizo el estudio con ambas posibilidades.
De estos supuestos saco los siguientes modelos
| Dataset | class visit | Modelo | R^2 |
|---|---|---|---|
| bwgC | Si | Boy MomEdLevel BlackMarried realMomAgeBlack CigsPerDayBoy MomWtGainBoy MomWtGain*Visit | 0.105719 |
| bwgC | No | Boy MomEdLevel BlackMarried realMomAgeBlack CigsPerDayBoy MomWtGainBoy Visit*CigsPerDay | 0.104320 |
| bwgW | Si | Boy MomEdLevel BlackMarried realMomAgeBlack CigsPerDayBoy pesoAgrupadoBoy pesoAgrupado*Visit | 0.104320 |
| bwgW | No | Boy MomEdLevel BlackMarried realMomAgeBlack CigsPerDayBoy pesoAgrupadoBoy Visit*CigsPerDay | 0.103479 |
Los valores de R^2 que obtenemos son muy bajos, siendo el mejor bwgC y con visit como variable de clase.
Esto me hace sospechar que tenemos algun fallo en los datos de entrada, y que puede que sea necesario mejorar los datasets iniciales.
Pese a ello, y por concluir el estudio, genero unas macros para hacer el estudio con distintas semillas y distinto metodo para realizar distintas simulaciones
%let libMSV = '/home/u38083750/unaiherran/Practica1/Output/resultados01.txt';
/*Macro Sin Agrupar MomWTGain y Visit class*/;
%macro macroSV (semi_ini, semi_fin, metodo);
%do semilla=&semi_ini. %to &semi_fin.;
ods graphics on;
ods output SelectionSummary=modelos;
ods output SelectedEffects=efectos;
ods output Glmselect.SelectedModel.FitStatistics=ajuste;
proc glmselect data=bwgC plots=all seed=&semilla;
partition fraction(validate=0.2);
class Black Boy Married MomEdLevel Visit;
model weight = Black Boy Married MomEdLevel Visit RealMomAge CigsPerDay MomWtGain
Black*Married realMomAge*Black CigsPerDay*Boy MomWtGain*Boy MomWtGain*Visit
/ selection=&metodo.(select=aic choose=validate) details=all stats=all;
run;
ods graphics off;
ods html close;
data union; i=12; set efectos; set ajuste point=i; run; *observación 12 ASEval;
data _null_;
semilla=&semilla;
metodo=&metodo.;
file &libMSV mod;
set union;put effects "," nvalue1 "," semilla ", &metodo., SV";run;
proc sql; drop table modelos,efectos,ajuste,union; quit;
%end;
%mend;
- macroSV (dataset sin agrupar y con visit como variable de clase)
- macroS (dataset sin agrupar y sin visit como variable de clase)
- macroAV (dataset agrupado y con visit como variable de clase)
- macroA (dataset agrupado y sin visit como variable de clase)
Y lo ejecutamos muchas veces con distintas semillas y distintos metodos.
%macroSV (12300,12350, stepwise);
%macroS (12300,12350, stepwise);
%macroAV (12300,12350, stepwise);
%macroA (12300,12350, stepwise);
%macroSV (12300,12350,, Backward);
%macroS (12300,12350, Backward);
%macroAV (12300,12350, Backward);
%macroA (12300,12350, Backward);
%macroSV (12300,12350, Forward);
%macroS (12300,12350, Forward);
%macroAV (12300,12350, Forward);
%macroA (12300,12350, Forward);
Los resultados los guardamos en ( resultados01.txt )
Despues de realizar 560 simulaciones, evaluamos los distintos modelos elegidos y su ASEVAL
proc import datafile = '/home/u38083750/unaiherran/Practica1/Output/resultados01.txt'
out = resultado
dbms = dlm
replace;
delimiter = ',';
getnames = no;
run;
data resultado_clean;
set resultado;
rename VAR1 = modelo
var2 = ASEEval
var3 = semilla
var4 = metodo
var5 = dataset;
run;
proc sort data=resultado_clean; by modelo;
proc freq data=resultado_clean;
Hay un modelo que se repite en multitud de ocasiones Intercept Boy MomEdLevel Black*Married realMomAge*Black CigsPerDay*Boy MomWtGain*Boy, pero los valores de ASEVAL son muy altos (entre 275810 y 298125) y creo que es necesario seguir dandole alguna vuelta.
En el estudio previo hemos considerado todos los datos del dataset original. Puede que los outliers en la variable objetivo distorsionen el resultado del modelo. Con lo que voy a realizar un nuevo estudio del mismo para intentar eliminarlo y mejorar el modelo.
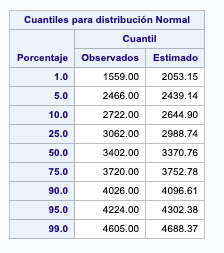
proc univariate data=bwgC normal plot;
var weight;
HISTOGRAM /NORMAL(COLOR=MAROON W=4) CFILL = BLUE CFRAME = LIGR;
INSET MEAN STD /CFILL=BLANK FORMAT=5.2;
run;
Eliminamos las observaciones extremas
data bwgCnO;
set bwgC;
if weight <= 1559 or weight >= 4605 then delete;
run;
y repetimos las macros para los nuevos datasets, obteniendo resultados02.txt
Analizando estos resultados vemos varios modelos que se repiten varias veces y que los valores de ASEeval han bajado.(217588-232091) siguen siendo muy altos.
Los modelos a estudiar son:
| Modelo | R^2 |
|---|---|
| Visit BlackMomEdLevel realMomAgeBlack CigsPerDayBlack BoyMarried CigsPerDayBoy MomWtGainBoy realMomAgeMarried CigsPerDayMarried realMomAgeMomEdLevel MomWtGainMomEdLevel CigsPerDayVisit MomWtGainVisit | 0.098710 |
Una vez optimizado y quitando los valores con un P Valor alto, el modelo que mejor resultado ha tenido es:
(weight = Visit BlackMomEdLevel realMomAgeBlack BoyMarried CigsPerDayBoy MomWtGainBoy CigsPerDayMarried realMomAgeMomEdLevel MomWtGainVisit)
Este modelo tiene un R^2 peor que sin los outliers en cuanto al r^2 y mejor en el ASEVAL, pero ambos valores, y ambos modelos no parecen ser muy predictivos ya que tienen mucho error aleatorio. Probablemente sea necesario obtener alguna otra variable y repetir el estudio, para encontrar valores de error menores.
En cualquier caso, y sabiendo que el modelo se puede mejorar, paso a hacer el estudio con el Dataminer, a ver si aplicando algun otro modelo distinto en lugar del GLM podemos mejorar el resultado.
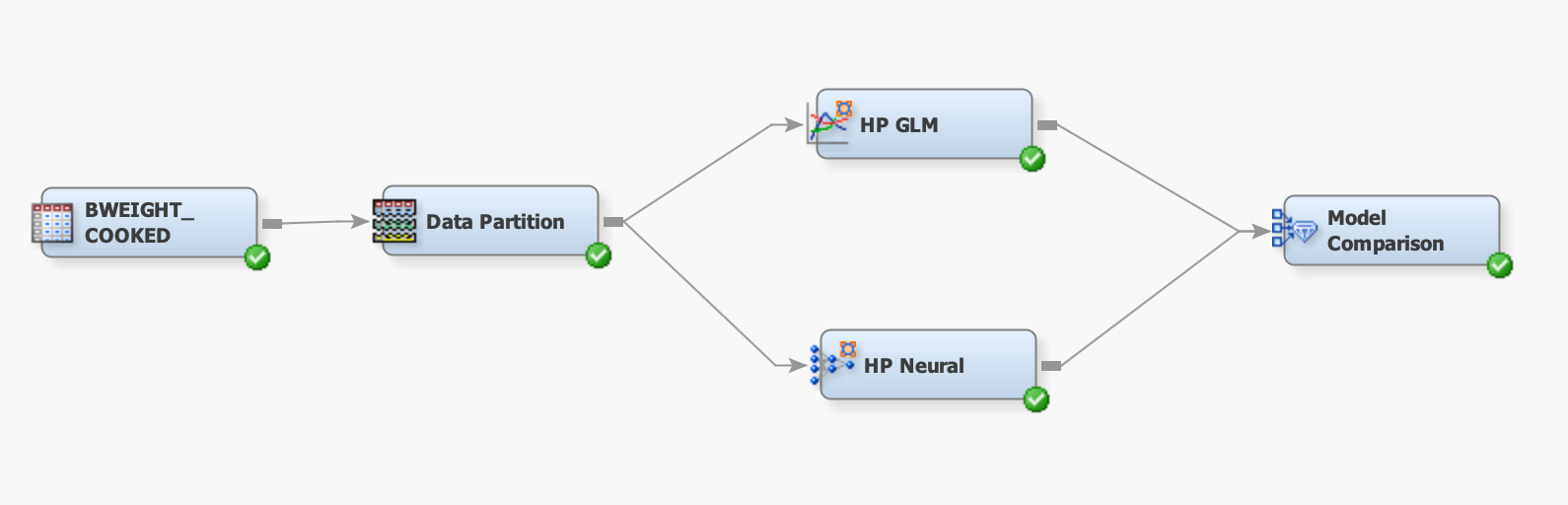
En Sas Studio exportamos el dataset con el que hemos trabajado, a un a librería ya 'cocinada'
data lib_prac.bweight_cooked;
set bwgCno;
run;
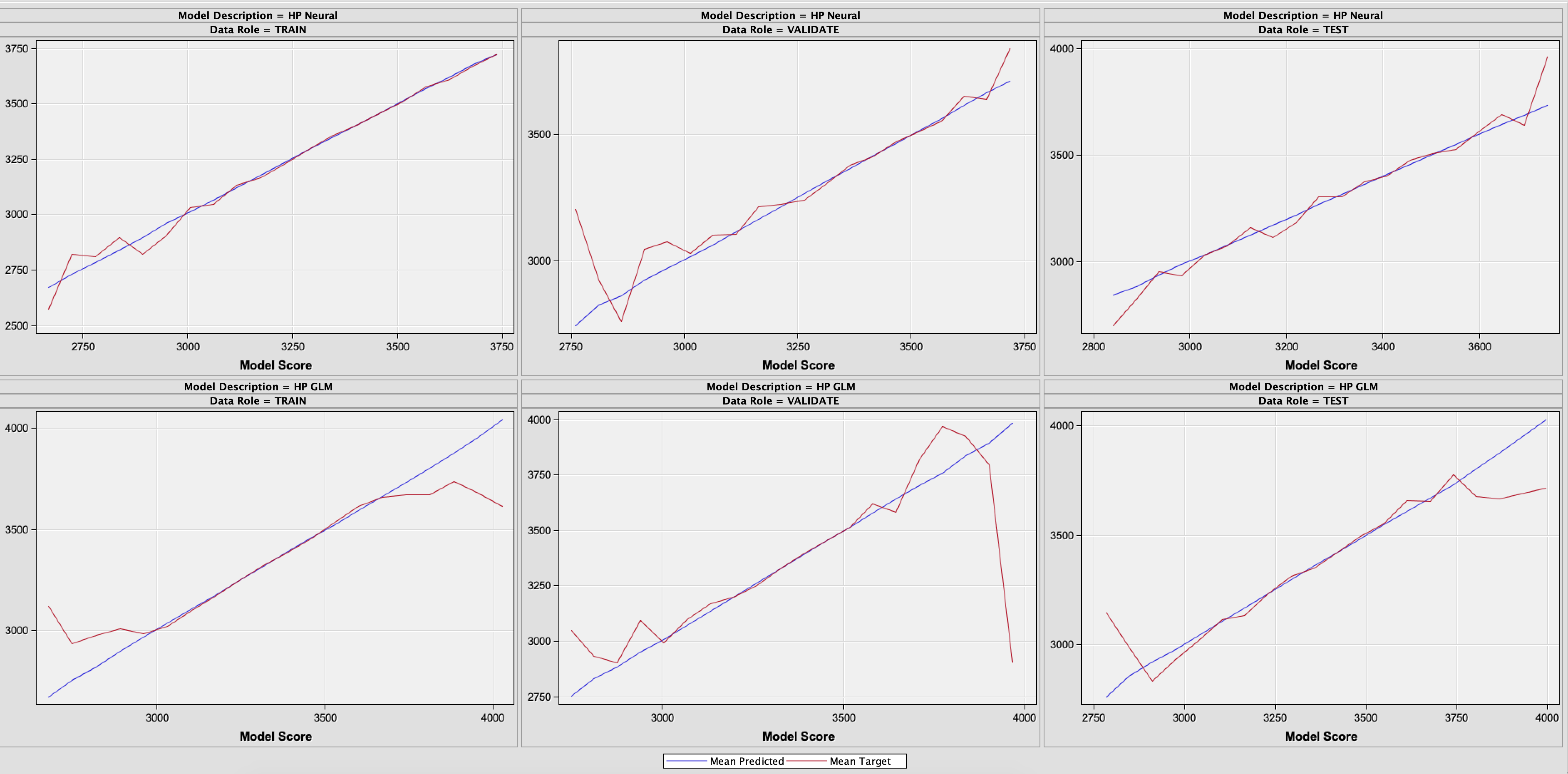
E importamos esta libreria en el Data Miner, partimos los datos en Training 70, Validation 15, Test 15 y los introducimos en dos modelos distintos, un GLM y una red neuronal, para despues comparar los modelos.
y vemos que tanto en los gráficos como en los valores el modelo elegido es la Red Neuronal. Pese a todo, y como se ha indicado previamente, el valor de error cuadratico medio (ASE) es muy elevado, y el modelo tendría que mejorarse.