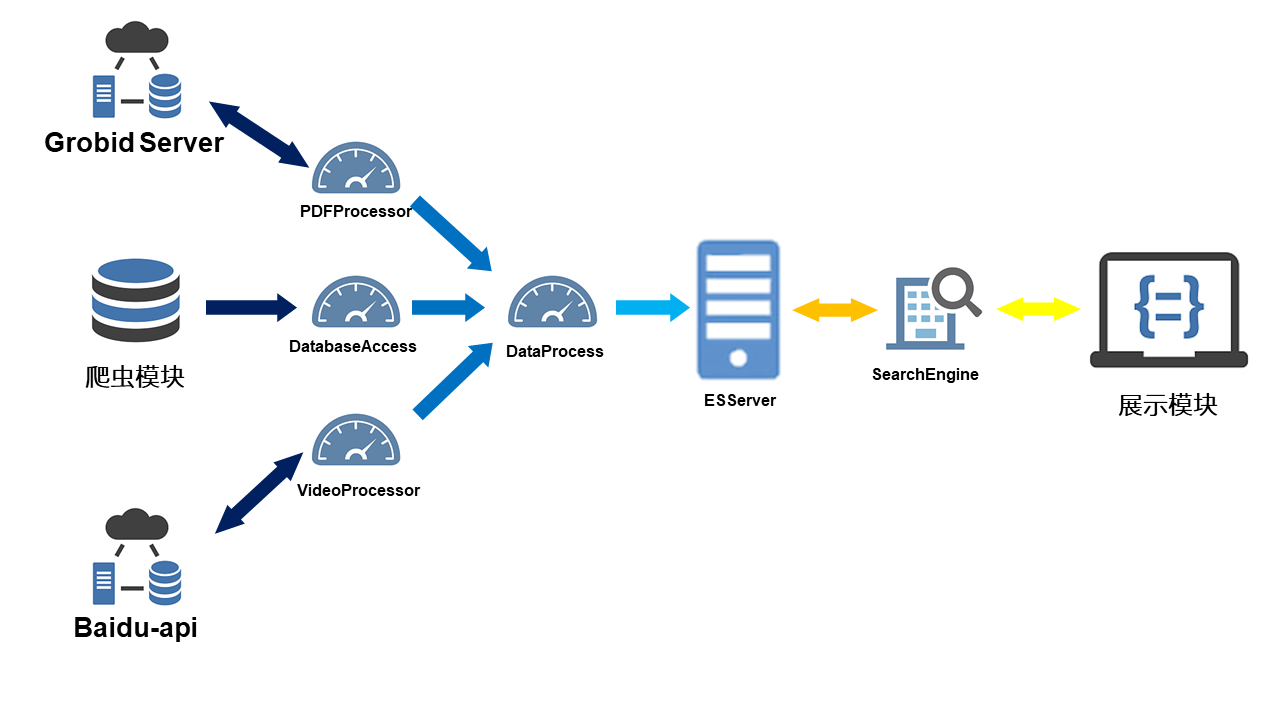
一款支持增量式数据更新的科技论文检索系统
- 一行代码即可完成服务端部署
- 一行代码即可完成客户端安装
- 简易的客户端接口使用
- 系统增量式动态更新
- 可同时与多个爬虫模块实现交互
- 检索系统快速恢复
- 检索系统支持多视频内容同时搜索定位
- PDF多线程并发解析
- PDF和视频文件采用“线下处理+线上取用”方案,复用数据解析结果,利于系统的快速响应
在接口约定方面,我们以数据流的形式为基础,通过json的格式实现了接口约定:
-
组内接口约定
- toES.json: 该文件定义了数据在elasticsearch内索引中的存储结构
-
组间接口约定
- fromCrawler.json: 该文件定义了从爬虫模块MongoDB server中取出来的数据结构
- fromWebsite.json: 该文件定义了展示模块发出请求的数据类型
- toWebsite.json: 该文件定义了向展示模块反馈的数据类型
| 组名 | 组长 | 分工 |
|---|---|---|
| HandsomeGuys | 方致远 | 从各个论文网站上爬取多模态数据,将pdf和视频等磁盘文件按增量批次传递给检索模块,并将相关元数据同步更新给MongeDB服务 |
| ICanOnlyCrawl | 何亮丽 | 从各个论文网站上爬取多模态数据,将pdf和视频等磁盘文件按增量批次传递给检索模块,并将相关元数据同步更新给MongeDB服务 |
| ratTailCollagen1 | 聂宇翔 | 接收来自于爬虫模块的增量更新,并进行处理,将处理后的结果放入elasticsearch服务的相应索引中,并为展示组提供数据展示的接口 |
| day2night | 刘雨程 | 调用检索模块的接口,展示检索的结果 |
如下所示,为我们项目的组织情况。其中ScienceSearcher为我们的包名。其中第一层存储了所有的功能类。在第二层中grobid-client中放置了访问grobid server相关的所有功能模块。data文件夹里放置的是cache和dataFlow文件夹,其中dataFlow文件夹中存放了所有的接口数据类型约定信息,而cache文件夹中存放了所有的硬盘文件,它们首先按(爬虫)组进行了划分,在组文件夹的内部,又按照批次进行了划分,在每一批次内部有两个文件夹PDFs和videos,分别存放pdf文件和视频文件。
.
├── images
└── ScienceSearcher
├── data
│ ├── cache
│ │ ├── demo
│ │ │ ├── 1
│ │ │ │ ├── PDFs
│ │ │ │ ├── videos
│ │ │ │ ├── videosstruct
│ │ │ │ └── XMLs
│ │ │ └── 2
│ │ │ ├── PDFs
│ │ │ ├── videos
│ │ │ ├── videosstruct
│ │ │ └── XMLs
│ │ ├── fzy
│ │ │ ├── 1
│ │ │ │ ├── PDFs
│ │ │ │ ├── videos
│ │ │ │ ├── videosstruct
│ │ │ │ └── XMLs
│ │ │ └── 2
│ │ │ ├── PDFs
│ │ │ ├── videos
│ │ │ ├── videosstruct
│ │ │ └── XMLs
│ │ └── hll
│ │ └── 1
│ │ ├── PDFs
│ │ ├── videos
│ │ └── XMLs
│ └── dataFlow
└── grobid_client注:下列服务可在同一台服务器上部署,也可在不同服务器上分别部署
- 在一台服务器上部署MongoDB服务,并获取访问该MongoDB服务所需要的
USER,PASSWORD,ADDRESS,PORT,SERVICE_NAME以及COLLECTION_NAME。 - 在一台服务器上部署Elasticsearch服务,并获取访问该服务所需要的
ES_IP和ES_PORT。 - 在一台服务器上部署Grobid Server,并获取该服务所在的IP地址
PDF_IP以及端口PDF_PORT。
elasticsearch==7.10.1
baidu_aip==2.2.18.0
pymongo==3.11.2
ipdb==0.13.4
Flask==1.1.2
requests==2.14.2
pydub==0.24.1
SpeechRecognition==3.8.1
wit==6.0.0
moviepy==1.0.3推荐采用Linux系统,您也可以选择Windows系统来部署。
首先从github上clone我们的项目,并安装依赖包
# If you want to obtain the latest version of our package, please clone the repository
git clone https://github.com/BITCS-Information-Retrieval-2020/search-rattailcollagen1.git
cd search-rattailcollagen1
# Make sure you have the versions of dependencies in requirements.txt
pip install -r requirements.txt然后通过一行代码进行服务端的部署:
python run.py --mode process --pdf_ip PDF_IP --pdf_port PDF_PORT --mongodb_service_path mongodb://USER:PASSWORD@ADDRESS:PORT/SERVICE_NAME --mongodb_service_name SERVICE_NAME --mongodb_collection_name COLLECTION_NAME --es_ip ES_IP --es_port ES_PORT --es_index_name ES_INDEX_NAME --cache_name GROUP_NAME您可以选择使用shell文件来保存该行命令,从而省去手动输入代码的麻烦。然后将该命令中的大写名称替换为相应的数值。
名称说明如下:
| 名称 | 说明 |
|---|---|
| USER | 连接数据库的用户名 |
| PASSWORD | 连接数据库的密码 |
| ADDRESS | 数据库所在服务器IP地址 |
| PORT | 数据库所在服务器端口号 |
| SERVICE_NAME | 数据库名称 |
| COLLECTION_NAME | 文档集合名称 |
| ES_IP | Elasticsearch所在服务器的IP地址 |
| ES_PORT | Elasticsearch所在服务器的端口号 |
| ES_INDEX_NAME | 所使用的Elasticsearch索引名称 |
| PDF_IP | Grobid Server所在服务器的IP地址 |
| PDF_PORT | Grobid Server所在服务器的端口号 |
| GROUP_NAME | 所对应的爬虫组名称 |
| SERVER_IP | 部署该系统的服务器的IP地址 |
| SERVER_PORT | 部署该系统的服务器的端口号 |
| DOWNLOAD_PORT | 客户端接收数据的端口号 |
其中,当部署前的准备工作做好之后,用户只需要自行拟定ES_INDEX_NAME和GROUP_NAME两项即可。
这两项指定了我们的系统所要服务的crawler是哪一个。如代码目录结构一节中所示,其中/ScienceSearcher/data/cache/fzy,/ScienceSearcher/data/cache/fzy和/ScienceSearcher/data/cache/hll为我们建立的三个示例文件夹,它们服务于三个爬虫模块。您也可以选择建立其他的为爬虫服务的文件夹。如果我们的本次的运行服务于爬虫demo,那么我们需要将ES_INDEX_NAME指定为demo_papers, 并将GROUP_NAME指定为demo。
只需要一行代码即可以实现客户端安装。
pip install ScienceSearcher -i https://pypi.python.org/simple客户端使用的一个简单例子(以demo爬虫系统为例,完整的使用示例参见example.py)为:
from ScienceSearcher.SearchEngine import SearchEngine
# initialization
se = SearchEngine(download_server_ip='SERVER_IP',
download_server_port=SERVER_PORT,
download_client_ip='0.0.0.0',
download_client_port=DOWNLOAD_PORT,
es_ip='ES_IP',
es_port=ES_PORT,
index_name='demo_papers',
video_index_name='demo_papers_video',
group_name='demo')
# 全文检索
query = {
"type": 0,
"top_number": 10,
"query_text": "Regularisation Methods"
}
res = se.search(query)
# 限于篇幅,我们将查询结果移至下文中“模块整体效果”一节的“全文检索”中
# 得到的结果为字典类型,此处直接展示出了其键值对
# 部分查询结果:
# title:Understanding Regularisation Methods for Continual Learning @ICML CL Workshop
# .
# .
# .
# 高级检索
query = {
"type": 1,
"top_number": 1,
"query_text": {
"title": "",
"authors": "Hasib Zunair",
"abstract": "",
"content": ""
},
"operator": ["", "OR", "", ""]
}
res = se.search(query)
# 限于篇幅,我们将查询结果移至下文中“模块整体效果”一节的“高级检索”中
# 部分查询结果:
# title:MICCAI-PRIME 2020 - Uniformizing Techniques to Process CT scans with 3D CNNs for TB Prediction
# authors:Hasib Zunair
# .
# .
# .
# 根据内容定位视频
query = {
"type": 2,
"top_number": 10,
"query_text": "i want"
}
res = se.search(query)
# 限于篇幅,我们将查询结果移至下文中“模块整体效果”一节的“根据内容定位视频”中
# 部分查询结果:
# title:X-Caps: Encoding Visual Attributes in Capsules for Explainable Medical Diagnoses | MICCAI 2020
# .
# .
# .
# videoStruct:[{'timeStart': '00-03-38', 'timeEnd': '00-03-42', 'sentence': 'and if we want our models to be explainable at the human level'}]
# 根据id进行搜索
id_ = 0
res = se.search_by_id(id_)
# 限于篇幅,我们将查询结果移至下文中“模块整体效果”一节的“根据id进行搜索”中
# 部分查询结果:
# title:Let's Stop Incorrect Comparisons in End-to-end Relation Extraction! (EMNLP 2020)
# .
# .
# .
# _id:0
# 根据关键词查询标题
query = "learn"
res = se.auto_complete(query)
# 限于篇幅,我们将查询结果移至下文中“模块整体效果”一节的“根据关键词查询标题”中
# 查询结果为列表类型,此处直接展示标题
# 部分查询结果:
# ICML 2020: Learning De-biased Representations with Biased Representations
# .
# .
# .本模块的整体功能即为上文客户端使用中所展示的例子,所以效果展示为对应的top1结果的完整内容
- 全文检索
title:Understanding Regularisation Methods for Continual Learning @ICML CL Workshop
authors:Frederik Benzing
abstract:
publicationOrg:ICML
year:2020
pdfUrl:https://arxiv.org/pdf/2006.06357.pdf
pdfPath:G:\search-rattailcollagen1\ScienceSearcher/data/cache/demo/2/PDFs/UnderstandingRegularisationMethodsforContinualLearningICMLCLWorkshop.pdf
publicationUrl:https://arxiv.org/pdf/2006.06357.pdf
codeUrl:https://github.com/freedbee/continual_regularisation
datasetUrl:
videoUrl:https://www.youtube.com/embed/GHV302OC2Ns
videoPath:G:\search-rattailcollagen1\ScienceSearcher/data/cache/demo/2/videos/Understanding Regularisation Methods for Continual Learning @ICML CL Workshop.mp4
pdfText:Despite much progress, there remain many gaps between artificial and biological intelligence. Among ...
_id:21
- 高级检索
title:MICCAI-PRIME 2020 - Uniformizing Techniques to Process CT scans with 3D CNNs for TB Prediction
authors:Hasib Zunair
abstract:
publicationOrg:MICCAI
year:2020
pdfUrl:https://arxiv.org/pdf/2007.13224.pdf
pdfPath:G:\search-rattailcollagen1\ScienceSearcher/data/cache/demo/2/PDFs/MICCAIPRIME2020UniformizingTechniquestoProcessCTscanswith3DCNNsforTBPrediction.pdf
publicationUrl:https://arxiv.org/pdf/2007.13224.pdf
codeUrl:https://github.com/hasibzunair/uniformizing-3D
datasetUrl:
videoUrl:https://www.youtube.com/embed/IP6poudyny4
videoPath:G:\search-rattailcollagen1\ScienceSearcher/data/cache/demo/2/videos/MICCAI-PRIME 2020 - Uniformizing Techniques to Process CT scans with 3D CNNs for TB Prediction.mp4
pdfText:To learn the geometric properties of volumetric data, there are challenges imposed by the data itsel...
_id:34
- 根据内容定位视频
title:X-Caps: Encoding Visual Attributes in Capsules for Explainable Medical Diagnoses | MICCAI 2020
authors:Rodney LaLonde
abstract:
publicationOrg:MICCAI
year:2020
pdfUrl:https://arxiv.org/pdf/1909.05926.pdf
pdfPath:G:\search-rattailcollagen1\ScienceSearcher/data/cache/demo/2/PDFs/XCapsEncodingVisualAttributesinCapsulesforExplainableMedicalDiagnosesMICCAI2020.pdf
publicationUrl:https://arxiv.org/pdf/1909.05926.pdf
codeUrl:https://github.com/lalonderodney/X-Caps
datasetUrl:
videoUrl:https://www.youtube.com/embed/kJ4SLLJO0GM
videoPath:G:\search-rattailcollagen1\ScienceSearcher/data/cache/demo/2/videos/X-Caps- Encoding Visual Attributes in Capsules for Explainable Medical Diagnoses - MICCAI 2020.mp4
pdfText:In machine learning, predictive performance typically comes at the cost of interpretability . While ...
_id:16
videoStruct:[{'timeStart': '00-03-38', 'timeEnd': '00-03-42', 'sentence': 'and if we want our models to be explainable at the human level'}]
- 根据id进行搜索
title:Let's Stop Incorrect Comparisons in End-to-end Relation Extraction! (EMNLP 2020)
authors:mlia deeplearning
abstract:
publicationOrg:EMNLP
year:2020
pdfUrl:https://arxiv.org/pdf/2009.10684.pdf
pdfPath:G:\search-rattailcollagen1\ScienceSearcher
publicationUrl:https://arxiv.org/pdf/2009.10684.pdf
codeUrl:https://github.com/btaille/sincere
datasetUrl:
videoUrl:https://www.youtube.com/embed/sIG8g85D03k
videoPath:G:\search-rattailcollagen1\ScienceSearcher/data/cache/demo/1/videos/Let's Stop Incorrect Comparisons in End-to-end Relation Extraction! (EMNLP 2020).mp4
pdfText:...
_id:0
- 根据关键词查询标题
ICML 2020: Learning De-biased Representations with Biased Representations
Understanding Regularisation Methods for Continual Learning @ICML CL Workshop
[Spotlight at NeurIPS 2020] Probabilistic Linear Solvers for Machine Learning
[CVPR 2020 Award Nominee] Deep Geometric Functional Maps: Robust Feature Learning for Shape Correspondence
- 视频多线程并行处理
- 在服务端部署音频-文字转换模型,减少网络通信的开销
- 当MongoDB服务出现问题时,可实现检索系统按批次回滚操作
- PDF与视频文件利用脚本进行自动化传输(使整个系统与爬虫模块联动,达到完全自动化运行)
- 考虑定义doi号和arxiv号为id,减少论文冗余量
- 在DatabaseAccess端接入爬虫数据合法性检测的功能
- 将本系统的客户端与服务端模块完全解耦,在展示模块仅保留系统中的客户端