Do implementacji algorytmu skorzystałem z biblioteki pyeasyga. Algorytm uruchamiałem w kilku różnych konfiguracjach wykorzystując zdefiniowane przez siebie różne funkcje fitness. Poniżej zestawienie tychże konfiguracji.
| Nazwa | Wariant (funkcja fitness) | Liczebność populacja | Prawdopodobieństwo mutacji | Elityzm |
|---|---|---|---|---|
| B-WholeLine | BasicWholeLineVariant | 200 | 0.05 | Tak |
| B-EditDist | BasicEditDistanceVariant | 200 | 0.05 | Tak |
| B-Diff | BasicDiffsVariant | 200 | 0.05 | Tak |
| Ex-WholeLine | ExtendedWholeLineVariant | 200 | 1 | Tak |
| Ex-Diff | ExtendedDiffVariant | 200 | 1 | Tak |
| Ex-WholeLine2 | ExtendedWholeLineVariant | 500 | 1 | Tak |
| Ex-Diff2 | ExtendedDiffVariant | 500 | 1 | Tak |
| Ex-Diff3 | ExtendedDiffVariant | 3000 | 1 | Tak |
Najbardziej interesujące są warianty. Zdefiniowane są one w pliku variants.py.
Warianty zaczynające się od Basic wykorzystują reprezentację chromosomu w postaci 0/1 długości liczby pól nonogramu. 1 oznacza pole zamalowane, 0 - pole puste, a cały chromosom to zapis możliwego rozwiązania wiersz po wierszu.
Reprezentacja ta jest bardzo prosta, gdyż wykorzystuje domyślne funkcje mutujące z biblioteki.
Bardziej interesującą reprezentacją jest ta wykorzystana w wariantach zaczynających się od Extended. Koduje ona możliwe rozwiązanie jako listę długości liczby wierszy. Elementy tejże listy kodują poszczególne wiersze. Najlepiej zobrazuje to przykład. Taki wiersz:
zostanie zakodoway jako:
[1, 2, 4, 0]
Pierwsza liczba oznacza, ile wolnych pól jest przed pierwszym zamalowanym polem. Ostatnia analogicznie ile jest niezamalowanych pól po ostatnim zamalowanym. Liczby w środku także oznaczają liczbę niezamalowanych pól pomiędzy kolejnymi zamalowanymi - są jednak pomniejszone o 1 (pomniejszenie o 1 wynika ze sposobu generowania nowych chromosomów, które opisuję poniżej).
Mając taką reprezentację oraz opis instancji nonogramu, jesteśmy w stanie jednoznacznie odtworzyć wygląd planszy. Implementacja takiego odtwarzania znajduje się w metodzie NonogramConverter.convert_to_solution() w pliku converter.py.
O ile w przypadku reprezentacji Basic jest ono trywialne, o tyle w przypadku Extended już niekoniecznie. Istota problemu to generowanie losowej reprezentacji z jednakowym prawdopodobieństwem dla każdej.
Podczas pracy nad projektem dwukrotnie zmieniałem funkcję generującą. Rozpocząłem od następującej:
def create_row(clue, width):
if not clue:
return []
c = np.array(clue)
reserved = np.sum(c) + len(c) - 1
left = width - reserved
row = []
row.append(random.randint(0, left))
left -= row[-1]
for _ in range(len(c)-1):
row.append(random.randint(0, left))
left -= row[-1]
return rowGenerowała ona rozmiary kolejnych przerw, jednak przez to dużo bardziej prawdopodobne były ustawienia, w których ostatnia zamalowana kratka była ostatnią w wierszu (było małe prawdopodobieństwo, że będzie odstęp od prawej strony).
Druga wersja losowała, którą przerwę "poszerzyć" aż suma długości przerw była odpowiednia:
def create_row(clue, width):
if not clue:
return []
c = np.array(clue)
reserved = np.sum(c) + len(c) - 1
left = width - reserved
row = [0 for _ in range(len(c)+1)]
for _ in range(left):
chosen = random.randrange(0, len(row))
row[chosen] += 1
return row[:-1]Tutaj jednak dużo bardziej prawdopodobne były rozwiązania, które miały równomierny rozkład pomięzy przerwy.
Ostatecznie zaimplementowałem następującą funkcję ExtendedVariant.create_row() w pliku variants.py:
@classmethod
def create_row(cls, clue, width):
if not clue:
return []
c = np.array(clue)
reserved = np.sum(c) + len(c) - 1
left = width - reserved
return cls.create_repr(left, len(c)+1)
@staticmethod
def create_repr(left, length):
row = [random.randint(0, left) for _ in range(length)]
row.sort()
result = []
for prev, cur in zip([0, *row], [*row, left]):
result.append(cur - prev)
return resultGeneruje ona losowo tyle liczb, ile ma być przerw. Każda liczba jest z zakresu od 0 do sumarycznej liczby pustych kratek w danym wierszu. Następnie wszystkie liczby są sortowane, a różnice pomiędzy nimi wyznaczają rozmiary przerw.
W tym podejściu nadal nie każda konfiguracja jest tak samo prawdopodobna, ale rozkład prawdopodieństwa jest bardziej wyrównany niż w poprzednich propozycjach. Potwierdziły to także uruchomienia na danych testowych.
Poszczególne warianty różnią się także zastosowaną funkcją fitness.
-
WholeLine- w tej funkcji fitness liczone są ograniczenia, które nie zostały spełnione. Za każde odejmowany jest 1 punkt karny. Rozwiązanie optymalne będzie miało wartość 0, zaś najgorsze -(suma wysokości i szerokości nonogramu). -
EditDistance- funkcja ta konwertuje potencjalne rozwiązanie na opisy wierszy i kolumn, a następnie dla każdego wiersza i kolumny liczy odległość edycyjną pomiędzy opisem wygenerowanym, a oczekiwanym. Funkcja zwraca -(suma odległości). Optymalne rozwiązanie ma wartość 0, wszyskie inne zaś mają wartości ujemne. Funkcja ta jest mniej restrykcyjna, niż powyższa - bada podobieństwo poszczegónych wierszy i kolumn i zwraca wartości z większego zakresu niż$[0;1]$ . -
Diff- funkcja ta konwertuje wiersze i kolumny jak powyżej, jednak tym razem liczy sumę wartości bezwzględnych różnicy odpowiadających sobie elementów pomiędzy wygenerowanym napisem, a oczekiwanym. Jeśli któreś z napisów jest dłuższy to "wystające" elementy są również dodawane. Ostateczny wynik to -(suma wszystkich tak zdefiniowanych wartości). Ta funkcja jest najmniej restrykcyjna i najbardzie rozróżnia rozwiązania. Oczywiście optymalne rozwiązanie otrzymuje wartość 0.
def fitness_one_line(expected, actual):
min_len = min(len(expected), len(actual))
value_diff = np.sum(np.abs(np.array(expected[:min_len]) - np.array(actual[:min_len])))
missing_sum = sum(expected[min_len:])+sum(actual[min_len:])
return -(value_diff + missing_sum)Warto zauważyć, że w wariantach Extended wszystkie powyższe funkcje powinny zwrócić 0 podczas rozpatrywania wierszy, a liczyć będą się tylko punkty karne dla kolumn (to jedna z przewag reprezentacji Extended).
W wariantach Extended zastosowałem specyficzne operatory mutacji i crossover.
Podczas mutacj z pewnym prawdopodobieństwem następuje zastąpienie całego wiersza.
@classmethod
def mutate(cls, individual):
row_index = random.randrange(len(individual))
old_repr = np.array(individual[row_index])
individual[row_index] = cls.create_repr(np.sum(old_repr), len(old_repr))Operator crossover jest zaś wielopunktowy - dla każdego wiersza losouje, z którego rodzica wybrać rozwiązanie.
def crossover(parent_1, parent_2):
child_1 = []
child_2 = []
for index in range(len(parent_1)):
if random.randint(0,1) > 0:
child_1, child_2 = child_2, child_1
child_1.append(parent_1[index])
child_2.append(parent_2[index])
return child_1, child_2Swoje rozwiązania zacząłem testować na nonogramie motyla (butterfly0.non). Korzystałem także z przykładów z repozytorium nonogram-db. Niestety zawiera ona niewiele nonogramów, zwłaszcza tych o małych wymiarach. Skorzystałem więc tylko z formatu używanego tamże i napisałem własny generator losowy nonogramów. Znajduje się on w pliku generator.py. Jego uruchomienie wygląda następująco:
python generator.py istniejący_folder_docelowy wysokość szerokość liczba_nonogramówPrzykładowo:
python generator.py db/25x25 25 25 10Za pomocą tego skryptu wygenerowałem po 10 nonogramów w rozmiarach:
- 5x5
- 5x10
- 10x5
- 10x10
- 15x15
- 20x20
- 25x25
Aby uruchomić algorytm genetyczny należy wykonać plik main.py. Jeśli podamy mu argument - ścieżkę do pliku .non to program rozpocznie rozwiązywanie nonogramu z podanego pliku.
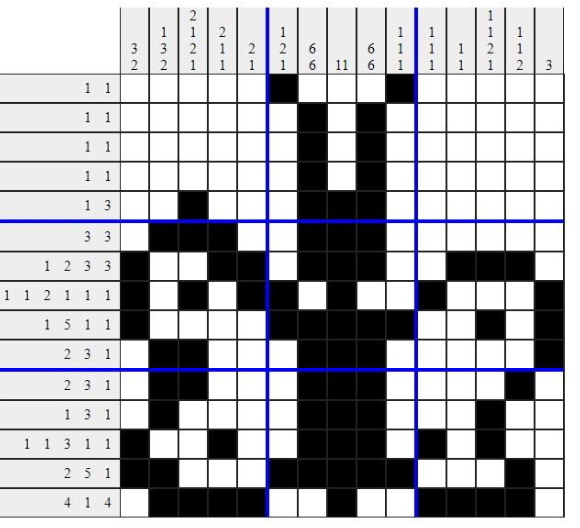
python main.py db/butterfly/butterfly0.nonNa samym początku program wyświetli oczekiwany rezultat (wczytany z pliku .non). Następnie będzie działał wyświetlając postęp z użyciem biblioteki tqdm, aby na koniec wyświetlić znalezione rozwiązanie oraz wykres działania.
Kod tworzący wizualizacje obrazków znajduje się w pliku visualize.py. Na wygenerowanym obrazku zastosowałem 2 stopniową skalę czerwieni obrazującą, czy dany wiersz bądź kolumna są poprawne czy nie.
Jeśli podczas uruchomienia ścieżka nie zostanie podana, program uruchomi się w trybie porównania konfiguracji. Wygeneruje on wtedy raporty, które znajdują się już w katalogu results.
W trybie porównania algorytm genetyczny uruchamiany jest w konfiguracjach określonych przez zestawy na nonogramach z katalogu db.
| Kategoria | Liczba generacji | Zestaw konfiguracji |
|---|---|---|
| 5x5 | 30 | 1 |
| 5x10 | 50 | 2 |
| 10x5 | 50 | 2 |
| 10x10 | 50 | 3 |
| 15x15 | 50 | 4 |
| 20x20 | 50 | 5 |
| 25x25 | 50 | 5 |
| butterfly | 100 | 6 |
- B-WholeLine
- B-EditDist
- B-Diff
- Ex-WholeLine
- Ex-Diff
- Ex-WholeLine2
- Ex-Diff2
- Ex-Diff3
- B-WholeLine
- B-EditDist
- B-Diff
- Ex-WholeLine
- Ex-Diff
- Ex-WholeLine2
- Ex-Diff2
- Ex-Diff3
- B-WholeLine
- B-EditDist
- B-Diff
- Ex-WholeLine
- Ex-Diff
- Ex-WholeLine2
- Ex-Diff2
- Ex-Diff3
- B-WholeLine
- B-EditDist
- B-Diff
- Ex-WholeLine
- Ex-Diff
- Ex-WholeLine2
- Ex-Diff2
- Ex-Diff3
- B-WholeLine
- B-EditDist
- B-Diff
- Ex-WholeLine
- Ex-Diff
- Ex-WholeLine2
- Ex-Diff2
- Ex-Diff3
- B-WholeLine
- B-EditDist
- B-Diff
- Ex-WholeLine
- Ex-Diff
- Ex-WholeLine2
- Ex-Diff2
- Ex-Diff3
Aby zredukować losowość wyników, w każdej kategorii uruchomiłem różne konfiguracje na różnych nonogramach i wyciągnąłem średnią z ostatecznej wartości funkcji fitness. Dzięki temu można bezpieczniej porównywać różne konfiguracje, pamiętając jednak o wykorzystywanych przez nie różnych funkcjach fitness.
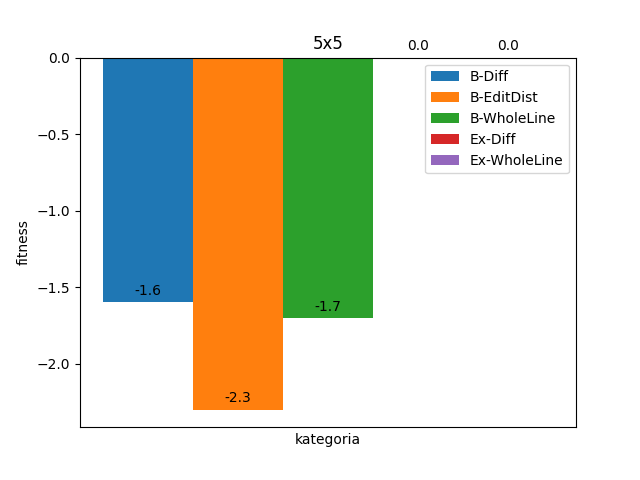
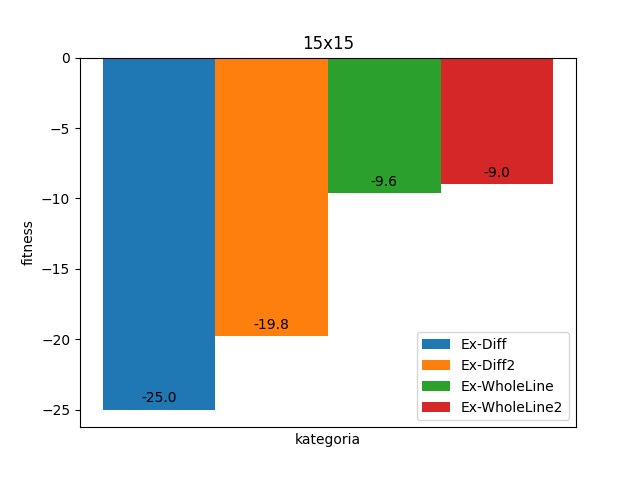
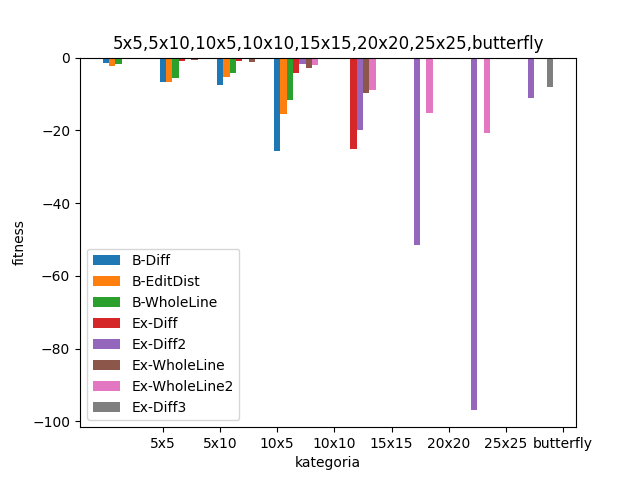
Warianty Basic już dla najmniejszych obrazków 5x5 nie zawsze znajdują optymalne rozwiązanie.
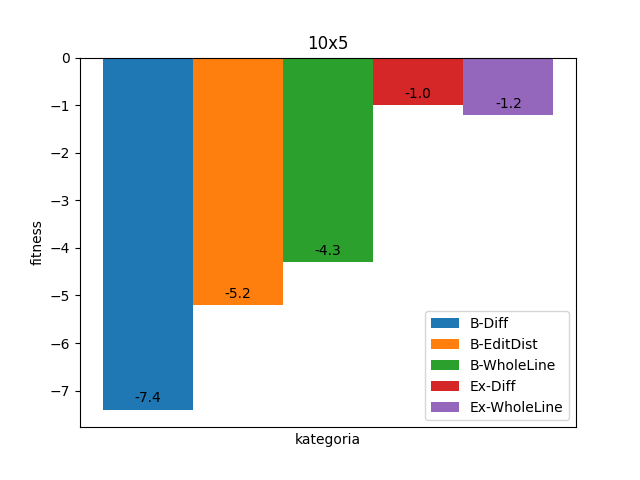
Słabość wariantów Basic potwierdza się także na większych obrazkach. Warianty Extended wypadają lekko gorzej, ale nadal staysfakcjonująco. Spodziewałem się jednak zauważalnej różnicy w działaniu na obrazkach 5x10 vs 10x5 wynikającej z reprezentacji chromosomu. Niczego takiego jednak nie widzę.
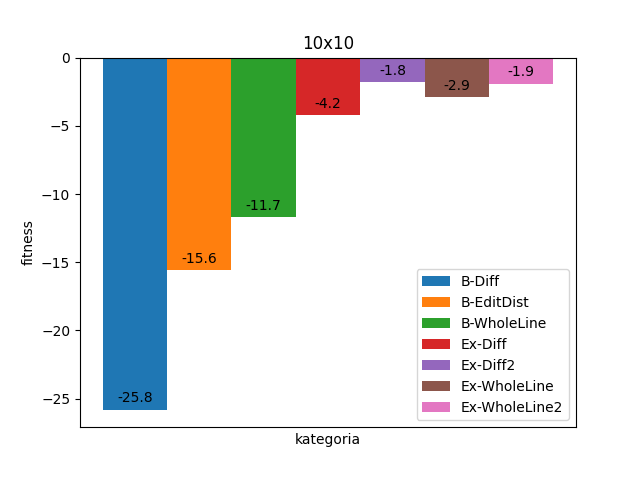
Dla obrazków 10x10 warianty Basic mocno odstają - przykładowo B-WholeLine nie jest w stanie spełnić średnio 12 wymagań (czyli np. żadna z kolumn oraz 2 wiersze nie są dopasowane). Możemy jednak zauważyć oczywisty fakt pomiędzy wariantami Extended 1 i 2 - te ze zwiększoną liczebnością populacji dają lepsze rezultaty.
Tutaj warto zaznaczyć, że funkcje fitness są liczone w różny sposób, więc warianty Diff niekoniecznie są gorsze (raczej jest wręcz odwrotnie).
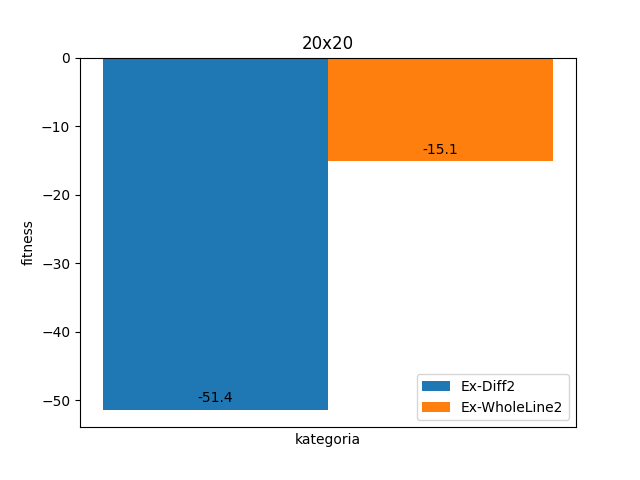
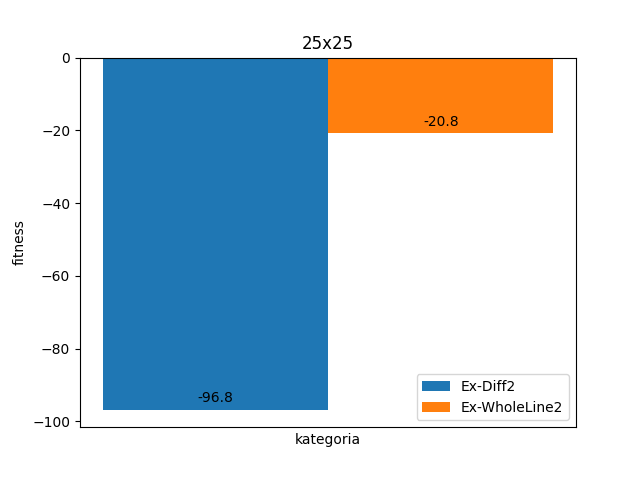
Dla większych obrazków nawet lepsze warianty nie dają już satysfakcjonujący wyników.
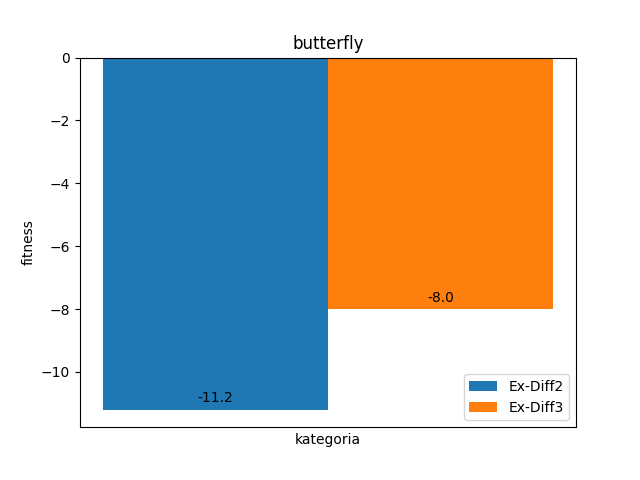
Dla przykładu z motylem nawet zwiększenie populacji do 3000 nie pozwala znaleźć optymalnego rozwiązania.
Tak prezentują się zaś powyższe diagramy razem.
Algorytmy genetyczne mają duży potencjał i pozwalają na rozwiązywanie problemów, które są zbyt trudne dla klasycznych algorytmów. Dla nonogramów łatwo wyobrazić sobie brutalne rozwiązanie, które działa w czasie $O^(2^{n \cdot m})$. Z pomocą techniki meet-in-the-middle moglibyś zapewne rozwiązać problem w czasie $O^(2^{\frac{n \cdot m}{2}})$ . Jednak już dla obrazków 10x10 to rozwiązanie będzie zbyt wolne.
Z drugiej strony niewiele większe obrazki, tj. już 20x20 nie są w satysfakcjonujący sposób rozwiązywane przez algorytmy genetyczne. Nawet mniejsze nonogramy nie mają 100% rozwiązywalności przez algorytmy genetyczne. Dlatego nie jest to rozwiązanie idealne.
Osobiście byłem lekko zawiedziony, że moje implementacje nie były w stanie rozwiązać nonogramu z motylem. Najlepsze znalezione przez nie rozwiązanie miało funkcję fitness -2. Najlepsze rozwiązanie z wizualizacją, które udało mi się uchwycić miało funkcję fitness -4.