交流群在:https://mp.weixin.qq.com/s/N9YFEkh0TjZ4BzZvP5OT7g
生信技能树联盟创始人jimmy手把手带你完成一个GEO数据库(GSE76275)挖掘实例,从必备R包安装,表达矩阵下载,PCA/boxplot/heatmp的数据检查,探针ID到基因ID的转换,根据生物学分组进行差异分析并且绘制火山图、热图,还有简单的超几何分布检验的KEGG等数据库注释结果。
表达矩阵进行GSEA/GSVA分析。
根据TNBC(三阴性乳腺癌)的生物学特征提取指定基因的表达量,使用genefu这个R包进行PAM50分类。
下载TCGA数据库的BRCA的芯片表达矩阵,同样进行PAM50分类,结合临床信息,并且对应比较GSE76275数据集。
下载TCGA数据库的BRCA的RNA-seq表达矩阵,同样进行PAM50分类,结合临床信息,并且对应比较GSE76275数据集。
下载GTEx数据库的RNA-seq表达矩阵,并且提取其中属于breast的样本,同样进行PAM50分类,结合临床信息,并且对应比较GSE76275数据集。
下载METEBRIC数据库的RNA-seq表达矩阵,同样进行PAM50分类,结合临床信息,并且对应比较GSE76275数据集。
需要自行下载学习R语言及熟练使用Rstudio编辑器,根据课程配套代码安装R包。
首先需要了解GEO数据库,建议通读: 解读GEO数据存放规律及下载,一文就够
然后根据课件代码使用GEOquery包进行下载,详细教程见:从GEO数据库下载得到表达矩阵 一文就够
示例数据集:GEO数据库(GSE76275) 对应的3篇SCI文章见课程附件。
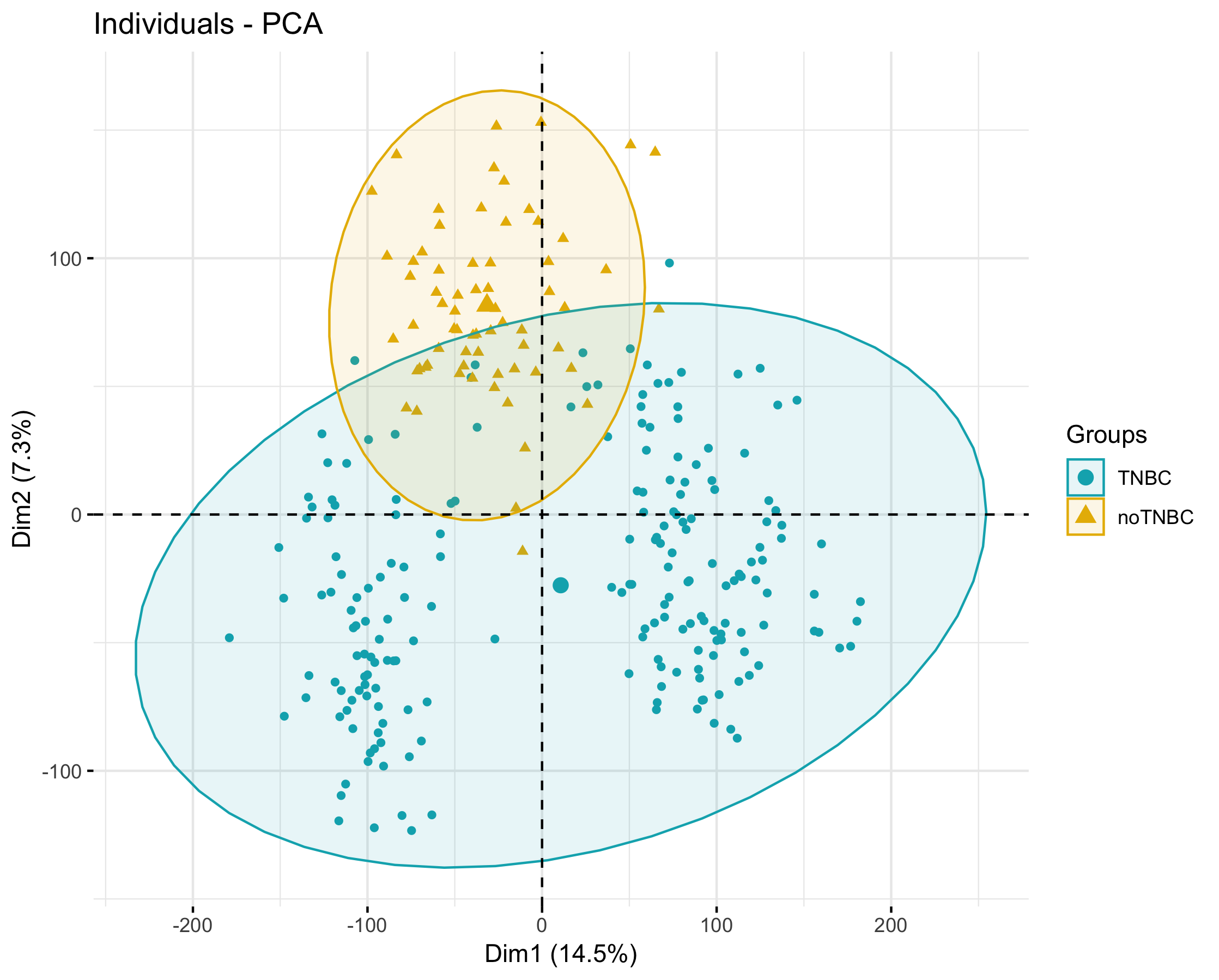
首先根据全局基因表达绘制PCA图如下:
代码见课程附件,具体参数视频里有介绍,结果很容易理解,可以看到那些不属于TNBC的样本跟TNBC组有着很清晰的界限,而且可以看到TNBC组本身也有着界限分明的两列。
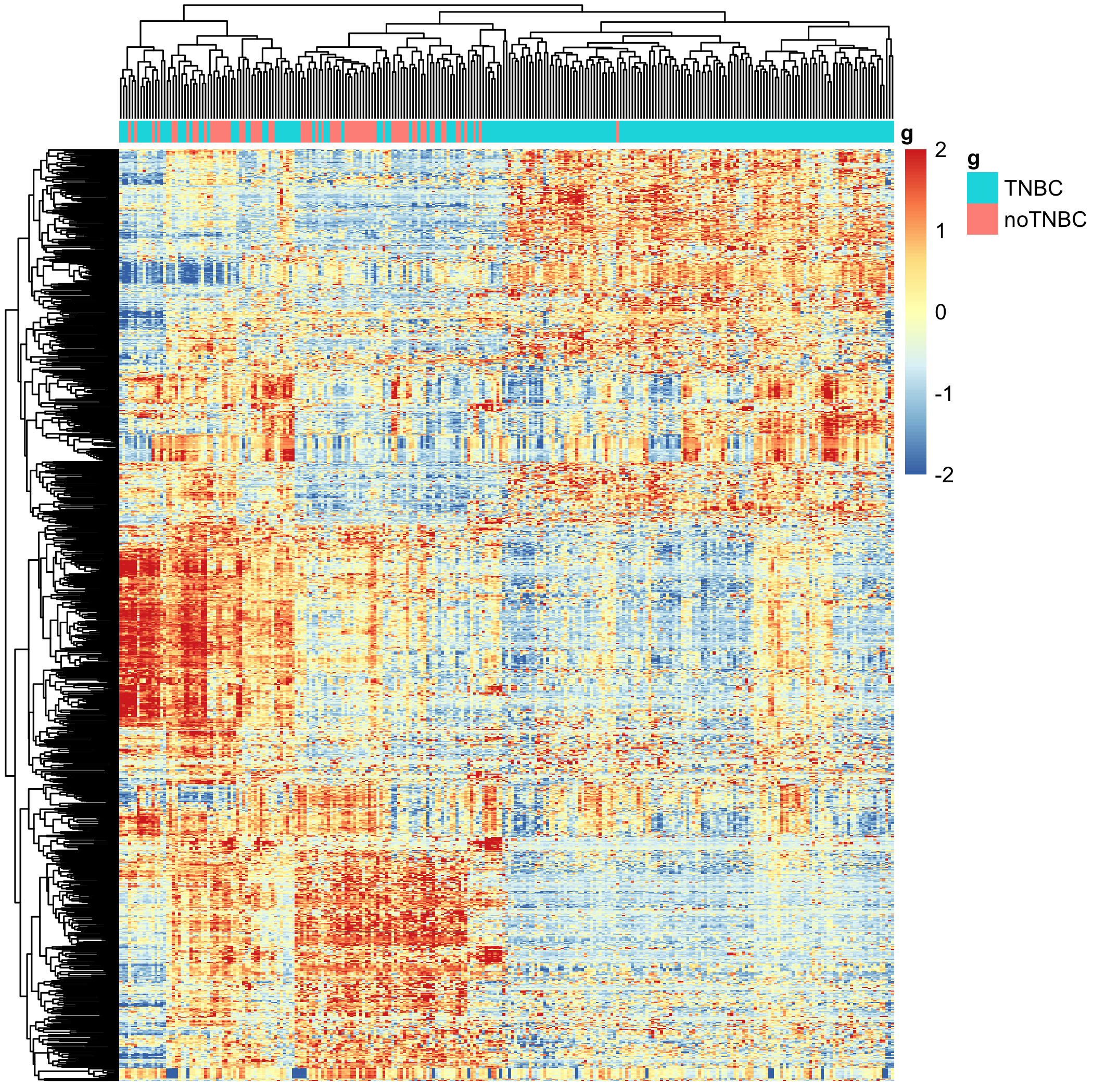
还有top 1000的sd的基因提取出来绘制热图:
这个结果跟PCA分析结果相呼应!
这里走标准的limma流程,详细推文介绍:根据分组信息做差异分析- 这个一文不够的
得到的差异分析结果,也可以走标准的火山图,热图,GO/KEGG数据库注释,见推文:差异分析得到的结果注释一文就够
代码在:https://github.com/jmzeng1314/GSE76275-TNBC
代码在:https://github.com/jmzeng1314/TCGA_BRCA
代码在:https://github.com/jmzeng1314/gtex_BRCA
代码在:https://github.com/jmzeng1314/METABRIC
对表达矩阵挑选top5000的MAD基因,以及top10000后,分别独立走WGCNA流程看结果。