Целью работы над проектом является поиск и изучение участков генома, где гистоновая метка H3K36me3 присутствует в местах образования вторичной структры ДНК G4.
Гистограмма длин участков до конвертации (версия генома human(hg38), ChIP-seq эксперимент ENCFF655QDU)
Количество пиков - 147306
Гистограмма длин участков после конвертации (версия генома human(hg19), ChIP-seq эксперимент ENCFF655QDU)
Количество пиков - 147202
Гистограмма длин участков до конвертации (версия генома human(hg38), ChIP-seq эксперимент ENCFF257ZFX)
Количество пиков - 70267
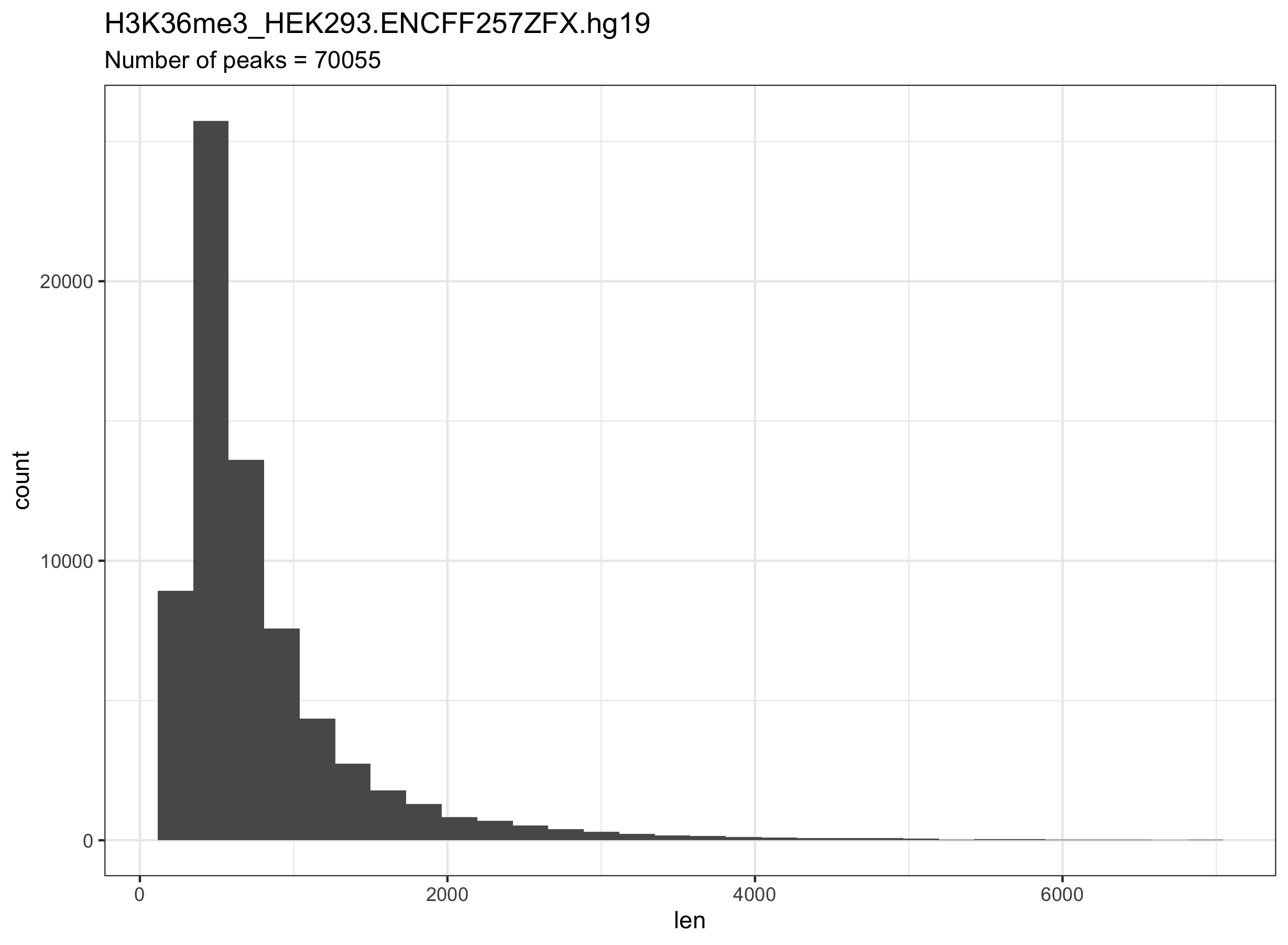
Гистограмма длин участков после конвертации (версия генома human(hg19), ChIP-seq эксперимент ENCFF257ZFX)
Количество пиков - 70246
Установим порог на длину 7000
Количество пиков - 145784
Количество пиков - 70055
Объединяем два набора отфильтрованных ChIP-seq пиков с помощью утилиты bedtools merge. Перед этим сортируем единый .bed файл. Для этого используем команду "cat *.filtered.bed | sort -k1,1 -k2,2n | bedtools merge > H3K36me3_HEK293.merge.hg19.bed"
Скачиваем файл со вторичной стр-рой ДНК G4_seq_Li_K (т.к файлов несколько, необходимо объединить их командой "cat *.max.K.w50.25.bed | sort -k1,1 -k2,2n | bedtools merge > G4_seq_Li_K.bed")
Количество пиков - 428624
Их количество - 28624
Ссылка на сессию в геномном браузере: http://genome.ucsc.edu/s/nicka1106/hse21_H3K36me3_G4_human
Скриншоты мест, где есть пересечение между гистоновой меткой и структурой ДНК:
Соответствующие геномные координаты: chr1:3,561,656-3,563,766
Соответствующие геномные координаты: chr1:1,452,583-1,458,915
Кол-во пиков, которые удалось проассоциировать с генами - 759 Общее кол-во уникальных генов - 552
Наиболее значимые категории, найденные в ходе GO-анализа:
- mitochondrial translation
- detection of chemical stimulus
- mitochondrial translational termination
- protein-containing complex disassembly
- mitochondrial gene expression
Полный список категорий находится в файле pantherdb_GO_analysis.txt