https://github.com/QuentinAM/VITK
- Python3
pip3 install -r requirements.txtpython3 src/main.pyVous pouvez modifier les variables globales dans le fichier src/main.py pour changer le debug et la visualisation des images.
Pour ce projet , nous avons fait du pair programming a l'aide de l'extension live share de vscode pour pouvoir travailler ensemble en distanciel sur un ordinateur.
- Jean-Yves CHEN (login: jean-yves.chen)
- Quentin ABEL MARCEAU (login: quentin.abel-marceau)
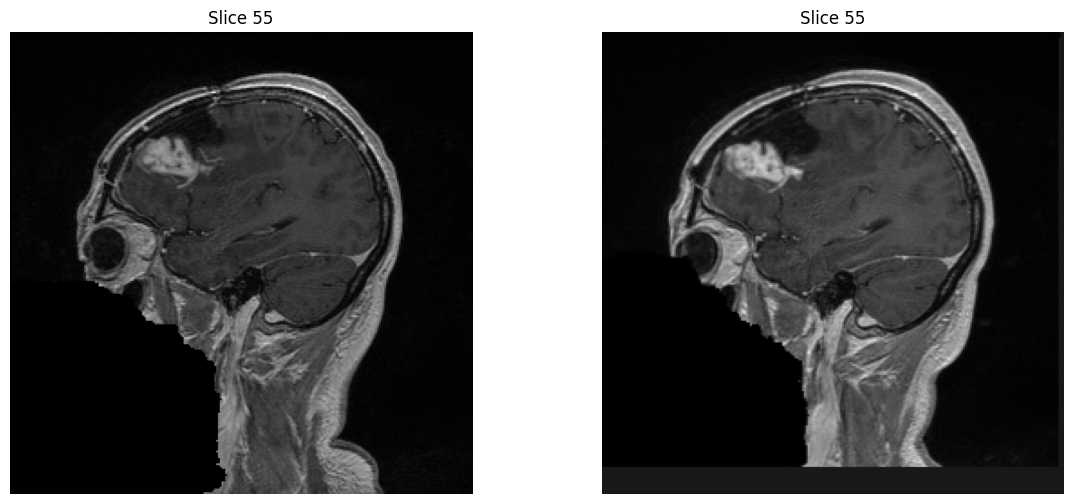
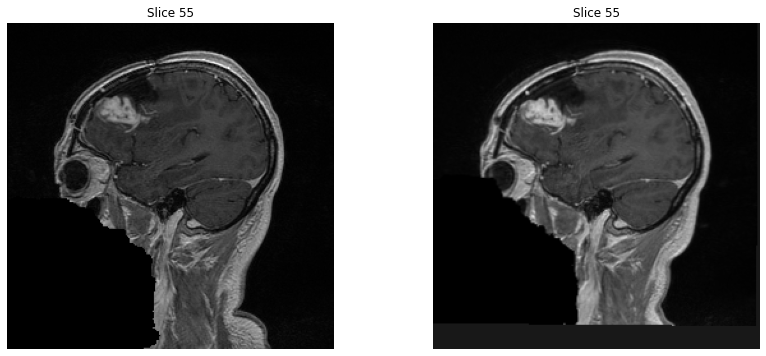
Nous avons testé deux méthodes de recalage : rigide et affine.
Les deux méthodes donne un résultat similaire mais en terme de temps la méthode rigide est environ 2 fois plus rapide. Pour vérifier quelle méthode est la plus optimale nous avons les résultats suivant :
Différence sur l'image rigide
| Statistic | Value |
|---|---|
| Mean | 51.14967721138341 |
| Variance | 10654.82721334736 |
| Sigma | 103.22222247824041 |
| Sum of Squares | 153073508367.94022 |
| Standard Deviation | 103.22222247824041 |
| Minimum | 0.0 |
| Maximum | 1368.4420166015625 |
Différence sur l'image affine
| Statistic | Value |
|---|---|
| Mean | 46.917091927121554 |
| Variance | 9074.344851899896 |
| Sigma | 95.25935571847994 |
| Sum of Squares | 130056069715.91301 |
| Standard Deviation | 95.25935571847994 |
| Minimum | 0.0 |
| Maximum | 1358.0 |
En analysant les résultats, on voit directement que la méthode affine réduit davantage la différence entre l'image fixe et l'image enregistrée que la méthode rigide. En effet, la différence totale entre les deux images est plus faible avec la méthode affine, ce qui suggère qu'elle est supérieure à la méthode rigide pour mesurer la différence entre les images.
Nous allons donc partir sur la méthode affine pour le recalage d'image.
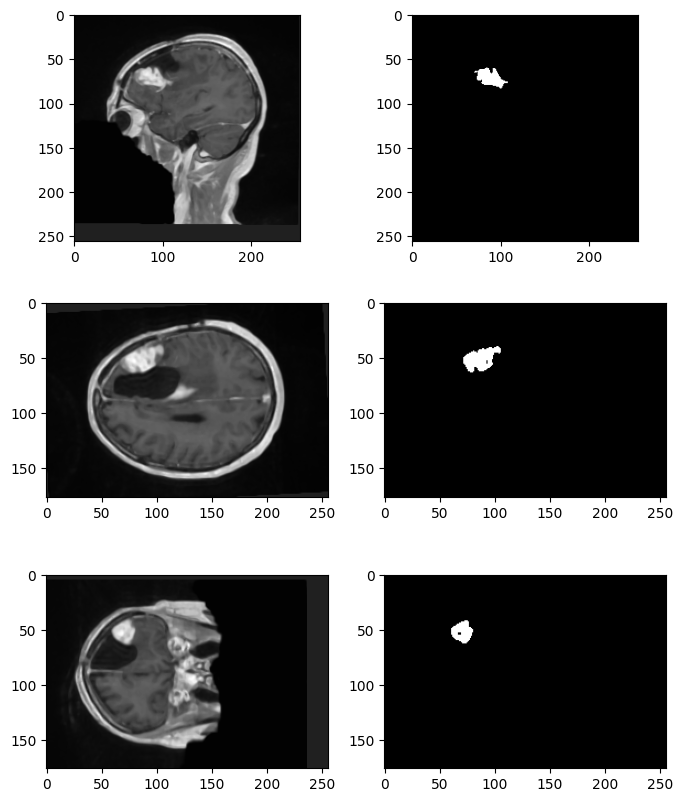
L’algorithme de segmentation que nous avons utilisé est un filtre d’image par seuillage connecté. Il s’agit d’une méthode de segmentation semi-automatique qui permet d’isoler une région d’intérêt dans une image en appliquant des seuils précis.
Pour segmenter la tumeur, nous avons défini un seuil supérieur et un seuil inférieur pour délimiter l’intensité des voxels de la tumeur. Nous avons également précisé la position d’un voxel initial appartenant à la tumeur, en utilisant les coordonnées de la tumeur observée dans l’image.
Les seuils et la position de la graine pour nos deux images sont les suivants :
lower_threshold = 500
upper_threshold = 800
seed_position = (90, 70, 51)
L’algorithme procède alors à la propagation de la segmentation à partir de la graine dans l’image, en examinant les voxels adjacents. Si la valeur d’un voxel voisin se situe entre les seuils inférieur et supérieur, ce voxel est considéré comme faisant partie de la région d’intérêt et est étiqueté avec une valeur spécifique
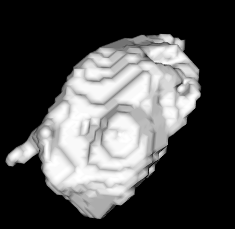
Pour la visualisations des changements, nous sommes parti sur 3 axes différents :
- Similarité de Dice
- Différence volumique
- Différence d'intensité
Le coefficient de similarité de Dice, qui est de 0.0032 dans notre cas, indique un très faible chevauchement entre les deux régions où la tumeur est. Cela signifie que l'intersection des régions tumorales est extrêmement réduite par rapport à leur union.
La différence de volume entre les deux régions tumorales est de 72930.0 unités cubes, ce qui montre une disparité significative de taille entre elles. La deuxième tumeur, dont le volume est de 2119560.0 unités cubes, est plus grande que la première, qui a un volume de 2046630.0 unités cubes.
La différence d'intensité des valeurs de voxels entre les deux régions tumorales est de 0.06296. Cela reflète une dissimilarité dans les intensités des pixels entre les deux tumeurs. Une différence d'intensité plus élevée suggère que les deux tumeurs ont des caractéristiques d'intensité de pixels distinctes.
Dans ce projet, nous avons pu expérimenté avec ITK et brièvement VTK. ITK est très puissant pour la segmentation, il produit des résultats plus que satisfaisants assez rapidement. La seule difficulté réside dans l'adaptation de la seed pour chaque modèle. Peut être que ce problème peut être réglé par apprentissage de machine learning ? Nous sommes tout de même satisfaits d’avoir appris à utiliser ces outils dans le cadre du projet et espérons pouvoir les utiliser à nouveau à l’avenir.