Финальный проект по курсу биоинформатики
Скачиваем данные
mkdir data ; cd data
wget https://www.encodeproject.org/files/ENCFF291DHI/@@download/ENCFF291DHI.bed.gz
gunzip ENCFF291DHI.bed.gz
cut -f1-5 ENCFF291DHI.bed > H3K27me3_ZDNA.ENCFF291DHI.hg38.bed
wget https://www.encodeproject.org/files/ENCFF695ETB/@@download/ENCFF695ETB.bed.gz
gunzip ENCFF695ETB.bed.gz
cut -f1-5 ENCFF695ETB.bed > H3K27me3_ZDNA.ENCFF695ETB.hg38.bedТак как версия генома - hg38, то приводим к версии hg19. LiftOver скачан с http://hgdownload.cse.ucsc.edu/admin/exe/macOSX.x86_64/liftOver.
cd ..
wget https://hgdownload.cse.ucsc.edu/goldenpath/hg38/liftOver/hg38ToHg19.over.chain.gz; gunzip hg38ToHg19.over.chain.gz
cd data
liftOver H3K27me3_ZDNA.ENCFF291DHI.hg38.bed ../hg38ToHg19.over.chain H3K27me3_ZDNA.ENCFF291DHI.hg19.bed H3K27me3_ZDNA.ENCFF291DHI.unmapped.bed
liftOver H3K27me3_ZDNA.ENCFF695ETB.hg38.bed ../hg38ToHg19.over.chain H3K27me3_ZDNA.ENCFF695ETB.hg19.bed H3K27me3_ZDNA.ENCFF695ETB.unmapped.bedСтроим гистрограммы: скрипт находится в текущем репозитории, дополнительно создаем директорию для сгенерированных гистрограмм
cd .. ; mkdir histsКоличество пиков в H3K27me3_ZDNA.ENCFF291DHI
Количество пиков в H3K27me3_ZDNA.ENCFF695ETB
В качестве порога для фильтрации пиков выбрано значение 500.
После фильтрации
До фильтрации
После фильтрации
До фильтрации
Число пиков после фильтрации:
- H3K27me3_ZDNA.ENCFF291DHI 19930
- H3K27me3_ZDNA.ENCFF695ETB 24747
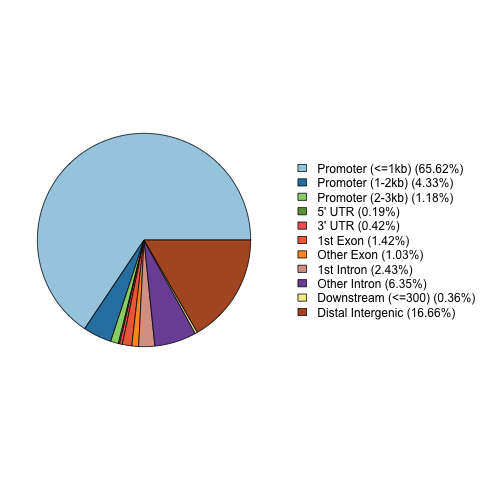
Расположение пиков относительно аннотированных генов
Для эксперимента ENCFF291DHI
Для эксперимента ENCFF695ETB
Объединим метки из двух отфильтрованных наборов
cd data
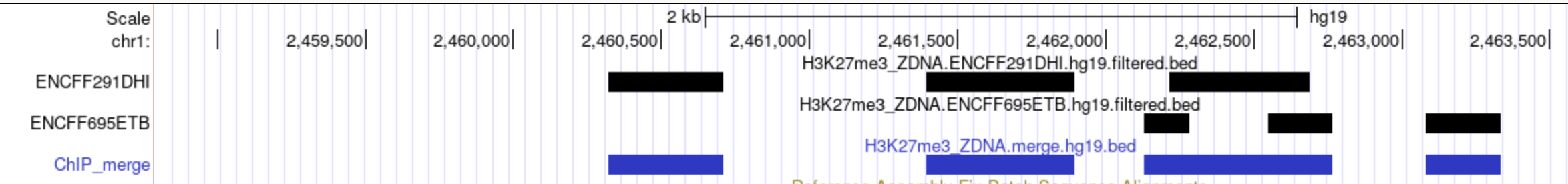
cat *.filtered.bed | sort -k1,1 -k2,2n | bedtools merge > H3K27me3_ZDNA.merge.hg19.bedВизуализируем данные в UCSC браузере
Треки:
track visibility=dense name="ENCFF291DHI" description="H3K27me3_ZDNA.ENCFF291DHI.hg19.filtered.bed"
https://raw.githubusercontent.com/Romanchenko/hse21_H3K27me3_ZDNA_human/main/data/H3K27me3_ZDNA.ENCFF291DHI.hg19.filtered.bed
track visibility=dense name="ENCFF695ETB" description="H3K27me3_ZDNA.ENCFF695ETB.hg19.filtered.bed"
https://raw.githubusercontent.com/Romanchenko/hse21_H3K27me3_ZDNA_human/main/data/H3K27me3_ZDNA.ENCFF695ETB.hg19.filtered.bed
track visibility=dense name="ChIP_merge" color=50,50,200 description="H3K27me3_ZDNA.merge.hg19.bed"
https://raw.githubusercontent.com/Romanchenko/hse21_H3K27me3_ZDNA_human/main/data/H3K27me3_ZDNA.merge.hg19.bedВзглянув на картинку, убеждаемся в корректности работы утилиты merge
Теперь повторим все этапы, кроме фильтрации, для вторичной структуры ДНК.
Скачиваем файл
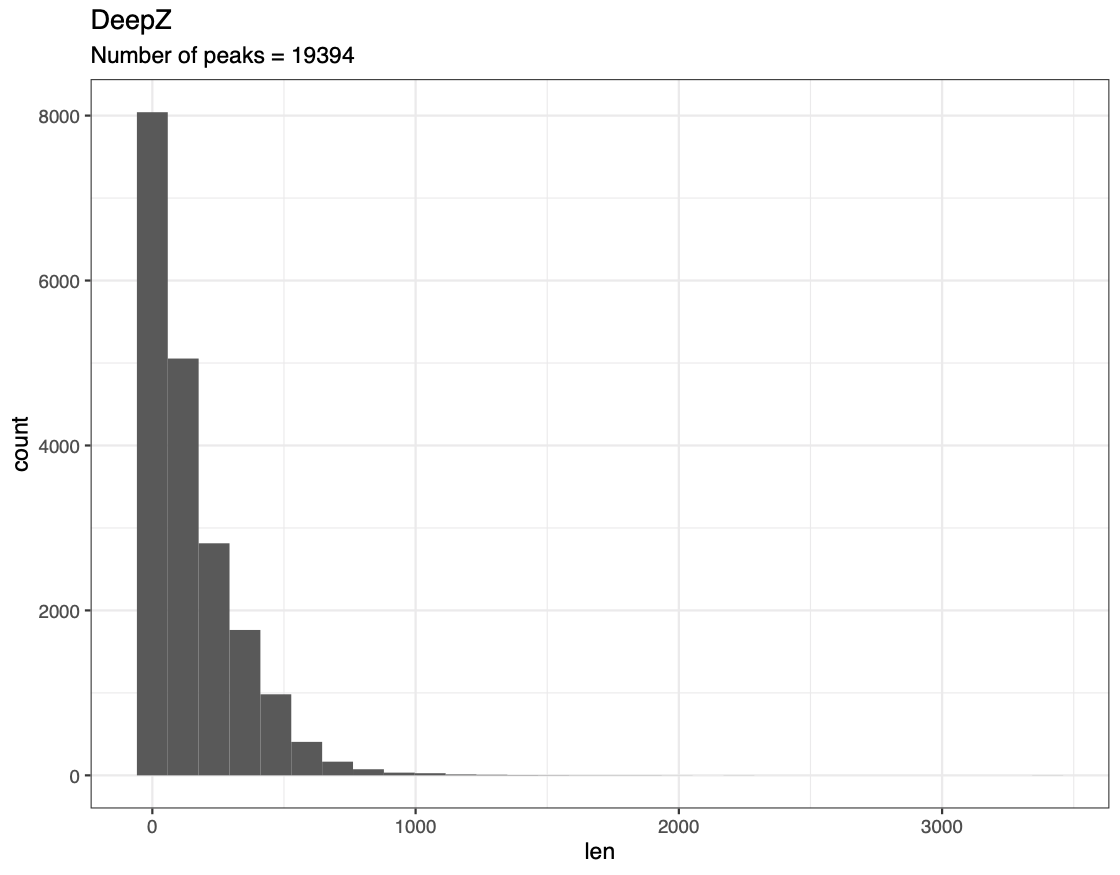
cd data ; wget https://raw.githubusercontent.com/Nazar1997/DeepZ/master/annotation/DeepZ.bedЧисло пиков: 19394
Распределение длин пиков:
Скрипт для определения расположения пиков структуры относительно аннотированного генома взят из пункта для пиков из экспериментов (просто изменены пути).
Пересекаем bed-файлы со структурой ДНК и с метками из экспериментов
intersect -a DeepZ.bed -b H3K27me3_ZDNA.merge.hg19.bed > H3K27me3_ZDNA.intersect_with_DeepZ.bedЧисло пиков: 837
Распределение длин пиков
Треки для геномного браузера:
track visibility=dense name="DeepZ" color=0,200,0 description="DeepZ"
https://raw.githubusercontent.com/Romanchenko/hse21_H3K27me3_ZDNA_human/main/data/DeepZ.bed
track visibility=dense name="intersect_with_DeepZ" color=200,0,0 description="H3K27me3_ZDNA.intersect_with_DeepZ.bed"
https://raw.githubusercontent.com/Romanchenko/hse21_H3K27me3_ZDNA_human/main/data/H3K27me3_ZDNA.intersect_with_DeepZ.bed
Сессия в браузере - http://genome.ucsc.edu/s/Romanchenko/hw_bioinf
Хорошие позиции в браузере
-
chr1:27,286,914-27,286,985 -- тут есть пересечение с аннотированным геном RefSeq Gene KDF1 https://www.genecards.org/cgi-bin/carddisp.pl?gene=KDF1
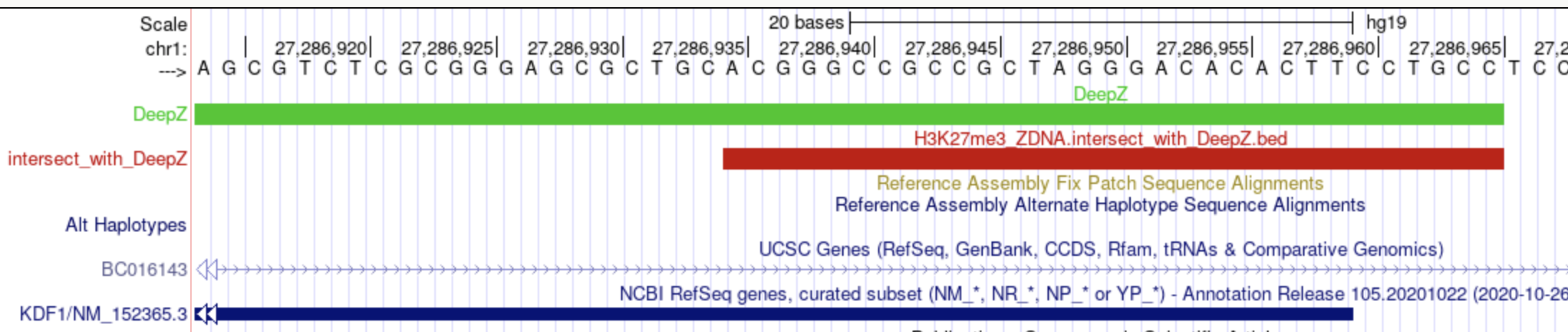
-
chr1:166,916,626-166,916,805 -- пересечение с интроном Human Gene ILDR2 https://www.genecards.org/cgi-bin/carddisp.pl?gene=ILDR2
Ассоциирование с имеющимися аннотированными генами дало результаты:
- всего ассоциаций пиков с генами 332
- уникальных генов 248
Результаты GO-анализа
Некоторые наиболее значимые категории
- cell proliferation involved in metanephros developmen FDR = 1.66E-02
- regulation of resting membrane potential FDR = 2.63E-02
- adenylate cyclase-activating dopamine receptor signaling pathway FDR = 3.16E-02