Prevendo o comportamento da segunda onda de COVID-19 no Brasil utilizando Evolução Diferencial e combinação de pacotes de onda Gaussianos.
*Analysis of time series datasets of recorded cases and deaths from COVID-19 and cases and deaths from SARS (severe acute respiratory syndrome) using Differential Evolution and the combination of Gaussian distribution waves to forecast the behavior of the second wave of COVID-19 in Brazil. *
Atenção: I'm currently working on more accurate models, so, I wont update this repository any more. New repository with a more accurate model: https://github.com/adsmendesdaniel/Forecasting-the-COVID-19-behavior-with-Differential-Evolution-and-a-Maxwell-Boltzmann-like-model
Análise de datasets contendo séries temporais de casos e óbitos registrados de Covid-19 e casos e óbitos por SRAG (síndrome respiratória aguda grave) utilizando Evolução Diferencial e combinação de ondas de distribuição gaussiana na previsão do comportamento da segunda onda de COVID-19 no Brasil.
Atenção: Estou trabalhando em modelos mais precisos, e não irei mais atualizar este repositório. Novo repositório com um modelo mais preciso: https://github.com/adsmendesdaniel/Forecasting-the-COVID-19-behavior-with-Differential-Evolution-and-a-Maxwell-Boltzmann-like-model
Evolução Diferencial é um algorítmo evolutivo simples da categoria de algorítmos meta-heurísticos, que através da criação de populações de soluções, suas seleções e recombinações, é capaz de obter um solução ótima ou aproximadamente ótima para um problema.
Desde que tenhamos dados o suficiente, é possível, facilmente, utilizar Evolução Diferencial para ajustar curvas/séries temporais provenientes de fenômenos físicos, por exemplo. Assim, podemos ajustar curvas geradas por fenômenos antropológicos como os relacionados ao COVID-19, desde que escolhamos um modelo matemáticos conveniente, que possa ser ajustado aos dados.
Pelo Teorema do Central do Limite, podemos encontrar que utilizar uma função de distribuição Gaussiana pode aproximar a solução de nosso problema já que variáveis aleatórias independentes podem estar gerando nosso conjunto de dados (na verdade, estas variáveis no caso da análise de dados de COVID-19 são tão independentes assim, logo, o ideal seria utlizar alguma função de distruibuição correspondente à uma Gaussiana Modificada). Apesar de não ser a melhor opção neste caso, podemos utilizar a sobreposição de dois pacotes gaussianos como função objetiva na solução de nosso problema de otimização com Evolução Diferencial. Assim, simplificadamente, podemos escrever:
f(α1, β1, γ1, α2, β2, γ2) = α1exp[-(x-β1)^2/2γ1^2] + α2exp[-(x-β2)^2/2γ2^2]
E encontrar os parâmeros que melhor ajustam a curva do modelo aos devidos dados utilizando Evolução Diferencial como algoritmo de otimização.
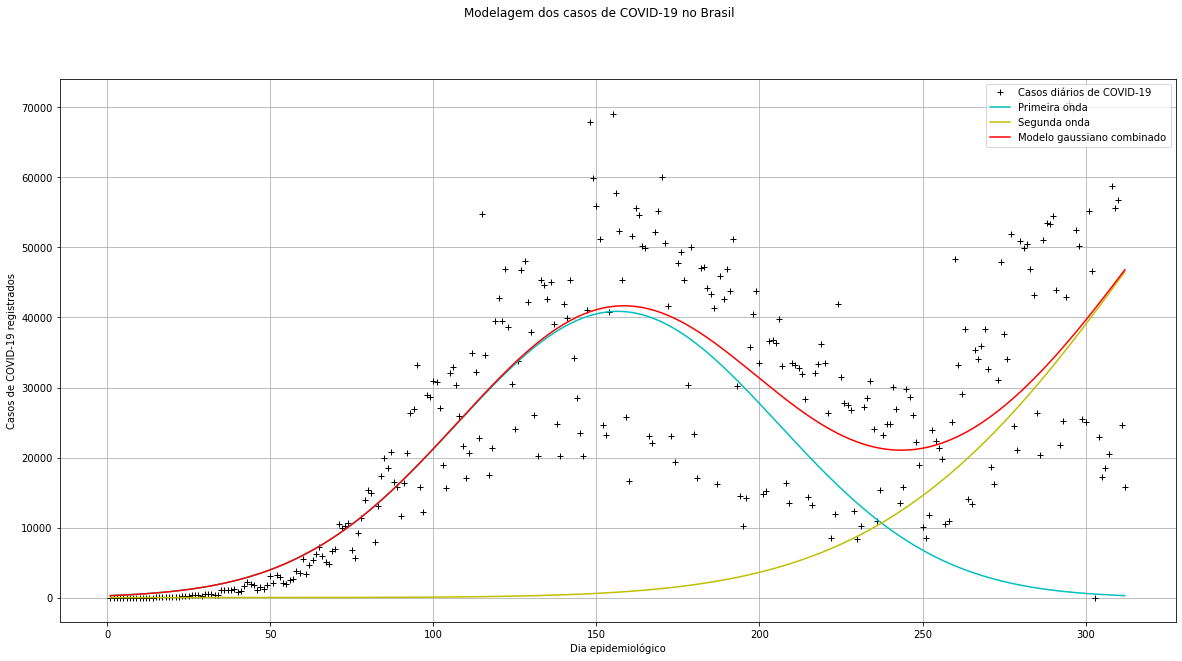
Ajuste feito em 03/01/2021 (Média dos parâmetros de 20 runs do algoritmo de ED):
Ajuste atualizado em 03/03/2021 (Média dos parâmetros de 10 runs do algoritmo de ED):
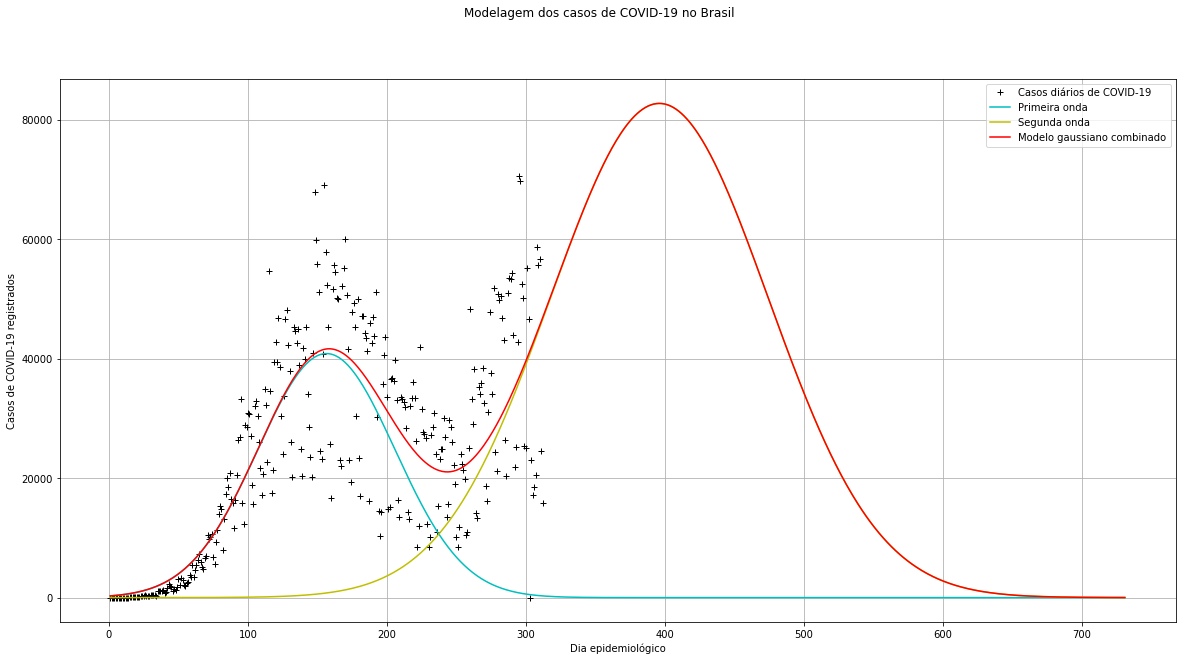
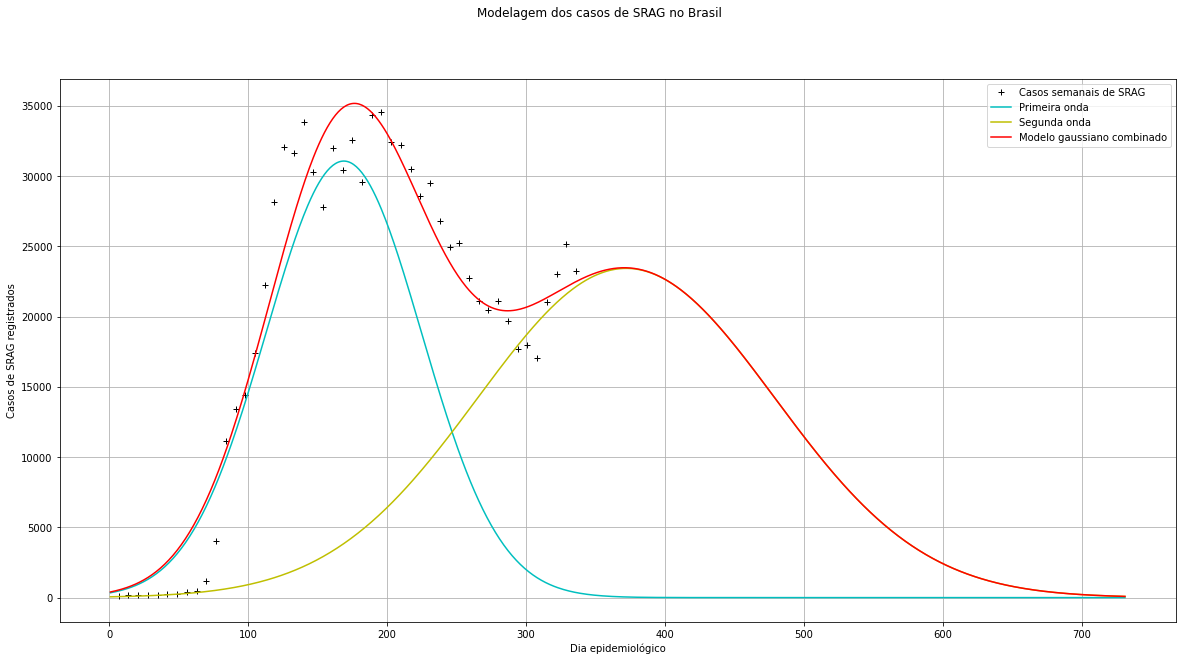
Previsão dos próximos dias epidemiológicos feita em 03/01/2021 (Média dos parâmetros de 20 runs do algoritmo de ED):
Previsão dos próximos dias epidemiológicos atualizada em 03/03/2021 (Média dos parâmetros de 10 runs do algoritmo de ED):
Ajuste feito em 03/01/2021 (Média dos parâmetros de 10 runs do algoritmo de ED):
Ajuste atualizado em 03/01/2021 (Média dos parâmetros de 20 runs do algoritmo de ED):
Previsão dos próximos dias epidemiológicos feita em 03/01/2021 (Média dos parâmetros de 10 runs do algoritmo de ED):
Previsão dos próximos dias epidemiológicos atualizada em 03/01/2021 (Média dos parâmetros de 20 runs do algoritmo de ED):
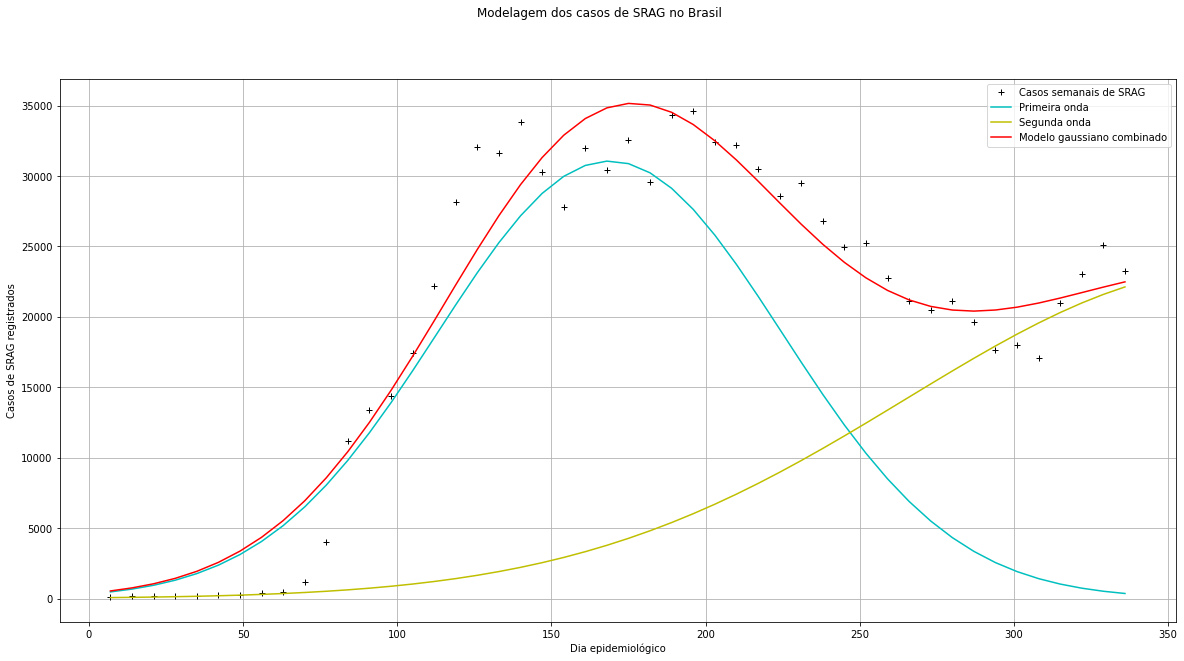
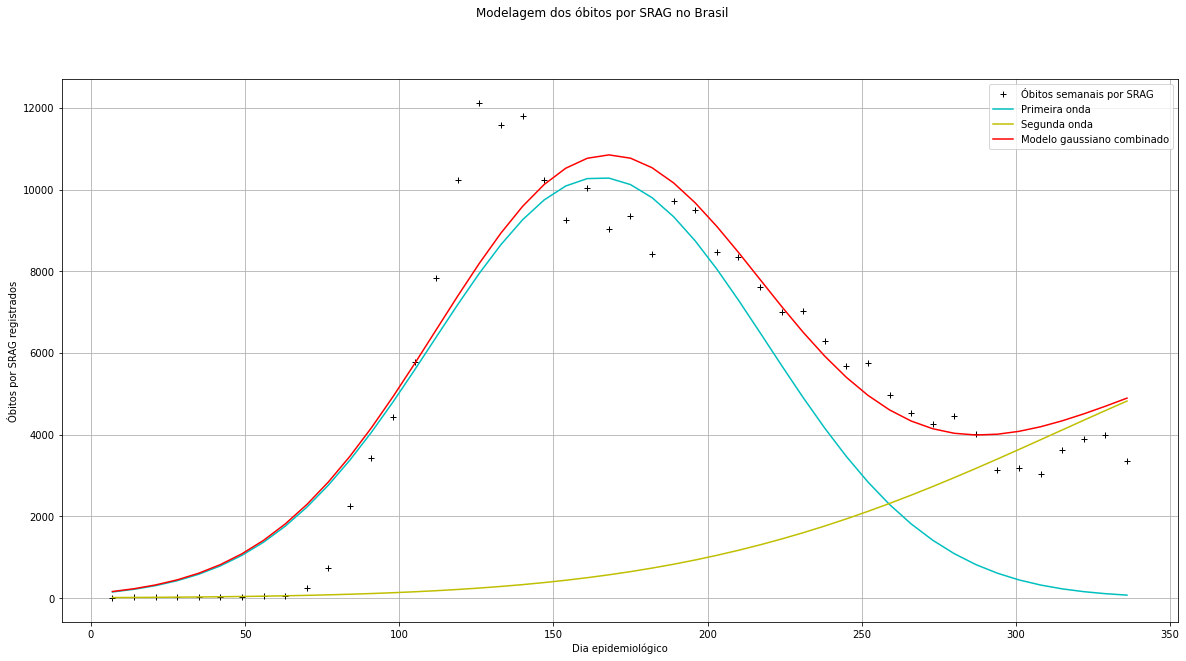
Uma forma de tentarmos prever o comportamento da segunda onda de COVID-19 é analisando os casos e óbitos por Síndrome Respiratória Aguda Grave, já que devido à subnotificação de casos e mortes no Brasil, é possível tentar estimar, por exemplo, óbitos subnotificados por COVID-19 através da análise dos dados de SRAG.
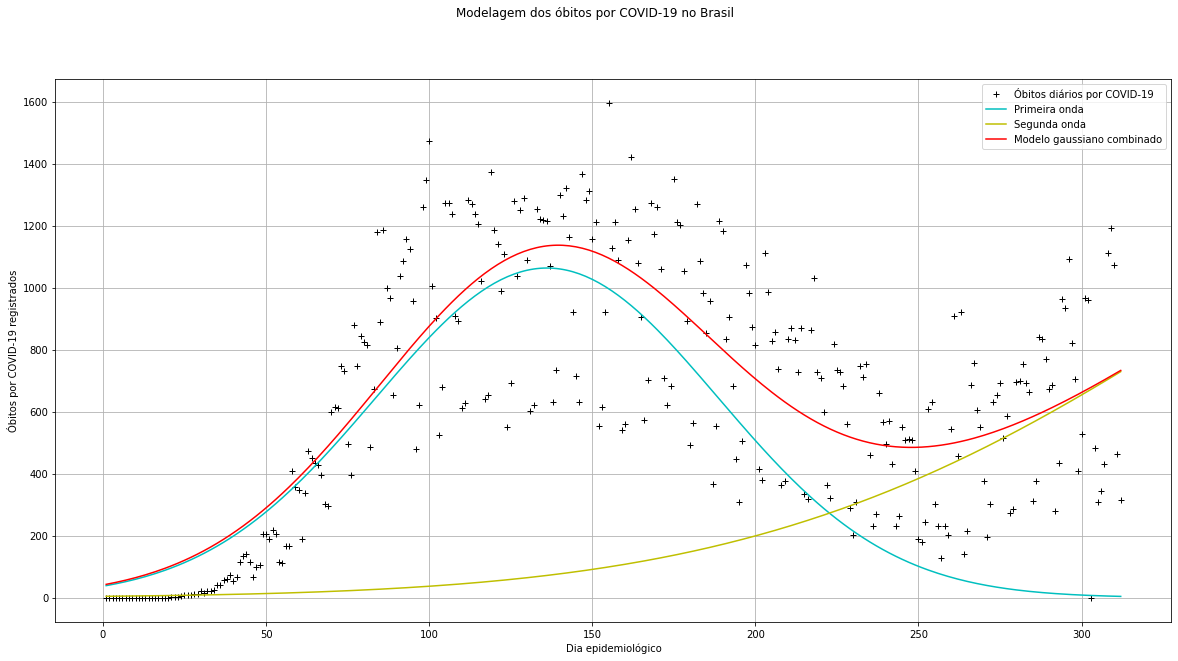
Ajuste feito em 03/01/2021 (Média dos parâmetros de 10 runs do algoritmo de ED):
Ajuste atualizado em 03/03/2021 (Média dos parâmetros de 10 runs do algoritmo de ED):
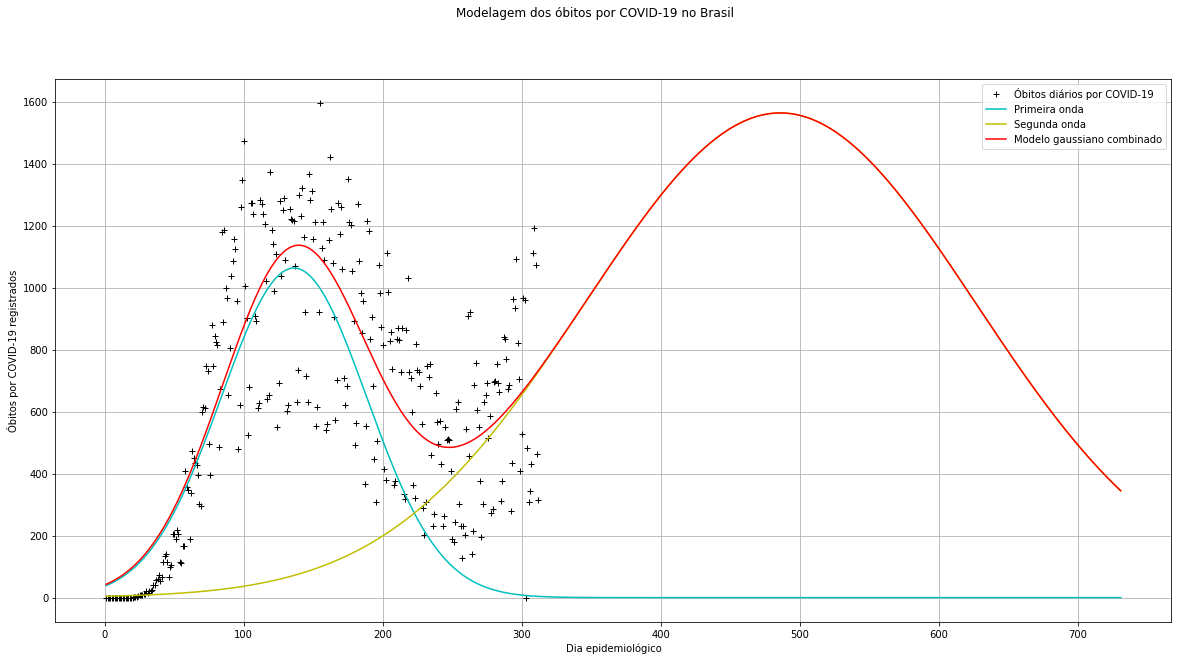
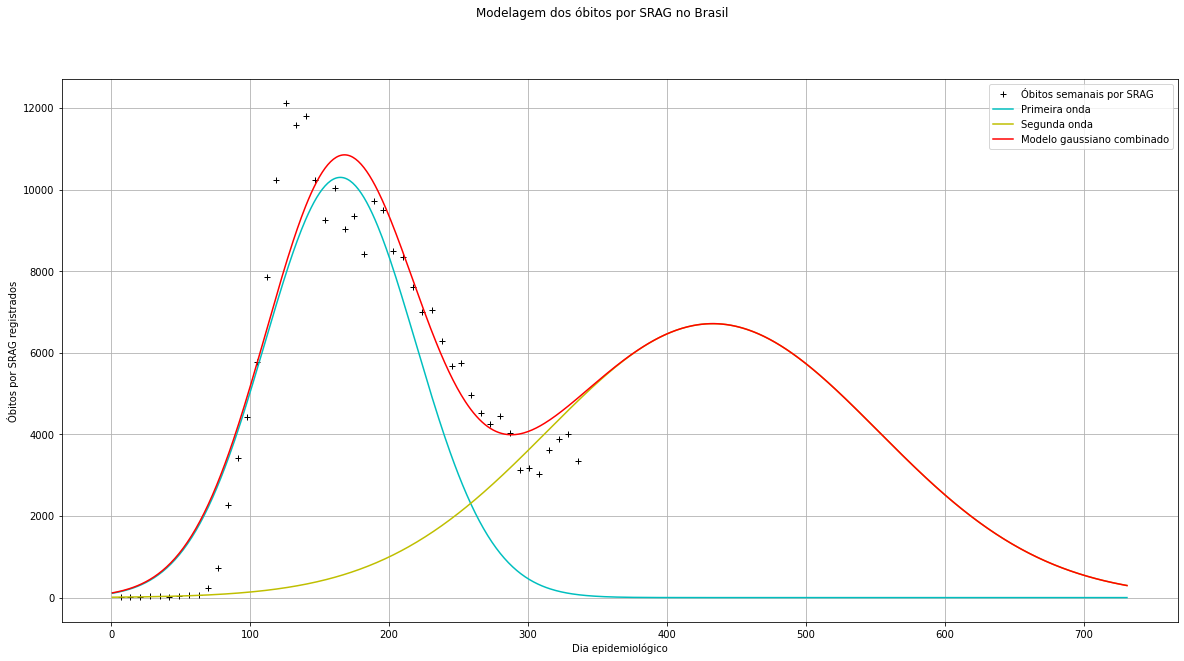
Previsão dos próximos dias epidemiológicos feita em 03/01/2021 (Média dos parâmetros de 10 runs do algoritmo de ED):
Previsão dos próximos dias epidemiológicos atualizado em 03/03/2021 (Média dos parâmetros de 10 runs do algoritmo de ED):
Ajuste feito em 03/01/2021 (Média dos parâmetros de 5 runs do algoritmo de ED):
Ajuste atualizado em 03/03/2021 (Média dos parâmetros de 5 runs do algoritmo de ED):
Previsão dos próximos dias epidemiológicos feita em 03/01/2021 (Média dos parâmetros de 5 runs do algoritmo de ED):
Previsão dos próximos dias epidemiológicos atualizada em 03/01/2021 (Média dos parâmetros de 5 runs do algoritmo de ED):
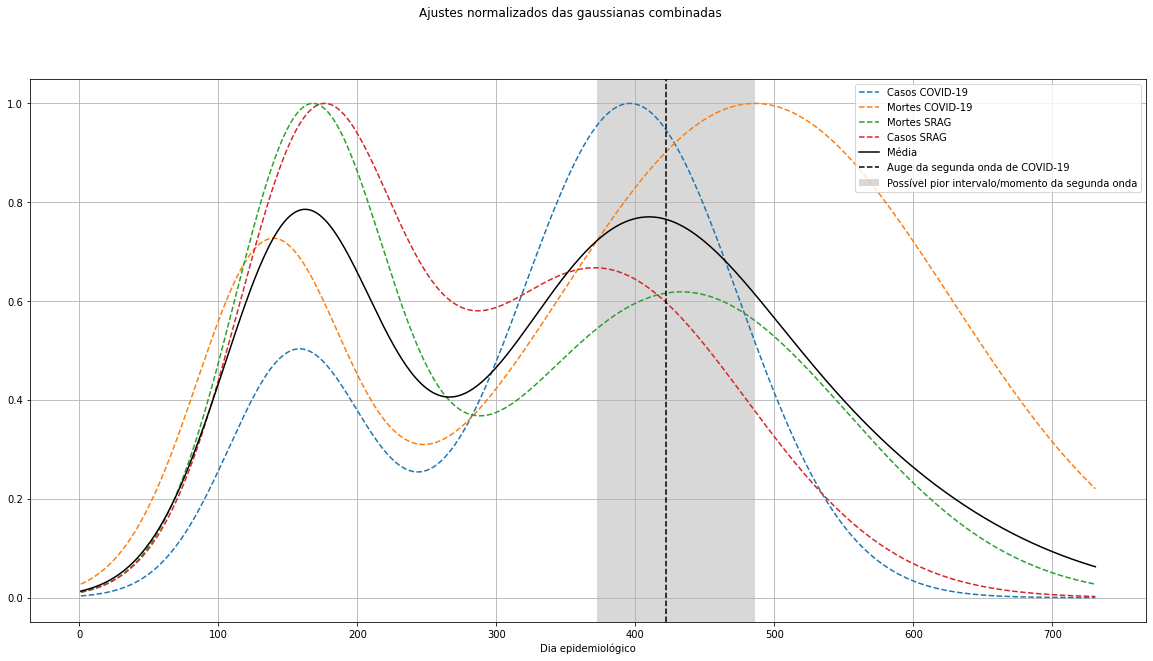
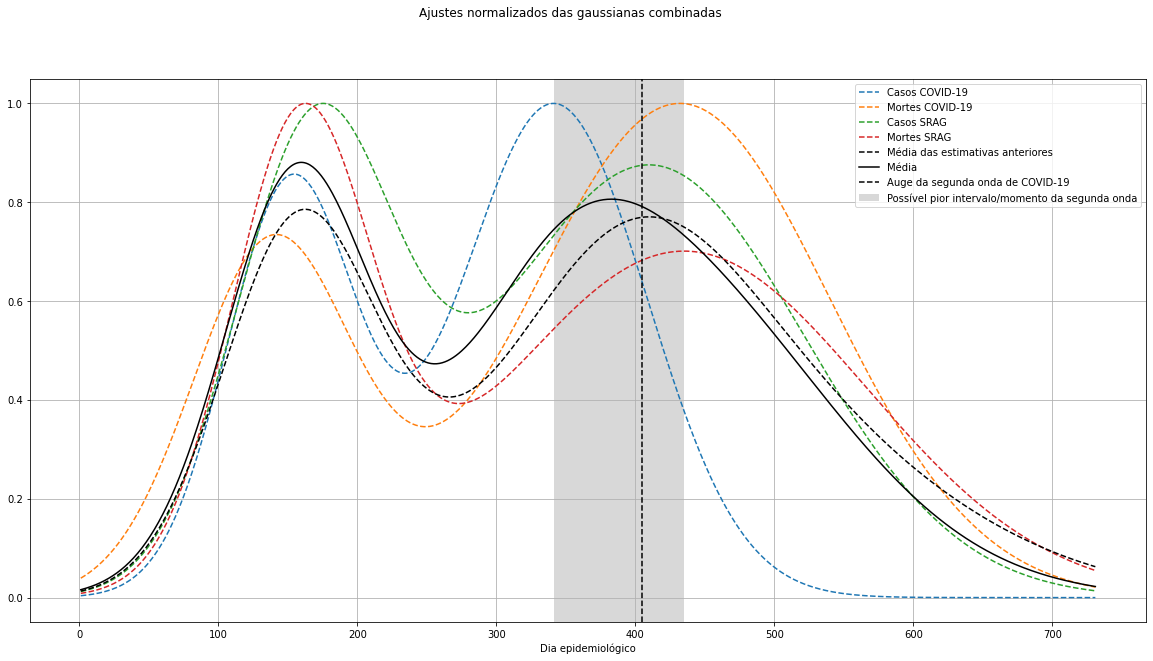
Para tentarmos visualizar o comportamento do auge da segunda onda de COVID-19 no Brasil podemos plotar as curvas correspondentes às gaussianas combinadas dos ajustes anteriores de forma normalizada:
Normalizações feitas em 03/01/2021:
Normalizações atualizadas em 03/03/2021:
Em nosso modelo, o parâmetro βi representa o centro do máximo de cada curva gaussiana, assim, podemos ver que, segundo nossa estimativa:
Estimativas feitas em 03/01/2021:
- 13 de abril (aproximadamente) será o dia com mais casos de COVID-19 registrados na segunda onda;
- 12 de julho (aproximadamente) será o dia com mais óbitos por COVID-19 registrados na segunda onda;
- 20 de março (aproximadamente) será o dia com mais casos de SRAG registrados na segunda onda;
- 20 de maio (aproximadamente) será o dia com mais óbitos por SRAG registrados na segunda onda;
- Em média, 9 de maio (aproximadamente) será o "pior dia" da segunda onda, considerando todos os fatores anteriores.
Estimativas atualizadas em 03/03/2021:
- 17 de fevereiro (aproximadamente) foi o dia com mais casos de COVID-19 registrados na segunda onda;
- 19 de maio (aproximadamente) será o dia com mais óbitos por COVID-19 registrados na segunda onda;
- 27 de abril (aproximadamente) será o dia com mais casos de SRAG registrados na segunda onda;
- 22 de maio (aproximadamente) será o dia com mais óbitos por SRAG registrados na segunda onda;
- Em média, 22 de abril (aproximadamente) será o "pior dia" da segunda onda, considerando todos os fatores anteriores.
OBS: Considerando 13 de março de 2020 como o primeiro dia epidemiológico do Brasil
Não. O Teorema Central do Limite nos garante que a distribuição amostral, quando a amostra aumenta, se aproxima de uma distribuição normal, ou, distruibuição gaussiana. Dada a evolução dos pontos da série temporal proveniente dos dados gerados por fenômenos antropológicos e biológicos relacionados ao COVID-19, pode-se pensar que uma curva gaussina poderia ajustar muito bem estes pontos. Porém, as variáveis do fenômeno que geram os respectivos dados não são independentes, desta forma, não podem ser aproximados facilmente por este tipo de função. Necessitaríamos de algo um pouco mais sofisticado, como uma gaussiana modificada, o que faria, por exemplo, que a curva ajustasse os pontos iniciais de nossos datasets, o que claramente não acontece em nosso casos, e pode ser visualizado nos gráficos.
Outro aspecto a se atentar, é que talvez não tenhamos dados o suficiente da segunda onda para inferirmos um comportamento à longo prazo, principalmente nas previsões utilizando dados de SRAG, que parecem estar desatualizados e/ou subnotificados nos datasets investigados.
-
"MonitoraCovid-19" https://bigdata-covid19.icict.fiocruz.br/; (Visualizado em: 03/01/2021 nos primeiros ajustes e 03/03/2021 nos ajustes maisrecentes) (Daqui foram obtidos os datasets utilizados.
-
"InfoGripe" http://info.gripe.fiocruz.br/; (Visualizado em: 16/05/2020) (Onde os dados são divulgados originalmente. Daqui é possível obter datasets mais robustos com registros até o ano de 2009).