Μία από τις σημαντικότερες εξελίξεις των τελευταίων δεκαετιών ήταν η αποκρυπτογράφηση του ανθρώπινου γονιδιώματος. Οι τεχνικές που χρησιμοποιήθηκαν στην προσπάθεια αυτή μπορούν τώρα να χρησιμοποιηθούν για τη διερεύνηση γενετικών ασθενιών, τον εντοπισμό μεταλλάξεων, την κατανόηση γονιδιωμάτων αρχαίων οργανισμών που έχουν εκλείψει, και σε πλήθος άλλες εφαρμογές.
Τα γονίδια κωδικοποιούνται στο DNA, ένα μεγάλο οργανικό μόριο το οποίο αποτελείται από διπλές αλυσίδες τεσσάρων βάσεων: κυτοσίνη (cytosine, C), γουανίνη (guanine, G), αδενίνη (adenine, A), και θυμίνη (thymine, T). Κάθε μία από τις δύο αλυσίδες αποτελείται από μία σειρά βάσεων, π.χ. ACCGTATAG. Η άλλη αλυσίδα αποτελείται από βάσεις που συνδέονται μία προς μία με τις βάσεις της πρώτης αλυσίδας, με τους κανόνες: A-T, C-G. Έτσι αν η μία αλυσίδα είναι η ACCGTATAG, η άλλη θα είναι TGGCATATC.
Προκειμένου να βρούμε τη σύνθεση μιας αλυσίδας άγνωστου DΝA, εργαζόμαστε ως εξής. Με τεχνικές μοριακής βιολογίας δημιουργούμε πολλά αντίγραφα της αλυσίδας και τα σπάμε σε μικρά θραύσματα, για παράδειγμα θραύσματα με τρεις βάσεις το καθένα. Τέτοιου είδους θραύσματα μπορούμε εύκολα να τα ταυτοποιήσουμε με ειδικές συσκευές. Έτσι καταλήγουμε να έχουμε ένα σύνολο γνωστών θραυσμάτων. Το πρόβλημα που έχουμε να αντιμετωπίσουμε τότε είναι να συναρμολογήσουμε τα, γνωστά πλέον θραύσματα, στην αλυσίδα του DNA, οπότε θα ξέρουμε ακριβώς τη σύνθεσή της.
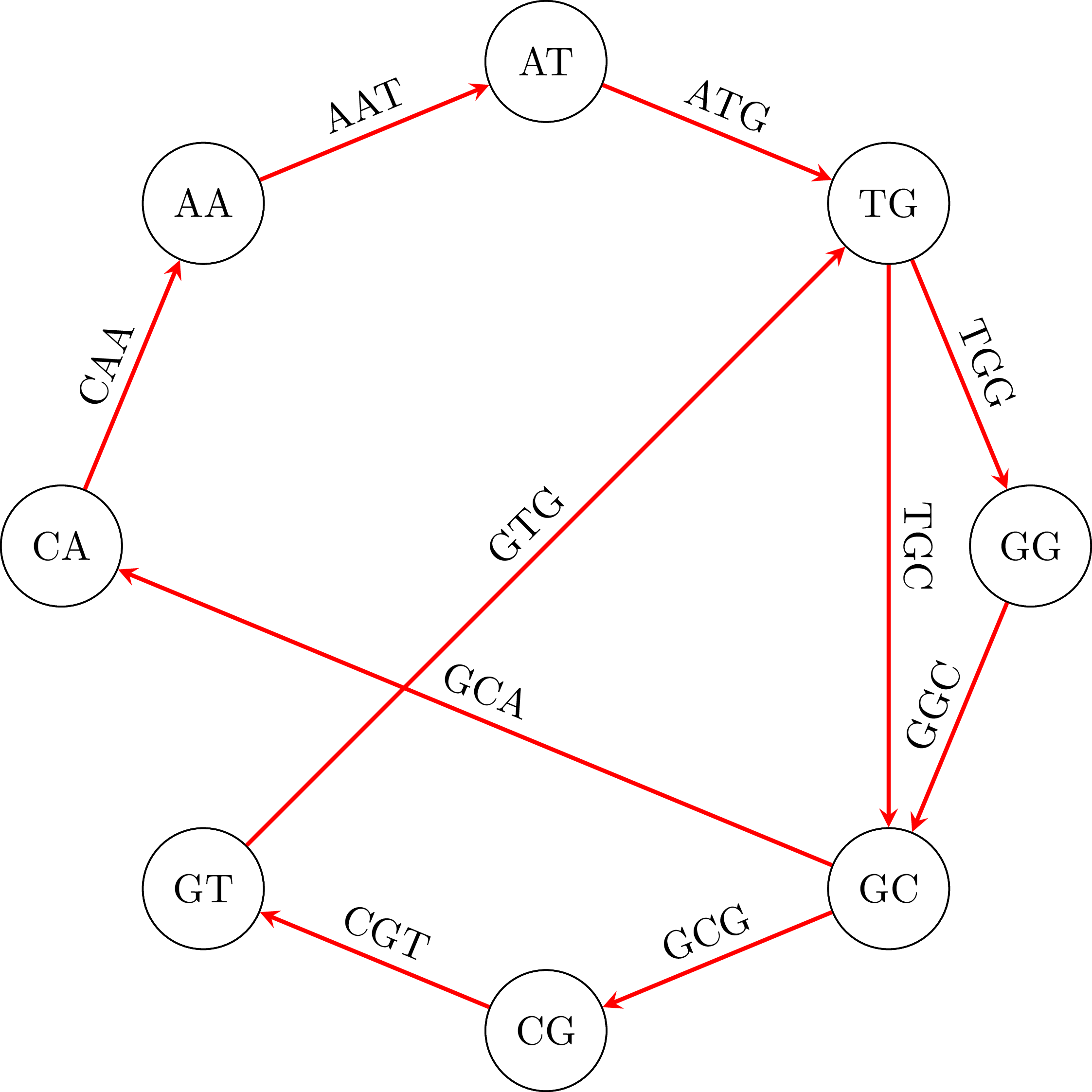
Έστω λοιπόν ότι έχουμε τα παρακάτω θραύσματα, ή πολυμερή (polymers) όπως ονομάζονται: GTG, TGG, ATG, GGC, GCG, CGT, GCA, TGC, CAA, AAT, ATG, AAT. Το κάθε ένα από αυτά έχει μήκος 3, γενικότερα όμως θα μπορούσαν να έχουν μήκος k. Για να βρούμε από ποια ακολουθία DNA προέκυψαν, φτιάχνουμε ένα γράφο, στον οποίο κορυφές είναι τα πολυμερή με μήκος 2 (ή γενικότερα k-1) τα οποία προκύπτουν από τα πολυμερή μεγέθους 3, παίρνοντας για κάθε ένα πολυμερές μήκους 3 (k) τα πρώτα 2 (k-1) και τα τελευταία 2 (k-1) πολυμερή. Έτσι από το GTG θα πάρουμε το GT και το TG, από το TGG θα πάρουμε το TG και το GG, κ.λπ. Στον γράφο προσθέτουμε μία ακμή για κάθε ένα από τα αρχικά πολυμερή μεγέθους 3 (k), από τα οποία προέκυψαν οι εκάστοτε δύο κορυφές. Στην ακμή δίνουμε το όνομα του αρχικού πολυμερούς. Έτσι, από το ATG πήραμε τις κορυγές ΑΤ και TG και τις συνδέουμε με μία ακμή, την οποία ονομάζουμε ATG. Στην παρακάτω εικόνα φαίνεται ο γράφος που προκύπτει στο παράδειγμά μας.
Στη συνέχεια, για να βρούμε την αρχική σελίδα DNA αρκεί να βρούμε ένα μονοπάτι μέσα στο γράφο το οποίο να επισκέπτεται όλες τις ακμές ακριβώς μία φορά. Ένα τέτοιο μονοπάτι ονομάζεται μονοπάτι Euler (Eulerian path) και υπάρχει αν και μόνο αν κάθε έχει ίδιο αριθμό εισερχομένων (in-degree) και εξερχομένων (out-degree) συνδέσμων, και όλες οι κορυφές ανήκουν σε μία συνιστώσα.
Για να εντοπίσουμε το μονοπάτι που θέλουμε στο γράφο που κατασκευάσαμε, χρησιμοποιούμε τον αλγόριθμο του Hierholzer:
- Επιλέγουμε έναν οποιοδήποτε κόμβο εκκίνησης,
v. - Προχωράμε από κόμβο σε κόμβο μέχρι να επιστρέψουμε στον
v. Το μονοπάτι που βρήκαμε μέχρι αυτή τη στιγμή δεν είναι απαραίτητο να καλύπτει όλες τις ακμές του γράφου. - Για όσο υπάρχει μία κορυφή
uπου ανήκει στο μονοπάτι που έχουμε βρει αλλά έχει ακμές που δεν ανήκουν στο μονοπάτι:- Ξεκινάμε ένα άλλο μονοπάτι από το
u, χρησιμοποιώντας ακμές που δεν έχουμε χρησιμοποιήσει μέχρι τώρα, μέχρι να επιστρέψουμε στοu. Τότε συνδέουμε αυτό το μονοπάτι με το μονοπάτι που έχουμε ήδη βρει.
- Ξεκινάμε ένα άλλο μονοπάτι από το
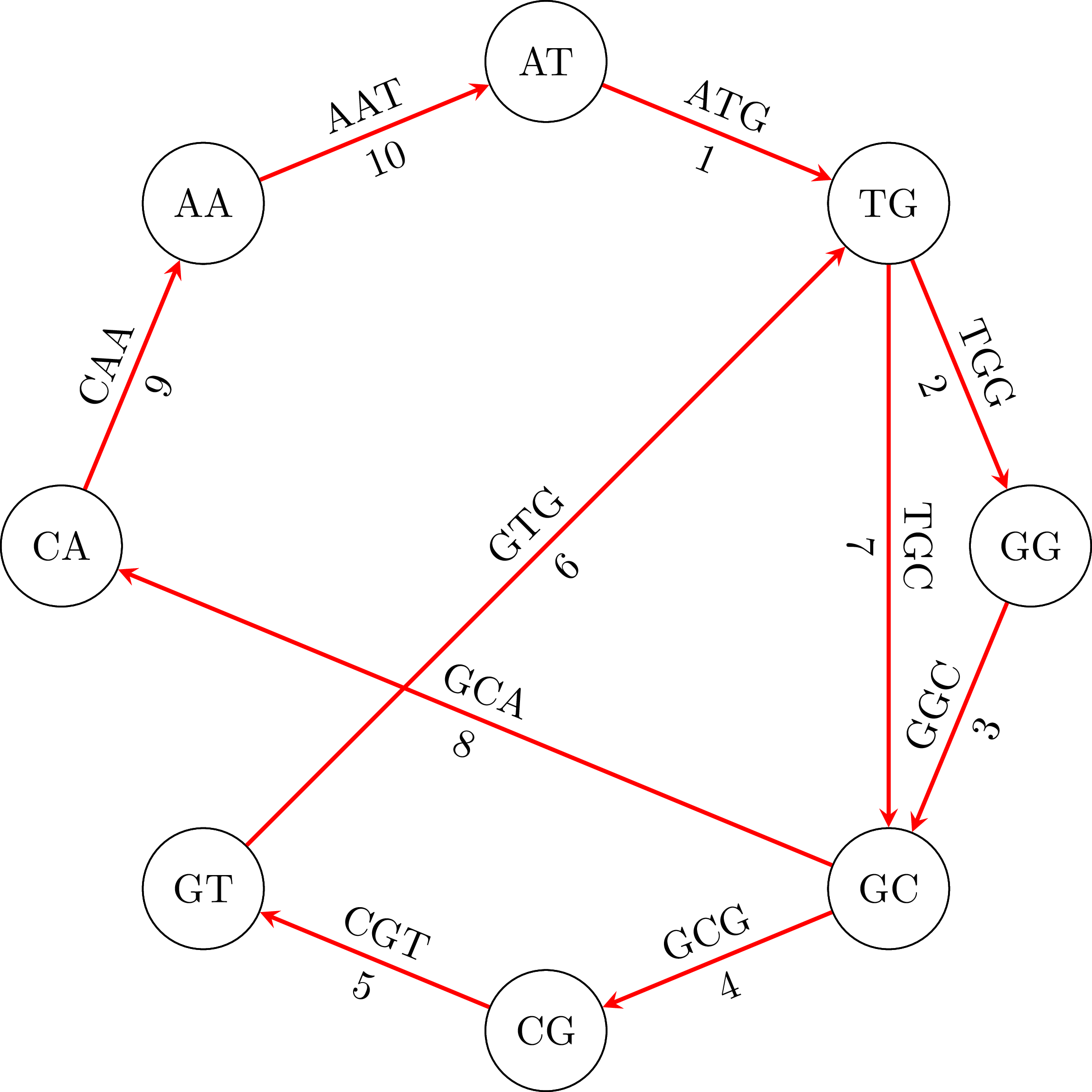
Εάν χρησιμοποιήσουμε τον αλγόριθμο αυτό στον παραπάνω γράφο, θα βρούμε το μονοπάτι Euler που εμφανίζεται στην παρακάτω εικόνα:
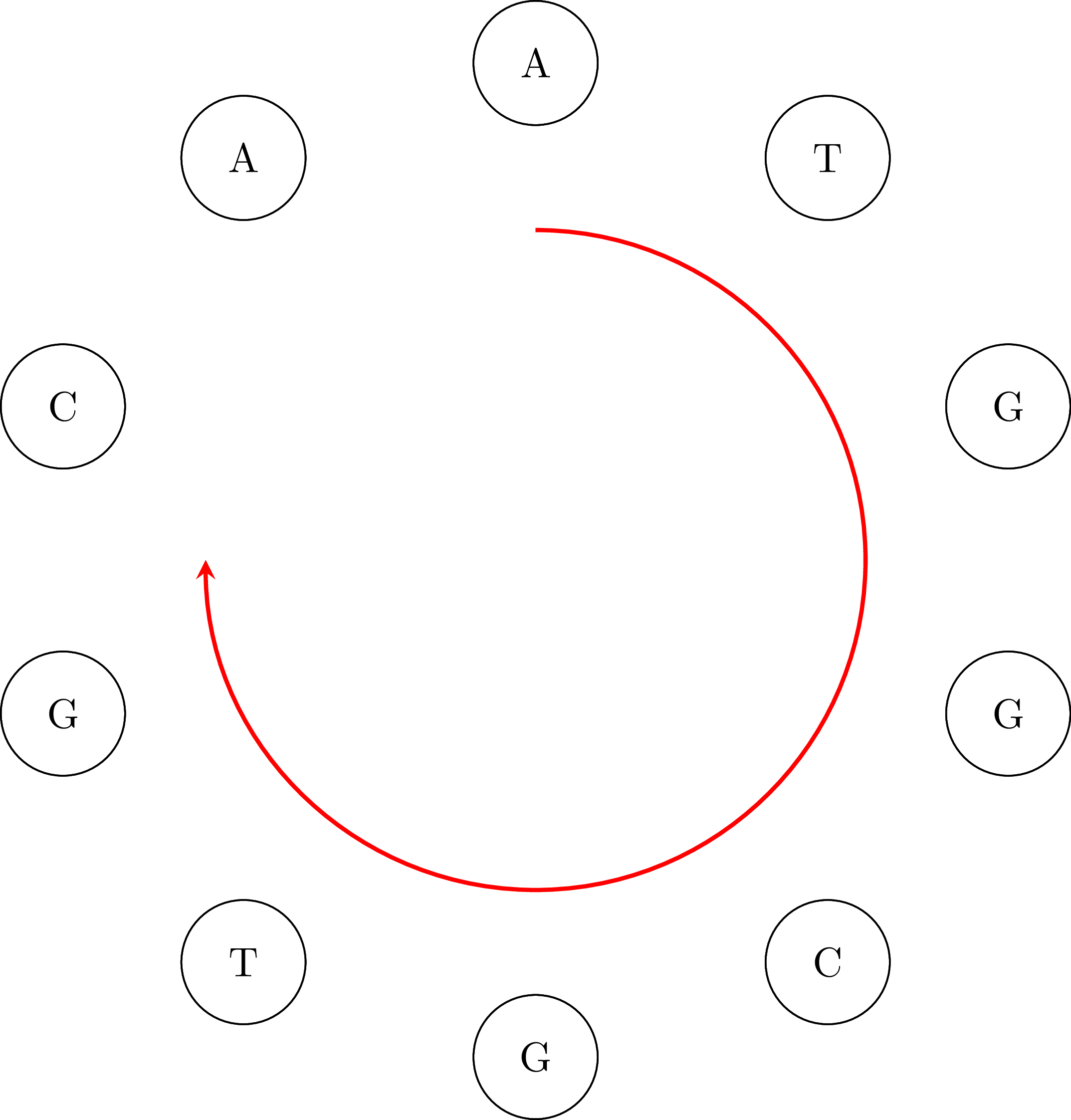
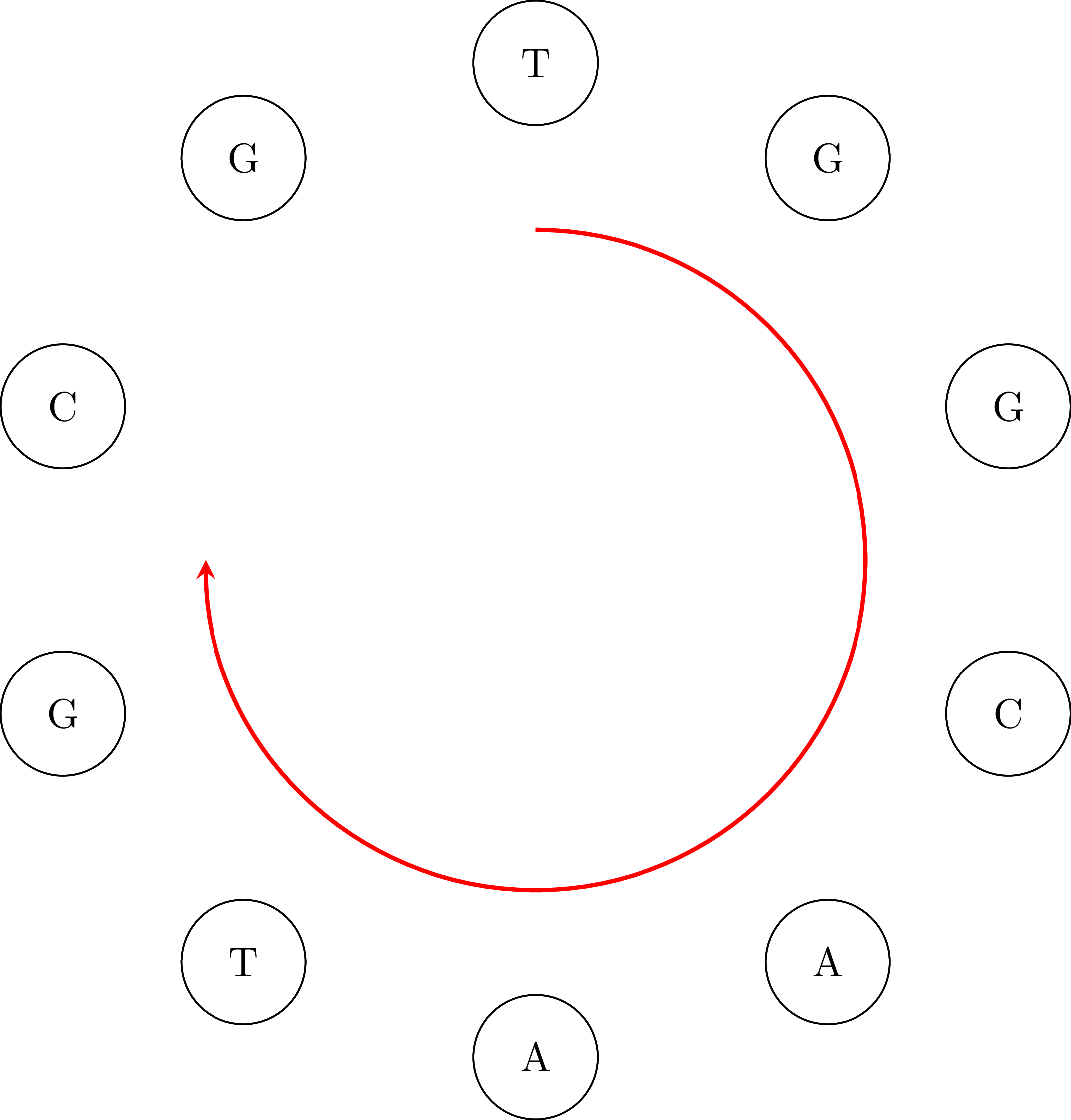
Διατρέχοντας τότε το μονοπάτι και ενώνοντας τους κόμβους, κρατώντας μόνο μία φορά την κοινή τους βάση, παίρνουμε την ακολουθία DNA ATGGCGTGCA. Προσέξτε ότι η ακολουθία είναι κυκλική: ο κόμβος ΑΑ υπάρχει στην ακολουθία ανακυκλώνοντας από το τέλος στην αρχή, άρα θα μπορούσαμε να πάρουμε και την ακολουθία GGCGTGCAAT, ή οποιαδήποτε άλλη που προκύπτει από την περιστροφή της πρώτης ακολουθίας. Επίσης, αναλόγως από το πού ξεκινάμε να διατρέχουμε το μονοπάτι και τι επιλογές κάνουμε όταν έχουμε περισσότερους από έναν εξερχόμενους συνδέσμους, η ακολουθία μπορεί να εμφανίζεται ότι αρχίζει και τελειώνει αλλού. Για παράδειγμα, αν ξεκινήσουμε από τον TG μπορούμε να πάρουμε την ακολουθία TGGCAATGCG, ή οποιαδήποτε άλλη που προκύπτει από την περιστροφή αυτής της ακολουθίας. Αυτό δεν μας ενοχλεί. Οι παρακάτω εικόνες δείχνουν τον κυκλικό χαρακτήρα της ακολουθίας.
Σκοπός της εργασίας είναι ακριβώς η κατασκευή προγράμματος που θα συναρμολογεί DNA με βάση θραύσματα που θα του δίνονται.
Κάθε φοιτητής θα εργαστεί στο προσωπικό του αποθετήριο στο GitHub. Για να αξιολογηθεί μια εργασία θα πρέπει να πληροί τις παρακάτω προϋποθέσεις:
- Όλη η εργασία θα πρέπει να βρίσκεται σε έναν κατάλογο
assignment-2016-2μέσα στο αποθετήριο του φοιτητή. - Το πρόγραμμα θα πρέπει να έχει όνομα
dna_assembly.py. - Εννοείται ότι δεν επιτρέπεται η χρήση έτοιμων βιβλιοθηκών γράφων ή τυχόν έτοιμων υλοποιήσεων των αλγορίθμων, ή τμημάτων αυτών.
- Το πρόγραμμα θα μπορεί να καλείται ως εξής:
python dna_assembly.py fragments_file
Η παράμετρος fragments_file δίνει το όνομα του αρχείου στο οποίο είναι αποθηκευμένα τα θραύσματα (αυτό δεν σημαίνει ότι το αρχείο ονομάζεται ντε και καλά fragments_file, ο χρήστης μπορεί να δίνει οποιοδήποτε όνομα). Το αρχείο θα είναι της μορφής:
ATG
GTG
TGG
GGC
GCG
CGT
GCA
TGC
CAA
AAT
ATG
AAT
...
δηλαδή θα έχει ένα πολυμερές ανά γραμμή.
Προσοχή: αν η έξοδος δεν είναι ακριβώς σύμφωνη με την περιγραφή της, η εργασία δεν θα μπορεί να αξιολογηθεί. Το πρόγραμμα θα πρέπει να εμφανίζει στην έξοδο την ακολουθία DNA που μπόρεσε να εντοπίσει. Έτσι στο παράδειγμά μας η έξοδος θα είναι:
ATGGCGTGCA
ή, όπως αναφέρθηκε παραπάνω, κάποια άλλη ισοδύναμή της (αφού είναι κυκλική), όπως:
TGGCAATGCG
ή
AATGGCGTGC
κ.λπ.
- Αν δώσετε το αρχείο fragment_file_2.txt θα πάρετε την ακολουθία:
AGTGGACCATGTATACTTCA
ή κάποια άλλη ισοδύναμή της, όπως π.χ.:
TGTACTTCATATGGACCAAG
- Αν δώσετε το αρχείο fragment_file_3.txt θα πάρετε την ακολουθία:
ATCTCAGACTTACACCATATGG
ή κάποια άλλη ισοδύναμή της, όπως π.χ.:
TCTCAGACTTACACCATATGGA
- Αν δώσετε το αρχείο fragment_file_4.txt θα πάρετε την ακολουθία:
GACTACCTGGTCTCGATCACGGA
ή κάποια άλλη ισοδύναμή της, όπως π.χ.:
CGGTCACTCTGGACCTACGAGAT
ή
TACTCGGACGAGATCACCTGGTC
Αν θέλετε να ελέγξετε εσείς ότι το αποτέλεσμα είναι σωστό, μπορείτε να γράψετε μια συνάρτηση η οποία θα ελέγχει ότι:
-
Όλα τα θραύσματα που δίνονται στο αρχείο εισόδου υπάρχουν στην ακολουθία που βρήκατε.
-
Όλα τα θραύσματα που βρίσκονται στην ακολουθία που βρήκατε υπάρχουν στο αρχείο εισόδου.
Καλή Επιτυχία.
-
Phillip E. C. Compeau, Pavel A. Pevzner, and Glenn Tesler, How to apply de Bruijn graphs to genome assembly, Nature Biotechnology, Vol. 29, no. 11, November 2011 (http://www.nature.com/nbt/journal/v29/n11/full/nbt.2023.html).
-
Pavel A. Pevzner, Haixu Tang, and Michael S. Waterman, An Eulerian path approach to DNA fragment assembly, Proceedings of the National Academy of Sciences (PNAS), Vol. 18, no. 17, August 14, 2001 (http://www.pnas.org/content/98/17/9748.long).