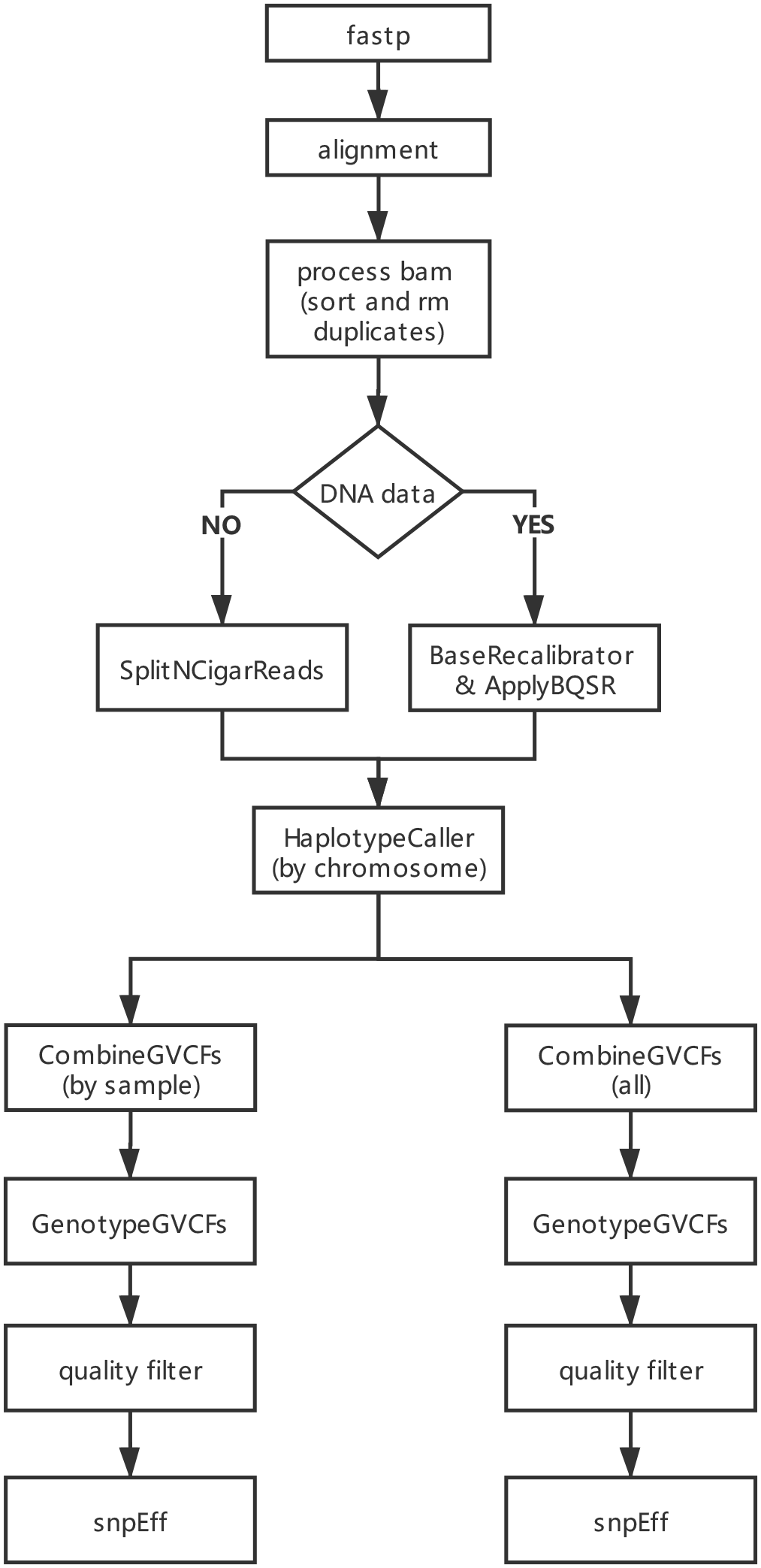
# quick start
nextflow run main.nf \
--reads 'fq_dr/*_R{1,2}.fastq.gz' \ # *通配样品名称
--outdir result_dir \
--known_vcf known.vcf.gz \ # 已知 snp 位点 vcf 文件,需要建立索引
--data_type exome \ # 数据类型 [exome, reseq, rnaseq],默认为外显子数据
--run_snp \ # 运行整个 snp 分析流程, 建议按照 run_fastp, run_align, run_snp 顺序依次运行
-profile slurm,conda \
-resume
基因组名称,默认为 tcuni1.1, 需在 --genomes_base 目录下提前准备
参考基因组,注释文件目录,需要准备的文件参考 conf/genomes.config
rnaseq mapping 软件 [star, hisat2],默认 star
snpEff 数据库路径
snpEff 数据库名称,false 代表不进行 SNP 注释
不生成合并所有样品的 vcf 文件
运行流程至 fastp 部分结束
运行流程至 alignment 部分结束
运行完整 snp 分析流程