I dati delle pubblicazioni periodiche dell'Istituto Superiore di Sanità (ISS) sull'epidemia da SARS-CoV-2 storicizzati e convertiti in formato machine-readable.
Attualmente questo repository contiene i dati storicizzati dei contagi e dei decessi verificatisi in Italia a seguito dell'epidemia da SARS-CoV-2 stratificati per classe di età e per sesso, estratti dal report denominato Dati della Sorveglianza integrata COVID-19 in Italia - Documento esteso, pubblicato in formato PDF con cadenza all'incirca settimanale sul sito istituzionale dell'ISS, nonché il codice per scaricare in locale tutti i predetti documenti ad oggi pubblicati e per effettuare su di essi le operazioni di estrazione dei dati (mediante PDF scraping) e di reshaping degli stessi.
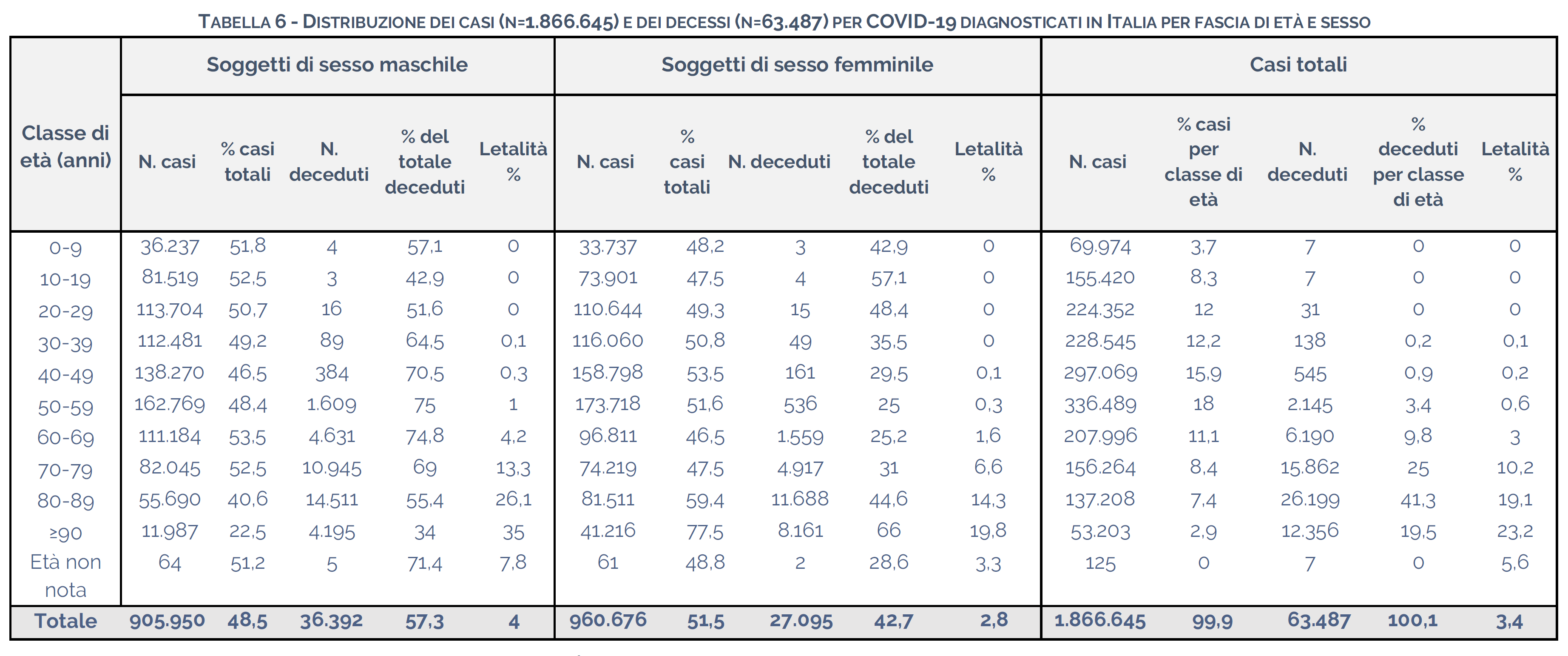
Più precisamente, i dati al momento disponibili in questo repository sono quelli raccolti nella tabella che, nel documento originale in formato PDF, appare come segue:
la quale contiene come detto il numero dei casi diagnosticati e dei decessi, stratificato per classe di età e per sesso, cumulato dall'inizio dell'epidemia alla data di pubblicazione del documento in questione (lo screenshot di cui sopra, che ha valenza puramente esemplificativa, è stato estratto dal bollettino del 16/12/2020).
Per quanto attiene alle operazioni di data reshaping, ogni singola tabella estratta dal corrispondente documento è stata serializzata in un'unica riga della tabella finale, indicizzata tramite la data di pubblicazione del documento, in modo da avere lo storico di tutte le tabelle pubblicate.
Nel tabella finale i dati, privati della struttura gerarchica originale, appaiono quindi distribuiti nelle seguenti colonne:
- la colonna chiave, denominata
date, valorizzata con la data in formato ISO 8601 (yyyy-mm-gg) del documento da cui è stata estratta la riga corrispondente; - le 10 colonne dei casi dei maschi per fascia d'età, denominate
cases_male_<fascia_eta>, dove la sottostringa<fascia_eta>assume (anche nel seguito) i 10 valori:0-9,10-19,20-29,30-39,40-49,50-59,60-69,70-79,80-89,90-; - le 10 colonne dei decessi dei maschi per fascia d'età, denominate
deaths_male_<fascia_eta>; - le 10 colonne dei casi delle femmine per fascia d'età, denominate
cases_female_<fascia_eta>; - le 10 colonne dei decessi delle femmine per fascia d'età, denominate
deaths_female_<fascia_eta>; - le 10 colonne dei casi senza stratificazione per sesso per fascia d'età, denominate
cases_total_<fascia_eta>; - le 10 colonne dei decessi senza stratificazione per sesso per fascia d'età, denominate
deaths_total_<fascia_eta>;
per un totale di 40 colonne (esclusa la colonna chiave).
Per quanto riguarda gli altri dati della tabella originale (percentuali e letalità), si è deciso di non acquisirli in quanto funzionalmente dipendenti dai dati predetti (si è fatta un'eccezione per le sole colonne dei totali sull'età) e quindi da essi derivabili, peraltro in maniera piuttosto semplice: si consulti il paragrafo Cookbook.
La tabella finale sopra descritta è presente nella directory data/ col nome di italy_cases_deaths_by_age_sex.csv e la sua struttura può essere immediatamente compresa anche tramite la semplice ispezione visiva della renderizzazione che ne offre GitHub.
Oltre alla predetta tabella, sempre nella directory data/, sono presenti anche altre due tabelle, denominate italy_cases_deaths_by_age_sex_interp_linear.csv e italy_cases_deaths_by_age_sex_interp_cubic.csv, le quali contengono i dati originali interpolati alla frequenza giornaliera, nel primo caso con interpolazione lineare e nel secondo con interpolazione cubica (ottenute mediante il metodo DataFrame.interpolate della libreria Python pandas). Queste ultime due tabelle risultano utili nel calcolo dei delta, in quanto le differenze calcolate sui dati originali, non essendo questi ultimi equispaziati nel tempo (si veda il punto 1 del paragrafo Limitazioni), risulterebbero riferite a intervalli di tempo di lunghezza diversa.
Per ispezionarne visivamente la struttura, le tabelle sopra descritte sono accessibili ai seguenti link:
- italy_cases_deaths_by_age_sex.csv
- italy_cases_deaths_by_age_sex_interp_linear.csv
- italy_cases_deaths_by_age_sex_interp_cubic.csv
I dati soffrono di alcune limitazioni, dovute alle modalità di pubblicazione degli stessi da parte dell'ISS:
- i valori della chiave (la data di pubblicazione) non sono equispaziati: l'ISS ha pubblicato i dati con cadenza grossomodo settimanale ma, di tanto in tanto, tra un documento e il successivo sono trascorsi intervalli più lunghi o più brevi di 7 giorni; per effettuare il calcolo dei delta sullo stesso periodo temporale, si possono utilizzare le due tabelle aggiuntive interpolate alla frequenza giornaliera
italy_cases_deaths_by_age_sex_interp_linear.csveitaly_cases_deaths_by_age_sex_interp_cubic.csvsopra descritte, oppure si può provvedere ad effettuare autonomamente interventi di interpolazione o decimazione dei dati originali; - i valori interpolati nelle due predette tabelle estese non sono, in generale, degli interi (e sono pertanto di tipo di tipo float), mentre, trattandosi di conteggi, ci si aspetterebbe degli interi: la cosa non dovrebbe costituire un problema nelle rappresentazioni grafiche, ma potrebbe esserlo in altri utilizzi dei dati, nel qual caso si raccomanda di eseguire le opportune operazioni di arrotondamento ad intero (invece i dati della tabella
italy_cases_deaths_by_age_sex.csvsono sempre interi, trattandosi di conteggi prelevati dai documenti originali senza alterazioni); - per giunta anche l'orario di estrazione dal proprio database dei dati pubblicati da parte dell'ISS è difforme da documento a documento, cosa che potrebbe avere un qualche impatto laddove il database dell'ISS venisse alimentato più volte al giorno (cosa che non sappiamo);
- benché i dati siano cumulati e quindi i valori debbano essere non decrescenti nel tempo, in alcune (rare) occasioni si osservano valori decrescenti da una data alla successiva (forse per via di intervenute redistribuzioni di alcuni dati tra le varie fasce d'età);
- i dati imputati nella tabella originale alla riga
Età non notanon sono stati acquisiti in quanto non interpretabili: i valori hanno una notevole erraticità per cui certamente non sono cumulati, ma non è chiaro se si tratti di dati differenziali o (più probabilmente) di dati che diminuiscono di valore per intervenuta attribuzione ad una specifica classe d'età (in ogni caso si tratta di valori piuttosto contenuti); - benché il primo documento di questa serie (pubblicato dall'ISS in data 09/03/2020) sia stato regolarmente scaricato e sia presente anche in questo repository, da esso non è stato possibile acquisire i dati, in quanto la tabella contenuta appare limitata alla sola stratificazione per classe d'età (senza stratificazione per sesso) e, per giunta, con le ultime due classi, che nei documenti successivi sono sempre separate (80-89 e ≥90), che in questo caso appaiono invece raccolte in un'unica classe (≥80); i dati estratti partono pertanto dal documento successivo, riferibile alla data 12/03/2020 (si tratta peraltro di uno dei casi in cui il tempo intercorso tra due documenti successivi è inferiore ai 7 giorni).
Ancorché in forma largamente subottimale, dal 08/12/2020 l'ISS ha iniziato a pubblicare questi stessi dati anche in formato machine-readable, attraverso un file xlsx aggiornato con cadenza giornaliera e reso disponibile su un'apposita pagina.
Tali dati presentano tuttavia almeno un paio di rilevanti limitazioni:
- non vengono storicizzati: il file contiene semplicemente i valori cumulati aggiornati alla data di pubblicazione; a tale proposito va detto che esiste un meritorio progetto indipendente che recupera periodicamente il predetto file e ne storicizza i valori;
- come già detto, i dati partono dai valori cumulati alla data 08/12/2020, quindi non è possibile recuperarne i valori precedenti (che costituisce il motivo principale per cui è nato il presente progetto).
Per quanto ci risulta, il presente progetto è l'unico attraverso il quale sia possibile accedere all'intero archivio storico dei dati dell'ISS in formato machine-readable.
I dati estratti, in formato CSV (con il carattere virgola - nome unicode: comma - come separatore di campo, con le intestazioni di colonna nella prima riga e caratteri del solo sottoinsieme ASCII a-z, -, 0-9, .), possono essere utilizzati direttamente, facendo riferimento ad uno dei seguenti file (corrispondenti alle tabelle sopra descritte):
- https://raw.githubusercontent.com/emazep/Machine-readable-Covid-19-ISS-data/master/data/italy_cases_deaths_by_age_sex.csv
- https://raw.githubusercontent.com/emazep/Machine-readable-Covid-19-ISS-data/master/data/italy_cases_deaths_by_age_sex_interp_linear.csv
- https://raw.githubusercontent.com/emazep/Machine-readable-Covid-19-ISS-data/master/data/italy_cases_deaths_by_age_sex_interp_cubic.csv
che conterranno sempre lo storico di tutti i dati aggiornati all'ultimo documento disponibile, che verrà acquisito il prima possibile, ci si propone entro qualche ora dalla pubblicazione sul sito dell'ISS.
Purtroppo l'acquisizione non può essere del tutto automatizzata, data l'impredicibilità dei nomi dei file pubblicati dall'ISS (che non seguono uno standard uniforme) e della modalità di esposizione dei dati all'interno di ognuno di essi, affetta occasionalmente da variazioni nei metadati o da cambiamenti nella formattazione del documento, che impattano purtroppo sulle euristiche di scraping.
I seguenti snippet fanno riferimento esclusivamente alla libreria Python pandas.
Caricamento di pandas:
import pandas as pdCaricamento dei dati (aggiornati) in un dataframe pandas (con riconoscimento e parsing automatico dell'indice in oggetti di tipo datetime):
SOURCE = 'https://raw.githubusercontent.com/emazep/Machine-readable-Covid-19-ISS-data/master/data/italy_cases_deaths_by_age_sex.csv'oppure, nel caso si desideri utilizzare una delle tabelle interpolate:
# interpolazione lineare
SOURCE = 'https://raw.githubusercontent.com/emazep/Machine-readable-Covid-19-ISS-data/master/data/italy_cases_deaths_by_age_sex_interp_linear.csv'oppure:
# interpolazione cubica
SOURCE = 'https://raw.githubusercontent.com/emazep/Machine-readable-Covid-19-ISS-data/master/data/italy_cases_deaths_by_age_sex_interp_cubic.csv'e quindi:
df = pd.read_csv(SOURCE, index_col=0, parse_dates=True)Se invece si desidera che l'indice sia di tipo date anziché datetime, per il caricamento si può utilizzare il seguente snippet alternativo (solo per pandas ≥ 0.15.0):
df = pd.read_csv(SOURCE, parse_dates=['date'])
df['date'] = df['date'].dt.date
df.set_index('date', inplace=True)Calcolo dei casi e dei decessi totali (maschi + femmine) per ogni classe di età:
AGE_CLASSES = ['0-9', '10-19', '20-29', '30-39', '40-49', '50-59', '60-69', '70-79', '80-89', '90-']
for age_class in AGE_CLASSES:
df['cases_' + age_class + '_TOTAL'] = df['cases_male_' + age_class] + df['cases_female_' + age_class]
df['deaths_' + age_class + '_TOTAL'] = df['deaths_male_' + age_class] + df['deaths_female_' + age_class]Aggiunta di una colonna con i valori incrementali (ovvero le differenze col valore precedente nella colonna, dette anche delta) per ogni colonna del dataframe:
for col in df.columns:
df[col + '_DELTA'] = df[col].diff()Si noti come per il predetto calcolo dei delta sia preferibile utilizzare i dati interpolati, in quanto i dati originali non appaiono purtroppo equispaziati nel tempo, come descritto al punto 1 del paragrafo Limitazioni.
Aggiunta degli andamenti delle percentuali dei delta di casi e decessi sui rispettivi delta totali per ogni classe di età (si assume che siano già stati eseguiti il calcolo dei totali e dei delta di cui ai due snippet precedenti, esattamente in quest'ordine):
total_cases_delta = total_deaths_delta = 0
for age_class in AGE_CLASSES:
total_cases_delta += df['cases_' + age_class + '_TOTAL_DELTA']
total_deaths_delta += df['deaths_' + age_class + '_TOTAL_DELTA']
for age_class in AGE_CLASSES:
df['cases_' + age_class + '_TOTAL_DELTA_PERC'] = (
df['cases_' + age_class + '_TOTAL_DELTA'] / total_cases_delta
) * 100
df['deaths_' + age_class + '_TOTAL_DELTA_PERC'] = (
df['deaths_' + age_class + '_TOTAL_DELTA'] / total_deaths_delta
) * 100Aggiunta di una colonna contenente il CFR (Confirmed Fatality Rate, dato dal rapporto tra i decessi cumulati alla data corrente e i casi cumulati alla data precedente disponibile) per ogni classe d'età, espresso in percentuale (si assume che sia già stato eseguito lo snippet che calcola i totali):
for age_class in AGE_CLASSES:
df['CFR_' + age_class] = (df['deaths_' + age_class + '_TOTAL'] / df['cases_' + age_class + '_TOTAL'].shift()) * 100L'uso degli script non è necessario, a meno che non si intenda scaricare i documenti dell'ISS ed estrarne i dati in autonomia.
Gli script sono stati redatti in codice Python e sono reperibili nella directory script/.
Per installarne i prerequisiti, eseguire:
pip install -r requirements.txtLo script bollettino_sorveglianza_integrata_download.py si incarica di scaricare e salvare in locale i documenti dell'ISS, nonché di normalizzarne i nomi dei file. Lo script al momento non accetta parametri dalla linea di comando, per cui se se ne vuole customizzare il funzionamento è necessario valorizzare opportunamente le variabili globali in testa al codice (sufficientemente documentate). Tuttavia il comportamento di default è quello che dovrebbe andare bene nella maggior parte dei casi: vengono scaricati tutti i documenti pubblicati sul sito dell'ISS, nell'ordine che va dal più recente al più remoto, e salvati in locale nella directory ../original_ISS_documents/bollettino_sorveglianza_integrata/ col nome file opportunamente normalizzato, mentre in caso di successiva esecuzione lo script si interrompe al primo documento trovato che risulta già scaricato (il più recente), in modo da poter essere lanciato periodicamente per scaricare i nuovi documenti senza che venga inutilmente ripetuto anche il download di quelli precedenti.
Il codice per l'estrazione dei dati dai documenti scaricati viene invece fornito come notebook Jupyter (nel file bollettino_sorveglianza_integrata_ETL.ipynb), data la sua natura maggiormente interattiva, dovuta al fatto che, sulla base dei documenti fin qui pubblicati, risulta purtroppo altamente probabile che quelli futuri presentino difformità nella presentazione dei dati tali da richiedere adattamenti nel codice di scraping (adattamenti che si è cercato comunque di limitare alla valorizzazione di un paio di parametri).
I dati in formato machine-readable presenti in questo repository verranno aggiornati entro qualche ora dalla pubblicazione dei nuovi report da parte dell'ISS.
Non si esclude che in futuro possano essere acquisiti anche altri dati dalle pubblicazioni dell'ISS.
Si consultino le segnalazioni aperte per la lista delle feature proposte (e dei problemi noti).
I contributi maggiormente apprezzati riguardano il controllo della correttezza dei dati estratti ma anche eventuali suggerimenti riguardo nuovi dati da estrarre, avendo cura in quest'ultimo caso di fornire i dovuti link e di controllare che non si tratti di dati già disponibili in formato machine-readable altrove (ad esempio sul repository GitHub della Protezione Civile).
Per quanto riguarda i contributi al codice, si prega di attenersi agli standard desumibili dal codice presente. A meno che non si tratti di contributi minimi, la procedura da seguire è:
- forkare il progetto;
- creare un Branch per le vostre Feature (
git checkout -b feature/AmazingFeature); - committare le modifiche/aggiunte apportate (
git commit -m 'Add some AmazingFeature'); - pushare le modifiche sul Branch (
git push origin feature/AmazingFeature); - aprire una Pull Request.
Il codice presente in questo repository è rilasciato sotto licenza MIT. Si consulti il file LICENSE.txt per ulteriori dettagli in merito.
Per quanto attiene ai dati, essi sono di proprietà dell'Istituto Superiore di Sanità, per cui si prega di contattare tale istituzione per le informazioni riguardo la relativa licenza d'uso.
The code in this repository is distributed under the MIT License. See the file LICENSE.txt for more information.
As far as the data, they are owned by the Istituto Superiore di Sanità, so please contact them for any information regarding their licensing terms.
Emanuele Zeppieri
Emanuele Zeppieri - @emazep
Project Link: https://github.com/emazep/Machine-readable-Covid-19-ISS-data