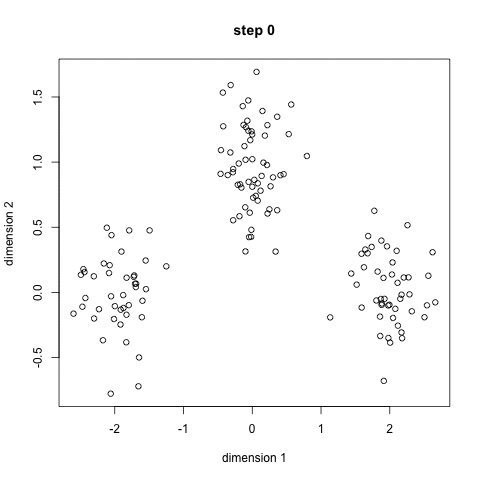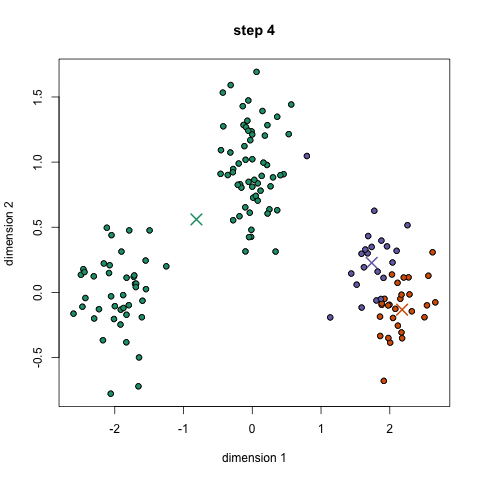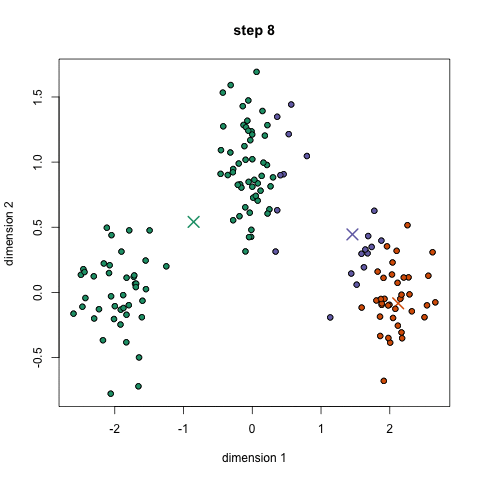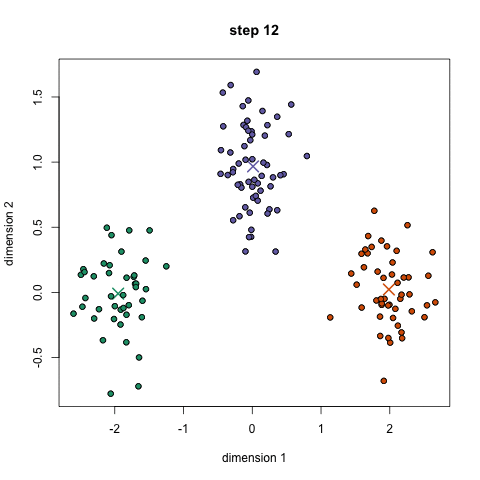
Neste trabalho proposto de Docência Orientada I, será feito a utilização do software RStudio para análise de agrupamento de dados. Será feita a demonstração de como utilizar o RStudio juntamente com os algoritmos: K-means, DBScan e Hierárquico.
O conjunto de dados que será utilizado como exemplo para efetuar a análise de agrupamento, será o Iris Data Set. O qual pode ser encontrado através do link: https://archive.ics.uci.edu/ml/datasets/Iris.
Esse conjunto de dados que utilizaremos, é um dos mais conhecidos e encontrados na literatura sobre reconhecimento de padrões. O Iris Data Set, possui 3 classes (cada classe se refere a um tipo de planta) de 50 amostras cada, totalizando 150 amostras. Esse modelo foi desenvolvido pelo biólogo e estatístico Ronald Fisher.
As informações contidas nesse conjunto de dados são separadas por 4 atributos, entre eles estão:
- Comprimento de sépalas (float);
- Largura de sépalas (float);
- Comprimento de pétalas (float);
- Largura de pétalas (float);
- Classe:
- Iris Setosa;
- Iris Versicolor;
- Iris Virginica.
Levando-se em consideração esses aspectos, usaremos os algoritmos K-means, DBScan e Hierárquico para efetuar o agrupamento dos dados.
O algoritmo K-means é considerado um aprendizado do tipo não supervisionado, isso significa que é necessário somente os dados, sem ser necessário conhecimento prévio das classes. Este algoritmo tem por objetivo encontrar uma alocação dos dados em clusters de maneira que, dentro de cada cluster, os dados estejam o mais próximos possível, assumindo que quanto mais próximos eles estiverem, mais parecidos eles são.
Para utilizar o algoritmo K-means, primeiramente é definido quantos clusters devem ser encontrados. Para isso é definido o K, o valor de K é definido através da dedução de quantos agrupamentos os dados sejam segmentados. É possível utilizar a técnica Elbow method (método do cotovelo) para ajudar a encontrar o número apropriado de clusters em um conjunto de dados. Também é definido o número de iterações, onde as iterações são o número de vezes que o algoritmo repetirá a atribuição do cluster e a movimentação dos centroides.
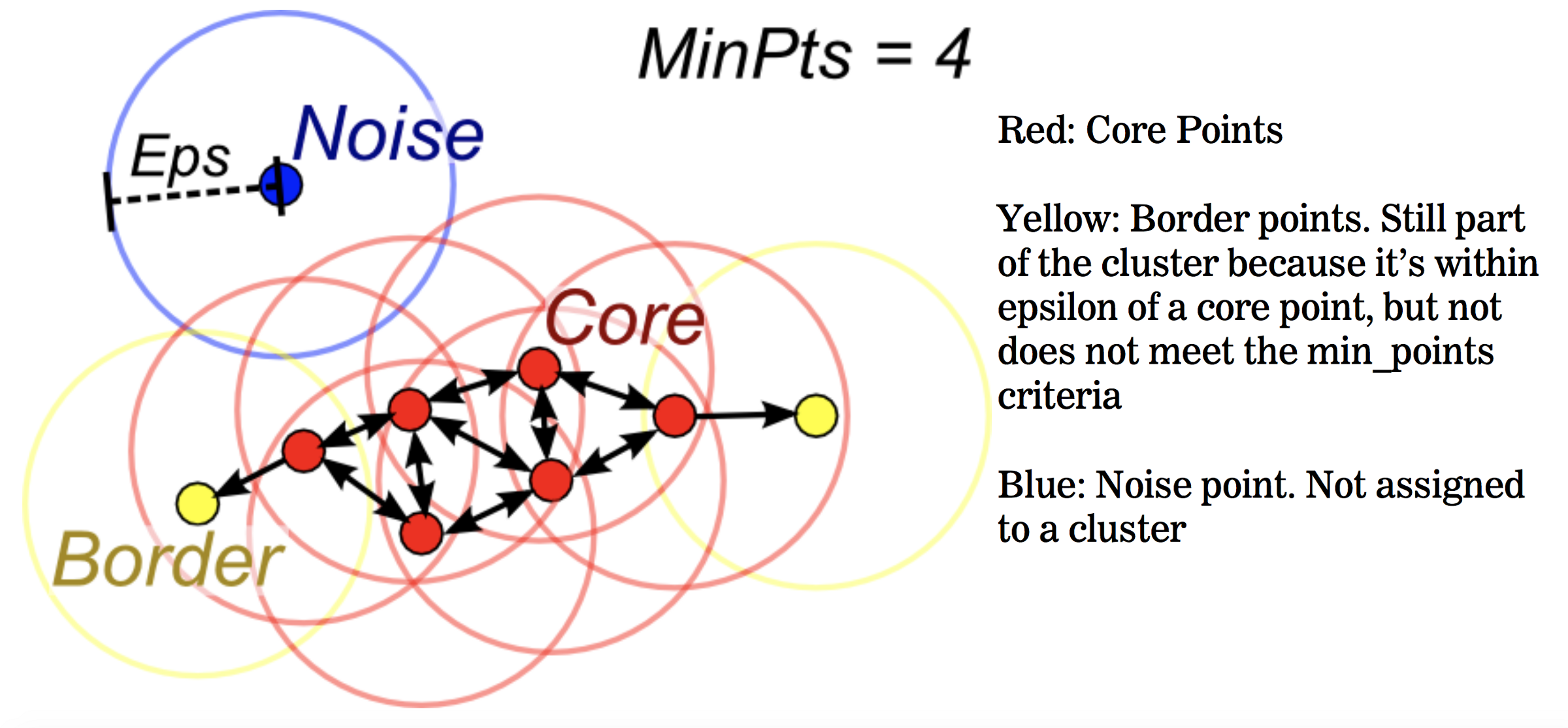
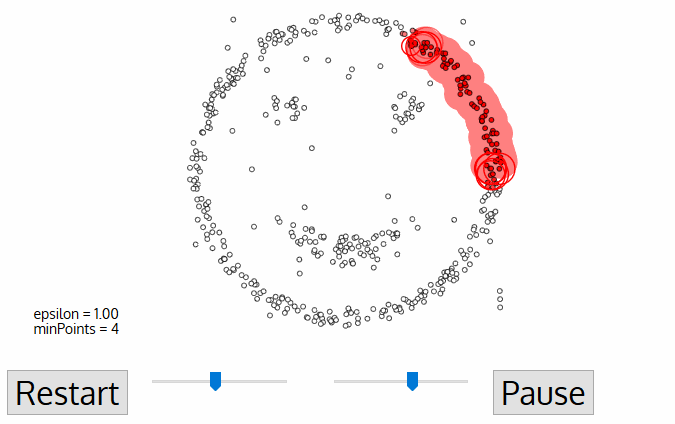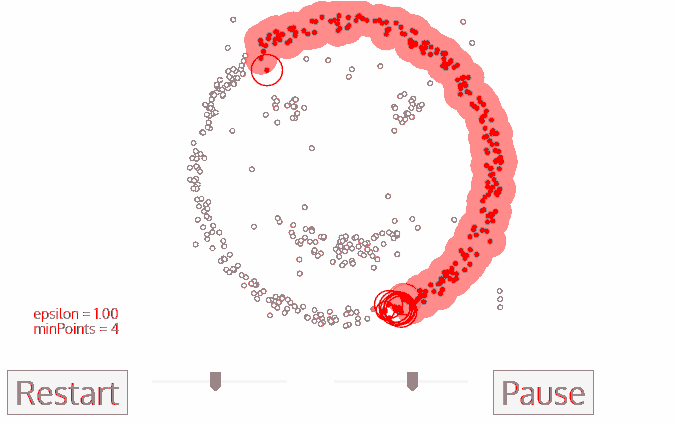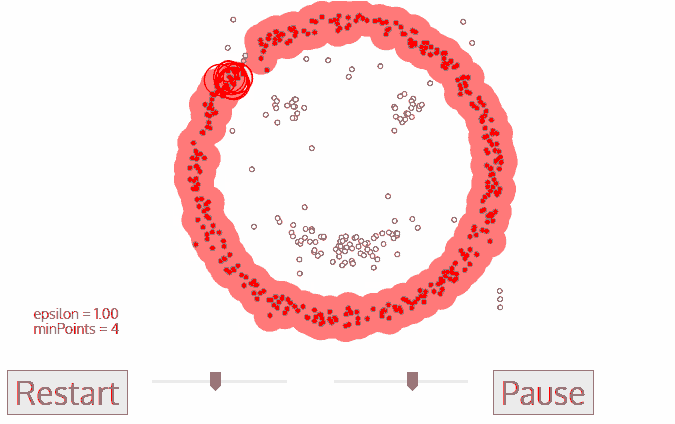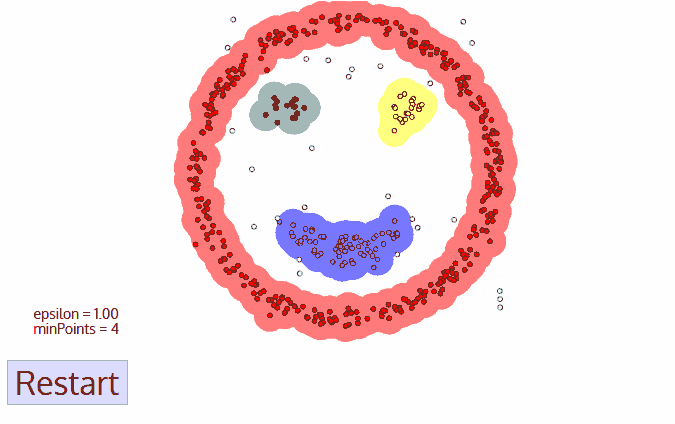
Density-Based Spatial Clustering of Applications with Noise, é o algoritmo baseado em densidade que agrupa pontos que são próximos uns dos outros com base em uma medida de distância (geralmente euclidiana) e um número mínimo de pontos. Esse algoritmo marca pontos que estão em regiões de baixa densidade como outliers e é considerado um aprendizado do tipo não supervisionado.
O funcionamento desse algoritmo funciona da seguinte forma:
- Divide o conjunto de dados em n dimensões;
- Para cada ponto no conjunto de dados, o DBSCAN forma uma forma n-dimensional em torno desse ponto de dados e, em seguida, conta quantos pontos de dados se enquadram nessa forma.
- O DBSCAN conta essa forma como um cluster. O DBSCAN expande iterativamente o cluster, passando por cada ponto individual dentro do cluster e contando o número de outros pontos de dados próximos. Exemplo:
- Vermelho: Pontos principais;
- Amarelo: Pontos de borda. Ainda faz parte do cluster porque está dentro do epsilon de um ponto central, mas não atende aos critérios min_points;
- Azul: Ponto de ruído. Não atribuído a um cluster.
Percorrendo o processo mencionado passo a passo, o DBSCAN começará dividindo os dados em n dimensões. Após o DBSCAN ter feito isso, ele começará em um ponto aleatório (neste caso, vamos supor que ele era um dos pontos vermelhos) e contará quantos outros pontos estão próximos. O DBSCAN continuará esse processo até que nenhum outro ponto de dados esteja por perto e, em seguida, ele procurará formar um segundo cluster.
O DBSCAN requer dois parâmetros:
- eps: distância entre os pontos. Significa que se a distância entre um ponto e outro é menor ou igual ao valor de eps, então estes dois pontos são considerados vizinhos.
- minPoints: número mínimo de pontos necessários para formar uma região densa. Por exemplo, se o minPoints é igual a 5 então são necessários 5 pontos para formar uma região densa. Para que seja determinado o melhor valor para eps, é calculado as distâncias vizinhas mais próximas em uma matriz de pontos. Essa técnica é chamada de knee (joelho), onde ocorre uma mudança acentuada ao longo da curva de distância K.
Nesse algoritmo ao usá-lo é criada uma árvore na qual os objetos são as folhas e os nós internos revelam a estrutura de similaridade dos pontos. Essa árvore é chamada de dendograma. Essa estrutura indica o número de clusters. Eles podem ser classificados em dois tipos: Aglomerativos e Divisivos.
- Métodos Aglomerativos – Nesse caso, todos os elementos começam separados e vão sendo agrupados em etapas, um a um, até que tenhamos um único cluster com todos os elementos. O número ideal de clusters é escolhido dentre todas as opções.
- Métodos Divisivos – No método divisivo todos os elementos começam juntos em um único cluster, e vão sendo separados um a um, até que cada elemento seja seu próprio cluster. Assim como no método aglomerativo, escolhemos o número ótimo de clusters dentre todas as possíveis combinações.
O comportamento deste algoritmo depende como o par de cluster mais próximo é definido, podendo ser:
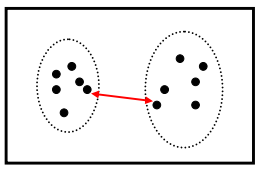
- Single Link: Distância entre dois clusters é a distância entre os pontos mais próximos. Também chamado “agrupamento de vizinhos”;
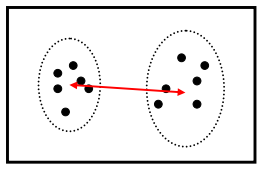
- Average Link: Distância entre clusters é a distância entre os centroides (agrupamento pela média);
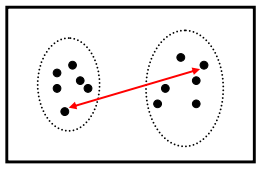
- Complete Link: Distância entre clusters é a distância entre os pontos mais distantes.
Coordenadas Discriminantes é um método algébrico que utiliza coorelações lineares para transformar vários campos em um único que descreva todos os campos involvidos; Esse valor único representa ocorrencias relacionadas aos campos representados.
- Evaristo José Do Nascimento
- Marcelo Garbin
- https://www.devmedia.com.br/data-mining-na-pratica-algoritmo-k-means/4584
- https://medium.com/@paulo_sampaio/entendendo-k-means-agrupando-dados-e-tirando-camisas-e90ae3157c17
- https://rstudio-pubs-static.s3.amazonaws.com/33876_1d7794d9a86647ca90c4f182df93f0e8.html
- https://medium.com/pizzadedados/kmeans-e-metodo-do-cotovelo-94ded9fdf3a9
- http://www.learnbymarketing.com/tutorials/k-means-clustering-in-r-example/
- https://towardsdatascience.com/how-dbscan-works-and-why-should-i-use-it-443b4a191c80
- https://medium.com/@elutins/dbscan-what-is-it-when-to-use-it-how-to-use-it-8bd506293818
- https://towardsdatascience.com/the-5-clustering-algorithms-data-scientists-need-to-know-a36d136ef68
- http://www.sthda.com/english/wiki/print.php?id=246
- http://dcm.ffclrp.usp.br/~augusto/teaching/ami/AM-I-Clustering.pdf
- https://www.datacamp.com/community/tutorials/k-means-clustering-r
- https://discuss.analyticsvidhya.com/t/interpretation-result-of-k-means-algorithm/17750/3
- http://www.sthda.com/english/wiki/wiki.php?id_contents=7940
- https://cran.r-project.org/web/packages/dbscan/vignettes/dbscan.pdf