Last Update: 2021-12-16
バイオインフォマティクス
- blast
- 2018
- 2016
- updates
- books
- enrichment
- gff
- gtf
- link
- refseq
- uniprot
- jsbi-nintei
- unclassified
- excel
- bird
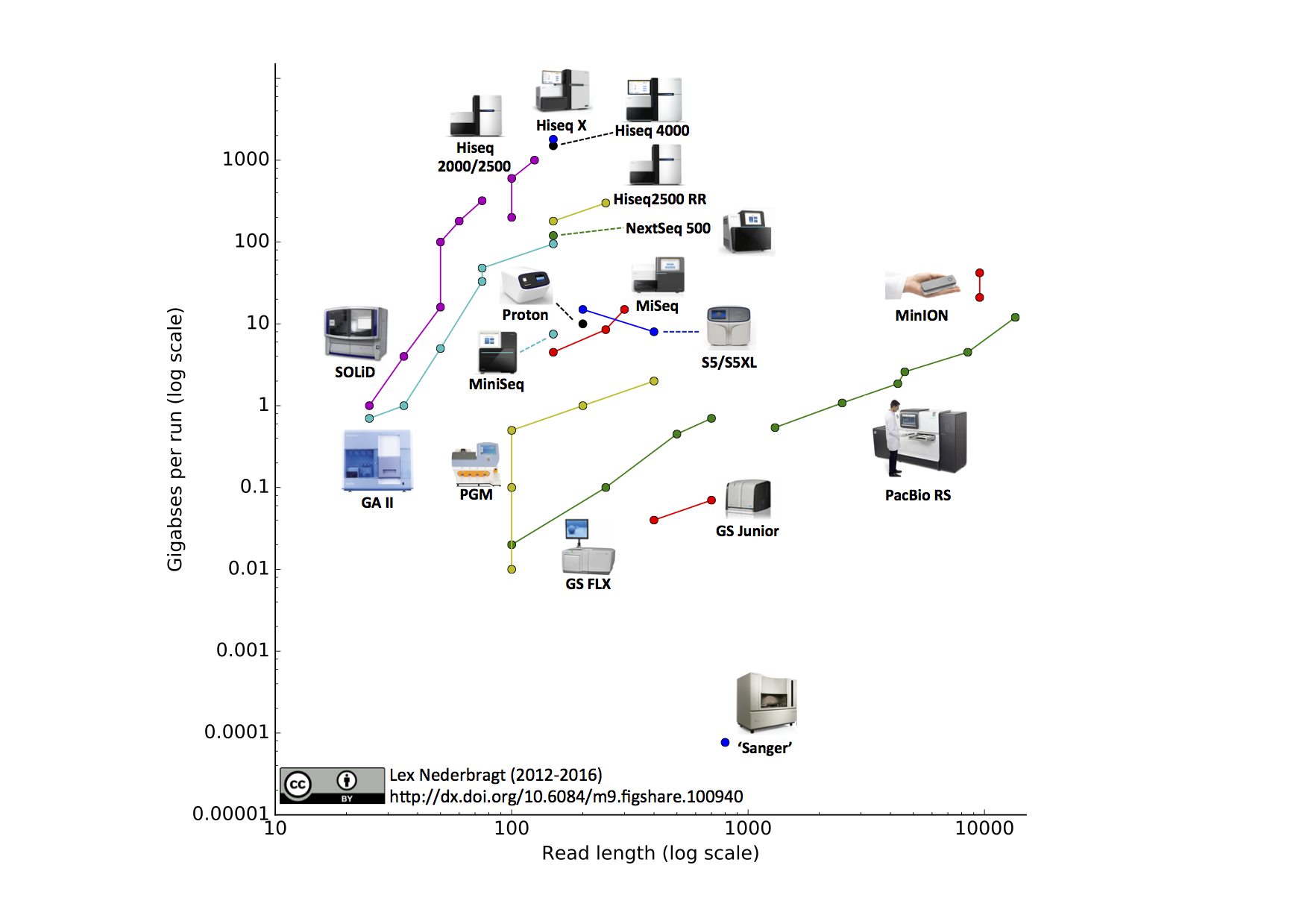
https://github.com/lexnederbragt/developments-in-next-generation-sequencing Developments in next generation sequencing
2:42 PM · Jul 9, 2020 https://twitter.com/dritoshi/status/1281101461112229889 ゲノムのほうの愛ちゃん🌙 🐧 on Twitter: "みなさま、ゲノム科学実験やゲノムインフォマティクス研究に必要な知識を得るための書籍やリンクをまとめています。おすすめを教えてください https://t.co/63UzSgpG8y" / Twitter
Sep 4, 2018 https://www.cs.rice.edu/~ogilvie/comp571/2018/09/04/pam-vs-blosum.html PAM vs BLOSUM score matrices | Species and Gene Evolution
https://bit.riken.jp/ja/howtostudybioinformatics/ How to study bioinformatics - Laboratory for Bioinformatics ResearchLaboratory for Bioinformatics Research
https://twitter.com/search?q=iNaturalist%20の
https://www.conference-service.com/conferences/bioinformatics.html Conferences and Meetings on Genomics and Bioinformatics
https://github.com/haruosuz/r4bioinfo/tree/master/R_Avril_Coghlan#pairwise-sequence-alignment
-
DDBJ 配列の記載に用いる略号
-
2017.06.03 配列解析 | 塩基配列解析とアミノ酸配列解析
-
24 July 2012 Aritalab:Lecture/Bioinformatics/Alignment - Metabolomics.JP
-
バイオインフォマティクス-ゲノム配列から機能解析へ-第3章3.4節配列アラインメントにおけるスコア行列とギャップペナルティの使用方法
http://www.dna.bio.keio.ac.jp/lecture/bioinfo/ 2021年度 バイオインフォマティクス 講義ノート 慶應義塾大学生命情報学科 榊原康文 https://www.dna.bio.keio.ac.jp/lecture/bioinfo/bioinformatics-3.pdf バイオインフォマティクス (第3回) 慶應義塾大学生命情報学科 榊原康文 配列解析(ペアワイズアライメント) ① ペアワイズアライメント ② 最長共通部分配列(LCS) ③ 大域アライメント,局所アライメント ④ スコア行列(置換行列)
https://www.jst.go.jp/nbdc/bird/jinzai/literacy/streaming/ ストリーミング配信 - BIRD
平成21年度ゲノムリテラシー講座
バイオインフォマティクスの基礎:分子生物学データベース(2009年9月26日開催) 講師:村上 勝彦 分子系統解析(講義3) 講義資料3 (PDF;1569 KB) https://www.jst.go.jp/nbdc/bird/jinzai/literacy/streaming/h21_4_3b.pdf
バイオインフォマティクスの基礎:配列解析、タンパク質立体構造解析(2009年9月12日開催) 講師:川端 猛 講義資料1 (PDF;2241 KB) https://www.jst.go.jp/nbdc/bird/jinzai/literacy/streaming/h21_3_1.pdf
2012.03.13 https://doi.org/10.7875/togotv.2012.033 【ゲノムリテラシー講座】分子系統解析(講義3) | 統合TV 本日の統合TVは、JSTバイオインフォマティクス推進センター(BIRD) ゲノムリテラシー講座において2009年9月26日に開催された、バイオインフォマティクスの基礎:分子生物学データベース から、村上 勝彦(社団法人バイオ産業情報化コンソーシアム)による「分子系統解析(講義3)」をお送りします。約51分です。 https://www.youtube.com/watch?v=K9Fy1LrtGco 【ゲノムリテラシー講座】分子系統解析(講義3) - YouTube
2012.03.10 https://doi.org/10.7875/togotv.2012.030 【ゲノムリテラシー講座】タンパク質立体構造解析(講義3) | TogoTV 本日の統合TVは、JSTバイオインフォマティクス推進センター(BIRD) ゲノムリテラシー講座において2009年9月12日に開催された、バイオインフォマティクス基礎講座:配列解析、タンパク質立体構造解析 から、川端 猛(奈良先端科学技術大学院大学)による「配列解析基礎(講義3)」をお送りします。約46分です。
2012.03.09 https://doi.org/10.7875/togotv.2012.029 【ゲノムリテラシー講座】配列解析基礎(講義2) | TogoTV 本日の統合TVは、JSTバイオインフォマティクス推進センター(BIRD) ゲノムリテラシー講座において2009年9月12日に開催された、バイオインフォマティクス基礎講座:配列解析、タンパク質立体構造解析 から、川端 猛(奈良先端科学技術大学院大学)による「配列解析基礎(講義2)」をお送りします。約67分です。
2012.03.08 https://doi.org/10.7875/togotv.2012.028 【ゲノムリテラシー講座】配列解析基礎(講義1) | TogoTV 本日の統合TVは、JSTバイオインフォマティクス推進センター(BIRD) ゲノムリテラシー講座において2009年9月12日に開催された、バイオインフォマティクス基礎講座:配列解析、タンパク質立体構造解析 から、川端 猛(奈良先端科学技術大学院大学)による「配列解析基礎(講義1)」をお送りします。約68分です。
2020年8月7日 13:30 https://japanese.engadget.com/amp/rename-genes-due-to-excel-043027343.html 「Excelが日付に変換してしまうから」一部遺伝子の名称を変更。ヒトゲノム解析の科学者ら
Aug 6, 2020, 8:44am EDT https://www.theverge.com/2020/8/6/21355674/human-genes-rename-microsoft-excel-misreading-dates Scientists rename human genes to stop Microsoft Excel from misreading them as dates - The Verge
12:08 AM · Aug 5, 2020 https://twitter.com/jannahutz/status/1290666010228514824 Janna Hutz on Twitter: "THRILLED by this announcement by the Human Gene Nomenclature Committee. https://t.co/BqLIOMm69d" / Twitter
03 August 2020 https://www.nature.com/articles/s41588-020-0669-3 Guidelines for human gene nomenclature | Nature Genetics
2016年08月29日 13時06分 https://gigazine.net/news/20160829-excel-gene-name-error/ Excelが遺伝子研究の誤りのもとになっていると研究者が指摘 - GIGAZINE
6:22 AM · Aug 26, 2016 https://twitter.com/DailyScan/status/768921595549143040 GenomeWeb Daily Scan on Twitter: "Didn't Quite Mean 1-Mar: Reliance on Excel leads to errors in gene name lists, @GenomeBiology paper reports https://t.co/CZ4Fx20N3s" / Twitter Aug 25, 2016 https://www.genomeweb.com/scan/didnt-quite-mean-1-mar#.Xy0nZBMzawQ Didn't Quite Mean 1-Mar | GenomeWeb
https://pubmed.ncbi.nlm.nih.gov/27552985/ Genome Biol . 2016 Aug 23;17(1):177. doi: 10.1186/s13059-016-1044-7. Gene name errors are widespread in the scientific literature Mark Ziemann 1, Yotam Eren 1 2, Assam El-Osta 3 4
http://bioinfo-dojo.net バイオインフォ 道場 [bioinfo-Dojo] – 日々、バイオインフォマティクス修行。
https://twitter.com/hashtag/museomejp https://sites.google.com/site/museumbioinfo/ Japan Museum Bioinformatics (Museomics) Working Group
https://twitter.com/ecSeq ecSeq Bioinformatics
https://www.sbj.or.jp/sbj/sbj_bioinformatics.html 生物工学会誌 –『バイオインフォマティクスを使い尽くす秘訣教えます!』 | 公益社団法人 日本生物工学会
http://data.dbcls.jp/~bono/lectures.html Dr. Bono's website Lectures 講義や講演
2:02 PM · Sep 4, 2020 https://twitter.com/DDBJ_topics/status/1301747553860333569 Bioinformation and DDBJ Center on Twitter: "日本バイオインフォマティクス学会年会・第9回生命医薬情報学連合大会(IIBMP2020)データサイエンティスト養成セッションの動画を YouTube にアップしました。 https://t.co/rBZPlnjf63" / Twitter https://www.ddbj.nig.ac.jp/training.html#2020
Jul 2, 2020 https://www.youtube.com/watch?v=h5FRVSNQYAI 【生命情報入門】配列解析(1): ドットマトリックス法 - YouTube
https://twitter.com/level3defless/status/1181781492931776512 Koichi Higashi on Twitter: "今日からPython講習会。実習でやる予定のColab資料。教師なし学習編。PCA, NMF, t-SNE, UMAP, (Louvain), Leiden, DBSCANなど。シングルセルRNA-seqデータを題材に。 https://t.co/jeFpAxlxQS" / Twitter 1:00 PM · Oct 9, 2019
https://www.ncbi.nlm.nih.gov/pubmed/30670080 Genome Biol. 2019 Jan 22;20(1):17. doi: 10.1186/s13059-019-1626-2. Where is genomics going next? Cheifet B1. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6341733/ Chris Mason, Weill Cornell Medicine
2018/07/21 http://www.kenkyuu2.net/cgi-biotech2012/biotechforum.cgi?mode=view;Code=7092 BioTechnicalフォーラム [プログラミングの勉強] バイオインフォマティシャンの需要が高まっていますが、 最初に習得すべき言語は何がいいでしょうか? 現在どの言語が多用されているのでしょうか。
January 17, 2018 https://github.com/yuifu/ajacs68/blob/master/README.md NGSデータから新たな知識を導出するためのデータ解析リテラシー 尾崎 遼 Haruka Ozaki
2017年12月6日 https://togetter.com/li/1178658 「いかにして『使える』データベースを維持し続けるか?」 - Togetter
http://www.iu.a.u-tokyo.ac.jp/lectures/AG14/ 14.農学生命情報科学特論IV | アグリバイオインフォマティクス教育研究ユニット 平成29年10月5日(木) 参考:講義ウェブサイト https://github.com/haruosuz/introBI/blob/master/2017/CaseStudy.md#2017-10-05
https://biosciencedbc.jp/gadget/human/170901_higashi_170831.pdf Hi-C解析(2017NGSハンズオン講習会-2017年9月1日) 2017年9月1日 国立遺伝学研究所 東光一
https://twitter.com/bonohu/status/1104623240775852032 bonohu on Twitter: "“How to study bioinformatics / シーケンスデータ解析などバイオインフォマティクスを学びたい方” / “How to study bioinformatics - Laboratory for Bioinformatics ResearchLaboratory for Bioinformatics Research” https://t.co/naIWZd7ajG" 1:01 AM - 10 Mar 2019 https://bit.riken.jp/ja/howtostudybioinformatics/ How to study bioinformatics - Laboratory for Bioinformatics ResearchLaboratory for Bioinformatics Research
https://twitter.com/vsbuffalo/status/1098269961145901056 Vince Buffalo on Twitter: "I'm teaching my partner bash. The biggest "wows!" have been: 1. control-r, type part of a past command, control-r again and again to cycle through history. 2. cd - 3. tmux for work on a server. 4. control-a / control-e / option arrow for jumping around. 5. hitting tab a lot." 12:15 PM - 20 Feb 2019
https://twitter.com/3Bgoripachi/status/1025225950902931456 ゴリぱち on Twitter: "ゲノム解析を学びたい人にとって有益な情報が詰まっている記事でした。 「NGSデータから新たな知識を導出するためのデータ解析リテラシー」 https://t.co/t5GGcj7mNC" 11:44 PM - 2 Aug 2018
https://twitter.com/eyemiyake/status/1023007444669095936 三宅正裕 MD, PhD, MPH @ 眼科 on Twitter: "eLIFE, 2018 ゲノムデータを操作変数として用いることで観察研究データから因果関係を評価するMendelian Randomization。そろそろやってみたいのでとりあえずこの論文で勉強しています 臨床、ゲノム、疫学のいずれもに知見とデータがあるうちの強みにマッチしそう。 https://t.co/CMSPLf3Dfk" 8:49 PM - 27 Jul 2018
https://wired.jp/2018/03/13/solve-genomics-with-blockchain/ 「ゲノム解析」のデータはブロックチェーンで守れるか|WIRED.jp
2015年1月号 https://www.natureasia.com/ja-jp/ndigest/v12/n1/誰もが%26ldquo%3Bバイオインフォマティシャン%26rdquo%3Bの時代/59368 誰もが“バイオインフォマティシャン”の時代 中村 保一、坊農 秀雅、粕川 雄也、仲里 猛留 Nature ダイジェスト Vol. 12 No. 1 | doi : 10.1038/ndigest.2015.150122
http://catway.jp/bioinformatics/index.html Tips for NGS Data Analysis 次世代DNAシーケンサーのデータ解析技術 (2013/02/01)
http://apprize.info/data/bioinformatics/index.html Bioinformatics Data Skills (2015)
https://github.com/haruosuz/books/blob/master/bds/README.md
http://www.kspub.co.jp/book/detail/1538559.html タンパク質機能解析のためのバイオインフォマティクス
http://www.bic.kyoto-u.ac.jp/toh/text.html 第2章 タンパク質の分子進化 2.4 分子進化の性質(3) - 保存のサイト特異性 2.4.3 アミノ酸残基の保存と変化 https://www.ncbi.nlm.nih.gov/pubmed/9010921 Protein Eng. 1996 Dec;9(12):1067-82. Glutathione-independent prostaglandin D synthase as a lead molecule for designing new functional proteins. Toh H1, Kubodera H, Nakajima N, Sekiya T, Eguchi N, Tanaka T, Urade Y, Hayaishi O.
第3章 タンパク質の 生化学的機能の解析 3.1 相同アミノ酸配列比較解析の手順 3.1.2 アラインメントからの情 報抽出 B 分子系統樹を利用した機能解析 https://www.ncbi.nlm.nih.gov/pubmed/10871367 Nucleic Acids Res. 2000 Jun 15;28(12):2353-62. Bacterial cryptochrome and photolyase: characterization of two photolyase-like genes of Synechocystis sp. PCC6803. Hitomi K1, Okamoto K, Daiyasu H, Miyashita H, Iwai S, Toh H, Ishiura M, Todo T.
https://github.com/haruosuz/r4bioinfo/blob/master/references/R.bio.md#enrichment
Nov 21, 2018 https://www.subioplatform.com/ja/info_technical/152/enrichment-analysis-tool Enrichment Analysis Tool の使い方 | Subio
https://home.hiroshima-u.ac.jp/naka/wiki/wiki.cgi?GSEA_%28Gene_Set_Enrichment_Analysis%29%A4%CB%A4%C4%A4%A4%A4%C6 GSEA_(Gene_Set_Enrichment_Analysis)について - http://www.riken.jp/pr/press/2016/20160510_1/ wPGSA法(weighted Parametric Gene Set Analysis)
https://www.ncbi.nlm.nih.gov/pubmed/27141961 Nucleic Acids Res. 2016 Jul 8;44(W1):W90-7. doi: 10.1093/nar/gkw377. Epub 2016 May 3. Enrichr: a comprehensive gene set enrichment analysis web server 2016 update. http://amp.pharm.mssm.edu/Enrichr
2012年12月20日 http://array.cell-innovator.com/?p=1452 Gene Set Enrichment Analysis (GSEA) – 遺伝子発現解析(マイクロアレイ解析, RNA-seq)
PAGEは正規分布(normal distribution)を仮定したパラメトリックなテストで、ノンパラメトリックテストのGSEAと比較して、計算が速く、検出感度が高い(統計的に有意な遺伝子が多い)。遺伝子セットの構成遺伝子数は最低10個
https://www.ncbi.nlm.nih.gov/pubmed/15941488 BMC Bioinformatics. 2005 Jun 8;6:144. PAGE: parametric analysis of gene set enrichment. Kim SY1, Volsky DJ. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1183189/ PAGE: Parametric Analysis of Gene Set Enrichment PAGE was statistically more sensitive and required much less computational effort than GSEA, , we set the minimal gene set size as 10.
2009/11/19 http://www.iu.a.u-tokyo.ac.jp/~kadota/r.html#page_go 解析 | 機能解析 | PAGE法(Kim_2005;統計量の変換なし)を用いてGene Ontology解析
https://www.t-kagaku.co.jp/seimeiken/top/xmlSupport.php?id=21004 PAGE解析(Parametric Analysis of Gene set Enrichment)とは
http://genomejack.net/download/gj31/ja/GenomeJackBrowserAppendix/browser_appendix_j/dataFileFormats/gff.html GFF (General Feature Format) はGFF standard file formatに準拠している行指向のフォーマットです。GFFの各行はそれぞれ9つのフィールドから構成されており、タブ区切りでなければなりません。スペースで区切られている場合、正しく表示されません。
2020年06月19日 https://qiita.com/Chronona/items/e04a0c39b2bdda92b220 genbankファイルをgffファイルに変換する - Qiita
http://kazumaxneo.hatenablog.com/entry/2019/08/26/073000 GFF ファイルのユーティリティ Gffread - macでインフォマティクス
https://mecobalamin.hatenablog.com/entry/2019/04/19/130901 フォーマット変換、GTF -> BED -> BAM - mecobalamin’s diary
2018年11月19日 https://oliversi.com/2018/11/05/file_format/ GTF/GFFファイル: 遺伝子のアノテーション等を記述するためのファイル形式 ⇒GFF3(GMOD)
2017年08月05日 https://qiita.com/MaedaTaro_Umiushi/items/48c628ca332abade1e98 Convert GFF3 to MSS for DDBJ submission - Qiita
2017年06月26日 https://qiita.com/MaedaTaro_Umiushi/items/4b9d37d23614e53a3cdf gff3 ↔gtf の変換 - Qiita
2017-07-25 http://kazumaxneo.hatenablog.com/entry/2017/07/25/234808 BEDOPSを使いVCF, GTF, GFF などを BED に変換 する。 - macでインフォマティクス GFF(GFF3)をbedに変換する。
2015-07-31 https://staffblog.amelieff.jp/entry/2015/07/31/143358 フォーマットもいろいろ - アメリエフのブログ
GFF(General Feature Format)はゲノムの配列に関連した特徴を示した9列からなるタブ区切りのフォーマットです。
それぞれの列の説明です。
1. seqname:染色体またはスキャフォールドの名前
2. source:データベースやプロジェクト名
3. feature:配列のタイプ(例:repeat, exon, promoter, etc)
4. start:配列の開始位置
5. end:配列の終了位置
6. score:任意のスコアが入る。ない場合は '.'
7. strand:+(forward)、-(reverse)または '.'
8. frame:0から2の数値で、翻訳を開始する塩基の位置を示す(0なら配列の1番目がコドンの1番目の塩基になる)。コーディング領域でない場合は '.'
9. attribute:グループ
Apr 23 2014 門田幸二(かどた こうじ) http://www.iu.a.u-tokyo.ac.jp/~kadota/20140423_kadota.pdf GFF3形式ファイルの例(シロイヌナズナ; TAIR10_GFF3_genes.gff) 遺伝子ごとに、どの染色体 のどの座標上に存在するの かなどの情報を含むタブ区 切りテキストファイル
http://genomejack.net/download/gj31/ja/GenomeJackBrowserAppendix/browser_appendix_j/dataFileFormats/gtf.html GTFはGFFと良く似ている形式です。GTFはタブ区切りのテキストファイルで、最初の8つのフィールドはGFFと同じです。GTFの9番目のgroupフィールドはGFFと異なり、そこには各行のデータの属性(gene_idやtranscript_id、exon number等)のリストが記述されます。ここで記述される属性はそれぞれtype(ex. gene_id) とvalue(ex. “AB000123.1”)がを持っていおり、各属性の後ろには;が付き、スペースで区切られます。
2017.06.11 https://bi.biopapyrus.jp/format/gtf.html GTFファイル | 遺伝子アノテーションファイルの処理
2017-06-10 http://kazumaxneo.hatenablog.com/entry/2017/06/10/160701 GTFとGFFフォーマット - macでインフォマティクス
http://staffblog.amelieff.jp/entry/2015/07/31/143358 フォーマットもいろいろ - アメリエフのブログ
https://cell-innovation.nig.ac.jp/SurfWiki/Tips.html
2013/02/05 https://cell-innovation.nig.ac.jp/SurfWiki/GTF_difference.html GTFファイルの細かな違い
https://twitter.com/NCBI/status/1228429011119988736 NCBI Staff on Twitter: "Important changes coming to prokaryotic Reference and Representative genome assemblies in Feb! https://t.co/vMPnTse9pv https://t.co/Wxxq92A2Uh" / Twitter February 14, 2020 https://ncbiinsights.ncbi.nlm.nih.gov/2020/02/14/assembly-changes/ NCBI Insights : Important changes coming to prokaryotic Reference and Representative genome assemblies
https://twitter.com/Tyu_Shi/status/1211638901405667331 Tyu_Shi on Twitter: "今やっている比較ゲノム解析、数ヶ月前に取得したRefSeqのゲノムデータ中にはその株の完全ゲノムがあったのに、最新のRefSeqでは無くなっていて不思議に思って調べてみたら、Anomalous assemblyとフラグが付いてRefSeqからエントリが除外されていた。 https://t.co/XGNgCf6fNi" / Twitter 10:23 PM · Dec 30, 2019 https://ncbi.nlm.nih.gov/assembly/help/anomnotrefseq/ Assembly Anomalies and Other Reasons a Genome Assembly may be Excluded from RefSeq
https://twitter.com/torstenseemann/status/1170944364765270016 Torsten Seemann on Twitter: ""25 reasons Genbank assemblies don't make it into Refseq" - my latest blog post #bioinformatics https://t.co/yjkwgpARXj" 3:17 PM · Sep 9, 2019· https://thegenomefactory.blogspot.com/2019/09/25-reasons-assemblies-dont-make-it-into.html
https://bi.biopapyrus.jp/db/refseq.html RefSeq | 詳細な注釈づけられている冗長性のない核酸データベース
NC_ DNA 1 個体のデータから集められた完全なゲノム配列 NZ_ DNA 不完全なショットガン配列
http://d.hatena.ne.jp/yuifu/20110426/1303820667 Entrez Gene と RefSeq の関係 - micro*macro
NC_ Genomic Complete genomic molecule, reference assembly NZ_b Genomic Unfinished WGS
https://www.biostars.org/p/209159/ What is the difference between NZ_ and NC_ identifiers at NCBI?
NC - "Complete genomic molecule, usually reference assembly" NZ - "Unfinished WGS"
https://www.ncbi.nlm.nih.gov/books/NBK21091/table/ch18.T.refseq_accession_numbers_and_mole/ NC_ Genomic Complete genomic molecule, usually reference assembly NZ_b Genomic Complete genomes and unfinished WGS data
https://www.ebi.ac.uk/training-beta/online/courses/uniprot-exploring-protein-sequence-and-functional-info/what-is-uniprot/ What is UniProt? | UniProt Watch this short introduction to UniProt to learn how to get started: May 7, 2014 https://www.youtube.com/watch?v=9IYI4QDVPa0 UniProt Intro - YouTube
https://www.ebi.ac.uk/training-beta/online/courses/uniprot-exploring-protein-sequence-and-functional-info/exploring-a-uniprotkb-entry/ Exploring a UniProtKB entry | UniProt Mar 15, 2016 https://www.youtube.com/watch?v=x9GNm2DLP-U UniProtKB Entry View intro (inc Feature Viewer) - YouTube
https://www.ebi.ac.uk/training-beta/online/courses/uniprot-exploring-protein-sequence-and-functional-info/how-to-use-uniprot-tools/ How to use UniProt tools | UniProt
https://www.ebi.ac.uk/training-beta/online/courses/uniprot-exploring-protein-sequence-and-functional-info/how-to-use-uniprot-tools/blast-sequence-similarity-searching/ BLAST sequence similarity searching | UniProt May 11, 2014 https://www.youtube.com/watch?v=UPaConHNP7E Blast intro - YouTube
https://www.ebi.ac.uk/training-beta/online/courses/uniprot-exploring-protein-sequence-and-functional-info/how-to-use-uniprot-tools/aligning-multiple-protein-sequences/ Aligning multiple protein sequences | UniProt May 11, 2014 https://www.youtube.com/watch?v=IAYFLfPQ0Gs Align intro - YouTube
https://www.uniprot.org/blast/ Target database UniRef90
http://www.uniprot.org/help/uniref UniRef90 is built by clustering UniRef100 sequences with 11 or more residues using the MMseqs2 algorithm (Steinegger M. and Soeding J., Nat. Commun. 9 (2018)) such that each cluster is composed of sequences that have at least 90% sequence identity to and 80% overlap with the longest sequence (a.k.a. seed sequence) of the cluster. Similarly, UniRef50 is built by clustering UniRef90 seed sequences that have at least 50% sequence identity to and 80% overlap with the longest sequence in the cluster. Prior to 2013 there was no overlap threshold, so clusters were more heterogeneous in length. UniRef90 and UniRef50 yield a database size reduction of approximately 58% and 79%, respectively, providing for significantly faster sequence similarity searches.
http://www.uniprot.org/help/fasta-headers UniRef
>UniqueIdentifier ClusterName n=Members Tax=TaxonName TaxID=TaxonIdentifier RepID=RepresentativeMember
- TaxonName is the scientific name of the lowest common taxon shared by all UniRef cluster members.
https://ja.wikipedia.org/wiki/Swiss-Prot UniProtの冗長性のないリファレンス (UniRef) のデータベース群[8] UniRef は、高速な類似性検索に供するため類縁性の強い配列を単一の配列データにまとめたデータベース群である。
http://kazumaxneo.hatenablog.com/entry/2019/08/26/205653 UniProt のUniRef データベース - macでインフォマティクス
https://www.jsbi.org/activity/nintei/ バイオインフォマティクス技術者認定試験
https://www.jsbi.org/activity/nintei/books/ 参考図書 | Japanese Society for Bioinformatics - JSBi
https://www.jsbi.org/activity/nintei/sankou_mondai_kako/ 参考問題・過去問 | Japanese Society for Bioinformatics - JSBi
過去の問題・解説
2019年度(平成31年度) 問題 解説 https://www.jsbi.org/media/files/activity/nintei/sankou_mondai_kako/2019_mondai.pdf https://www.jsbi.org/media/files/activity/nintei/sankou_mondai_kako/2019_kaisetsu_r.pdf
- https://twitter.com/DrKatHolt Kat Holt (@DrKatHolt) | Twitter
- https://twitter.com/phylogenomics Jonathan Eisen (@phylogenomics) | Twitter
- https://twitter.com/pathogenomenick Nick Loman (@pathogenomenick) | Twitter
- https://twitter.com/strnr Stephen Turner (@strnr) | Twitter
- https://twitter.com/jcamthrash Cameron Thrash (@jcamthrash) | Twitter
- https://twitter.com/mason_lab Christopher Mason (@mason_lab) | Twitter
- https://twitter.com/WvSchaik Willem van Schaik (@WvSchaik) | Twitter
- https://twitter.com/torstenseemann Torsten Seemann (@torstenseemann) | Twitter
- https://twitter.com/KojiYahara Koji Yahara @ National Institute of Infectious Diseases.
微生物の研究
- https://twitter.com/Tyu_Shi Tyu_Shi (@Tyu_Shi) | Bioinformaticsの研究者をしてます。
- https://twitter.com/level3defless Koichi Higashi (@level3defless) | NIG, Microbiology
- https://twitter.com/hornistyf Y. Furuta (@hornistyf) | バクテリアのゲノム比較解析とか、DNAメチル化とか。
- https://twitter.com/yokadzaki Yusuke Okazaki (@yokadzaki) | 湖に生息する微生物の研究をしてます。
- https://twitter.com/kmoooooog くまがい (@kmoooooog) | 博士(環境学) JAMSTECで海洋微生物学の研究
- https://twitter.com/ysknishimura Yosuke Nishimura (@ysknishimura) | (メタ)ゲノム情報を用いて海洋微生物の研究をしています。
- https://twitter.com/npoJSBi JSBi (@npoJSBi) | 日本バイオインフォマティクス学会(JSBi)
- https://twitter.com/bioinfowakate 生命情報科学若手の会 (@bioinfowakate) | Twitter
- https://twitter.com/chemo_wakate ケモインフォマティクス若手の会公式 (@chemo_wakate) | Twitter
- https://twitter.com/dbcls ライフサイエンス統合データベースセンター (@dbcls) | Twitter
- https://twitter.com/biohackathon
- https://twitter.com/togotv
- https://twitter.com/togotv_bot
- https://twitter.com/bonohu https://dbcls.rois.ac.jp/~bono/ 坊農秀雅のウェブサイト http://bonohu.jp/blog/ ぼうのブログ
- https://twitter.com/chalkless Nakazato T. (@chalkless) | Twitter
- https://twitter.com/yayamamo
- https://twitter.com/h_ono
- https://twitter.com/inutano
- https://twitter.com/meso_cacase
- https://twitter.com/tktym toshiaki katayama (@tktym) | Twitter
- https://twitter.com/skwsm
- https://twitter.com/tkwsm tk (風来風来亭) (@tkwsm) | カエル、ホヤ、ナメクジウオ、サンゴ、二枚貝、ギボシムシの研究を通じて、今はヌルヌルに興味があります。
- https://twitter.com/masanaoAtTheLab Masanao Sato/佐藤昌直 (@masanaoAtTheLab) | Systems biology/Virology/Genomics
- https://twitter.com/dritoshi
- https://twitter.com/fukunagaTsu 福永 津嵩 (@fukunagaTsu) | Twitter
- https://twitter.com/m_hama Michiaki Hamada 浜田道昭 (@m_hama) | Twitter
- https://twitter.com/yaskaz
- https://twitter.com/sounaka
- https://twitter.com/kazumachack macでインフォマティクス
- https://twitter.com/sesejun
- https://twitter.com/ogishima
- https://twitter.com/dancing_infobio
- https://twitter.com/maskot1977
- https://twitter.com/mkasahara
- https://twitter.com/gaou_ak
- https://twitter.com/nzmyachie
- https://twitter.com/iwasakiw
- https://twitter.com/yuifu
- https://twitter.com/antiplastics もじゃもじゃのほう (Koki Tsuyuzaki) (@antiplastics) | Twitter
- https://twitter.com/kztakemoto
- https://twitter.com/windowmoon
- https://twitter.com/YoshiKawahara Yoshihiro Kawahara (@YoshiKawahara) | つくばの農学系バイオインフォマティシャン。
- https://twitter.com/drk0311 内藤健 (@drk0311) | Twitter
- https://twitter.com/kfuku0502 Kenji Fukushima (@kfuku0502) | carnivorous plants + convergent evolution
- https://twitter.com/mishimahryk 三嶋博之 (@mishimahryk) | 長崎大学人類遺伝学 属性: 人類遺伝学/NGS/次世代シークエンサー/Ruby/BioRuby/R/バイオインフォマティクス/歯科医師
- https://twitter.com/ma_ko
- https://twitter.com/yokonaga405
- https://twitter.com/marimiya_clc
- https://twitter.com/sum_ichi
- https://twitter.com/Yh_Taguchi 田口善弘 (@Yh_Taguchi) | **大学理工学部物理学科教授。専門はバイオインフォマティクス。
- https://twitter.com/leeswijzer MINAKA Nobuhiro (@leeswijzer) | オモテの仕事は生物統計学の研究者,ウラの仕事は進化生物学者です.
https://twitter.com/WMicrobiomeDay World Microbiome Day (@WMicrobiomeDay) | Twitter June 27th is a day to celebrate all things Microbiome around the world. The 1st World Microbiome Day was launched by APC Microbiome Ireland in 2018.
- https://twitter.com/Med_Microbe_bot 感染微生物bot (@Med_Microbe_bot)
- https://twitter.com/biseibutsu__bot 微生物bot (@biseibutsu__bot)
- https://twitter.com/kamosumon_bot 醸して学ぶbot (@kamosumon_bot)
- https://twitter.com/cuteimouto_bot 微生物学専攻妹bot (@cuteimouto_bot)
- https://twitter.com/bactery_bot ばくてり~細菌擬人化bot~ (@bactery_bot)
- https://twitter.com/AAD_Japan Save ABx Japan (@AAD_Japan) | 抗菌薬、薬剤耐性菌