| typora-copy-images-to |
|---|
./images/ |
[TOC]
胡志峰 16307130177
使用De Brujin Graph来组装。只利用短串,首先通过反转、取补的操作扩充成更大的短串集合。然后对于每个短串将其打碎成kmer,基于这些kmer来构建De Bruijin Graph。
构建好DBG之后,我分析了该图的形态。首先知道对于DBG中的节点来说,其入度和出度至多为4。考虑以下的度数情况
- 入度或者出度为0,该节点为基因的起点或者终点
- 入度和出度都为1,该节点为基因路径上的一段
- 入度或者出度大于等于2,该节点为某个分支的开始或者结束
对于情况1和2都很好理解,而情况3则较为复杂。分析产生分支的原因有一下几点
由于某个短串在该位置测序错误,导致路径分裂了出去。我们需要把这部分分支路径合并回主要路径。我考虑了用以下条件来判断这个情况
-
两个分支中的一个经过次数明显小于另一个。因为错误发生导致的分支经过次数会明显小于正常的分支
if ((double)x->cvg / y->cvg < 0.7) { -
从两个分支一直往下走直到第一个入度大于1的点相同,并且分支的长度足够长
walkThroughBubble(x->node, xpath); walkThroughBubble(y->node, ypath);
当上述两个条件满足时删除经过次数低的那个分支即可。
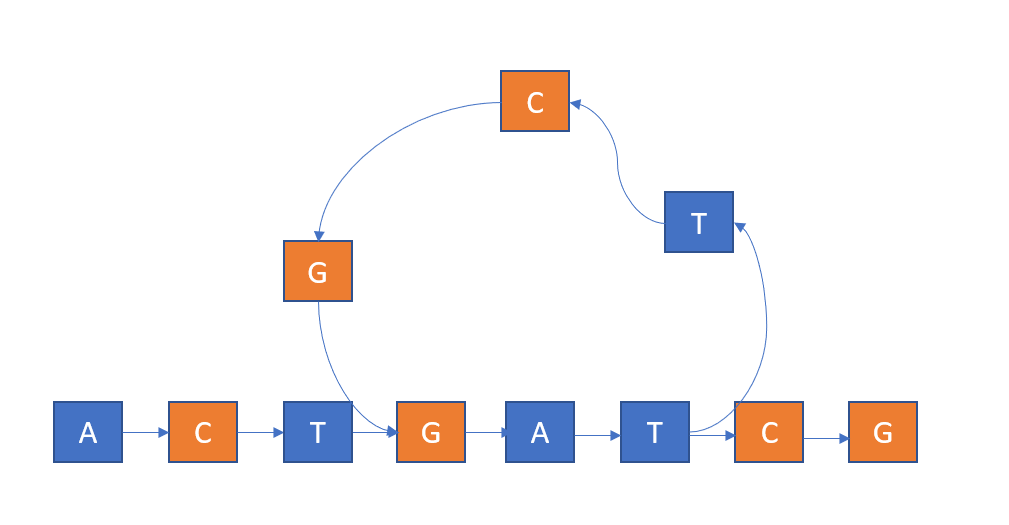
众所周知,基因中有大量的重复片段1。这导致不可避免地在DBG图上出现环
如上图所示,如果我们不考虑环,则基因路径为ACTGATCG,但是考虑到环的存在,我们会读出ACTGATTCGGATCG。因此我们需要找出DBG中的环。我修改了Tarjar的强连通分量算法,使用深度优先搜索标注出了图中指向的环的分支,在输出的时候优先走带环的分支。
在考虑完上述的情况后,DBG中便几乎不存在出度或者入度大于1的节点,因此可以开始输出。根据上述优先走环的方法,我们额外考虑逐步输出最长串的方法——迭代地通过深度优先搜索找到最长路径,输出后删除。为了降低覆盖率考虑只输出长度足够长的串。
我使用git来管理代码,因此可以通过检查git log来证明PJ的完成过程。代码将在PJ截止后公开,可以通过dna-assembly-cpp访问。
使用面向对象的方法完成程序。主要实现了Genome对象用来处理基本的字符串输入输出;DBGNode对象来表示De Brujin图中的节点,DBGEdge来表示图中的边,DeBrujinGraph来表示图。
因为使用c++完成项目,我编写了Makefile便于使用make命令来进行自动化的构建。在根目录使用make 1即可编译并运行处理第一个数据点,类似可以处理其余数据点。
结果复现。在获得结果后我及时commit了代码、可执行程序和结果。checkout到以下几个commit便可得到结果
| data | commit | result(NGA50) |
|---|---|---|
| 1 | 557dc6a53333694eabf5408b4732d52cbe74cef3 |
9983.0 |
| 2 | 750215dc949c9be5cbcfda988533e4179a914f7a |
9408.2 |
| 3 | 750215dc949c9be5cbcfda988533e4179a914f7a |
7859.2 |
| 4 | 64d47516a209c23e4b65d085d6a6c24c38795779 |
22275.4 |
举例来说在commit64d4751中执行make 4即可自动编译并运行获得第四个数据点的输出。不过要注意第四个点递归可能出现栈溢出,需要ulimit -s unlimited扩大栈空间。
mailto: wanjunpeng@foxmail.com