上周我们发布的四个R包基本上能进解决表达芯片数据挖掘的88%的问题,如下:
有趣的是,因为这些包存储在GitHub,而且每个包自带的数据是40~50M,对很多在**大陆的朋友来说, 几乎是不可能完成,所以我把这4个包整合成为了一个GitHub包(AnnoProbe)!总共不到5M,相信大家使用起来应该是很方便啦!
library(devtools)
install_github("jmzeng1314/AnnoProbe")
library(AnnoProbe)因为这个包里面并没有加入很多数据,所以理论上会比较容易安装,当然,不排除**大陆少部分地方基本上连GitHub都无法访问。
比如我在 (重磅!价值一千元的R代码送给你)芯片探针序列的基因组注释 提到的例子;关于
Human LncRNA Expression Array V4.0 AS-LNC-H-V4.0 20,730 mRNAs and 40,173 LncRNAs 8*60K
这个芯片探针的重新注释,一般文献里面的描述是:
- probe sequences 探针序列下载
- uniquely mapped to the human genome (hg19) by Bowtie without mismatch. 参考基因组下载及比对
- chromosomal position of lncRNA genes based on annotations from GENCODE (Release 23)坐标提取,最后使用bedtools进行坐标映射
但是大部分人是没有linux操作能力,无法完成这个流程,使用我们的包可以轻轻松松达到探针注释的目的!
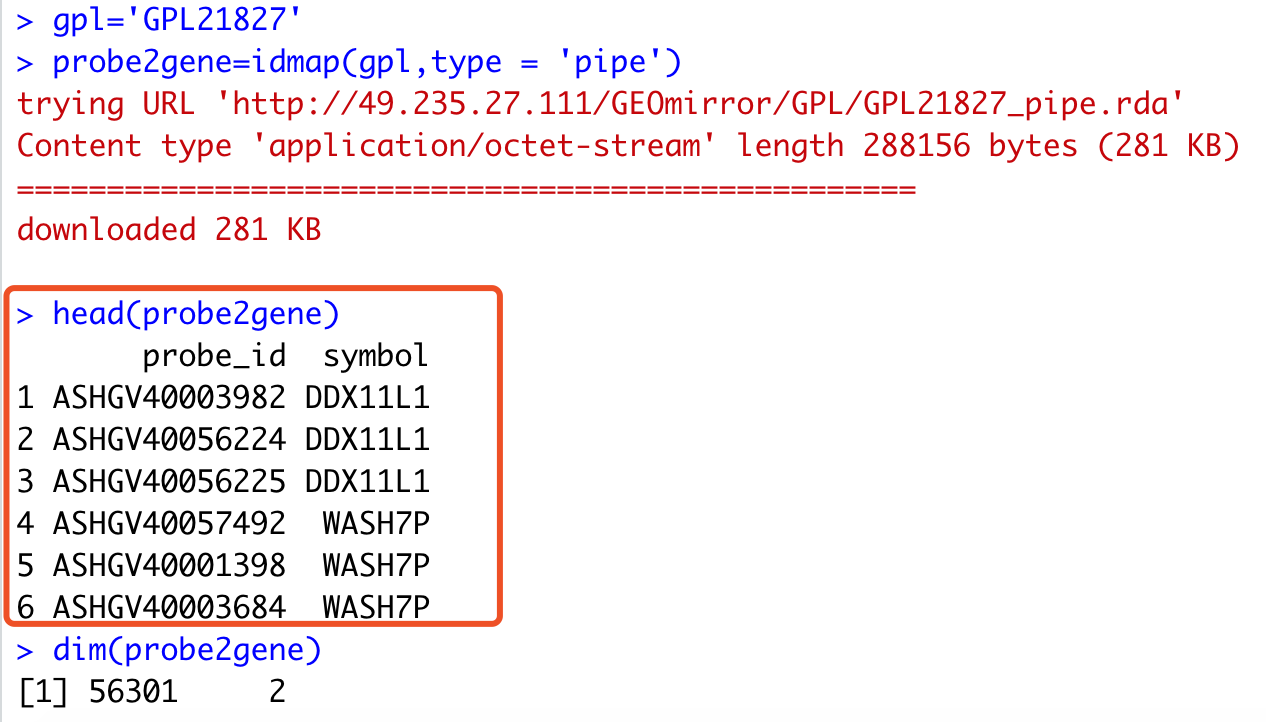
# GPL21827[Accession] - GEO DataSets Result - NCBI - NIH
# https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL21827
gpl='GPL21827'
probe2gene=idmap(gpl,type = 'pipe')
head(probe2gene)轻轻松松的几行代码,就拿到了探针的注释信息哦,就是我帮你跑完了芯片探针的重新注释,而且把注释好的结果返回给你。
是不是很激动。
rm(list = ls())
library(AnnoProbe)
suppressPackageStartupMessages(library(GEOquery))
gset=AnnoProbe::geoChina('GSE1009')
gset需要理解一下下载的ExpressionSet对象,主要是表达矩阵和临床信息啦!
# check the ExpressionSet
eSet=gset[[1]]
# extract the expression matrix and phenotype data
probes_expr <- exprs(eSet);dim(probes_expr)
head(probes_expr[,1:4])
boxplot(probes_expr,las=2)
probes_expr=log2(probes_expr+1)
boxplot(probes_expr,las=2)
## pheno info
phenoDat <- pData(eSet)
head(phenoDat[,1:4])这一个步骤也是在线下载我们的芯片注释信息
## check GPL and annotate the probes to genes.
(gpl=eSet@annotation)
checkGPL(gpl)
printGPLInfo(gpl)
probe2gene=idmap(gpl)
head(probe2gene)
genes_expr <- filterEM(probes_expr,probe2gene )
head(genes_expr)# do DEG
## define the group
group_list=factor(c(rep('Control',3),rep('Diabetes',3)))
table(group_list)
library(limma)
design=model.matrix(~factor(group_list))
design
fit=lmFit(genes_expr,design)
fit=eBayes(fit)
DEG=topTable(fit,coef=2,n=Inf)
head(DEG)## visualization
need_deg=data.frame(symbols=rownames(DEG), logFC=DEG$logFC, p=DEG$P.Value)
deg_volcano(need_deg,1)
deg_volcano(need_deg,2)
deg_heatmap(DEG,genes_expr,group_list)
deg_heatmap(DEG,genes_expr,group_list,30)
check_diff_genes('PLCE1',genes_expr,group_list)
check_diff_genes('MPP6',genes_expr,group_list)# 假设我这里对hsa03410感兴趣
library(KEGGREST)
cg <- KEGGREST::keggGet("hsa03410")[[1]]$GENE
cg=as.character(sapply(cg[seq(2,length(cg),by=2)], function(x) strsplit(x,';')[[1]][1]))
check_diff_genes( cg ,genes_expr,group_list)当然了,你需要有一点R语言基础知识啦,不然,上面的代码你不知道应该修改哪里。
马上试试看下面的数据集吧,我觉得蛮有意义的。
GSE1462
GSE18732
GSE20950
GSE21785
GSE26526
GSE32575
GSE43837
GSE474
GSE58979
GSE60291
GSE62832
GSE70529
GSE72158