郑重申明:此包idmap2存放在GitHub,仅仅是一个草稿,不会再维护,也不会更新,是为了说明我最后的annoprobe包的开发历程。
前面我们提到过表达芯片探针注释的3种方法,参见:第一个万能芯片探针ID注释平台R包, 并且帮助大家搞定了第一种bioconductor包的方法,大家无需下载几十个bioconductor包,然后自己一个个提取基因信息,我全部为大家做好啦,也就是 idmap1 这个目前host在GitHub的R包:
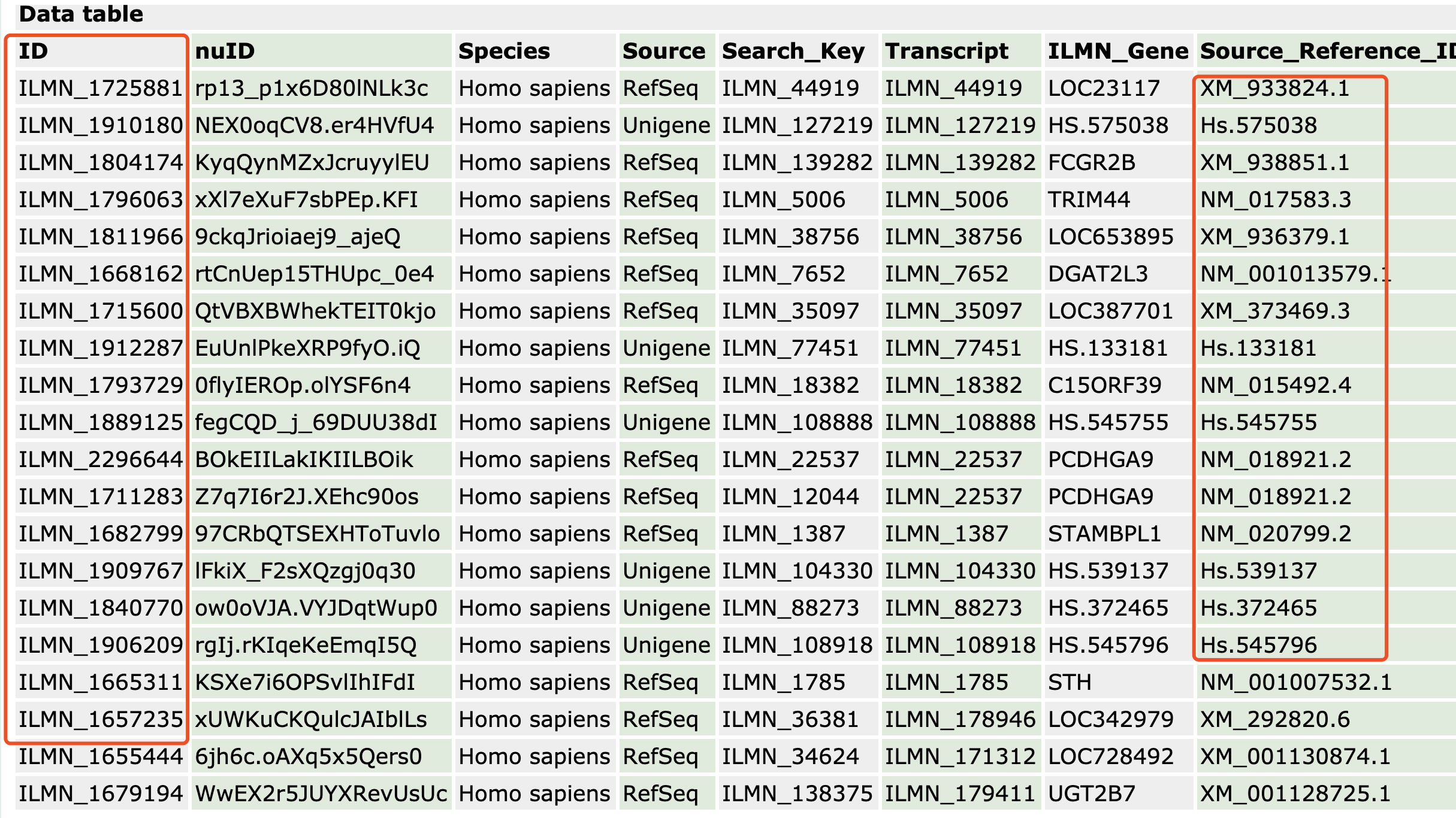
现在我们来说说第二个万能芯片探针ID注释平台包。一定要跟我们的 idmap1 区分开来哦,那个idmap1仅仅是把bioconductor里面有的37个芯片平台整合了一下,而我们的这个idmap2包不得了啦,有122个GPL之多,它们的6G多的soft信息被我下载整理成为了不到40M的R包。(PS : 因为有40多M,所以**大陆下载这个GitHub包会有点困难哦)
NCBI官网可以下载所有的gpl平台的soft信息,会比较大(100多个平台全部下载需要6G以上的空间)
不同的gpl平台的soft信息不一样,一般来说,大家关心的其实就是探针的ID,以及基因的symbol列。
有一些平台还会提供探针序列:
我已经帮助大家下载整理好了全部的GPL平台的芯片探针列,以及其对应的基因列,整理打包成为一个R包方便大家使用,直接获取探针对应的基因symbol即可。
一定要跟我们的 idmap1 区分开来哦,那个idmap1是把bioconductor里面有的37个芯片平台整合了一下,而我们的这个idmap2包不得了啦,有122个GPL之多,它们的6G多的soft信息被我下载整理成为了不到40M的R包。
library(devtools)
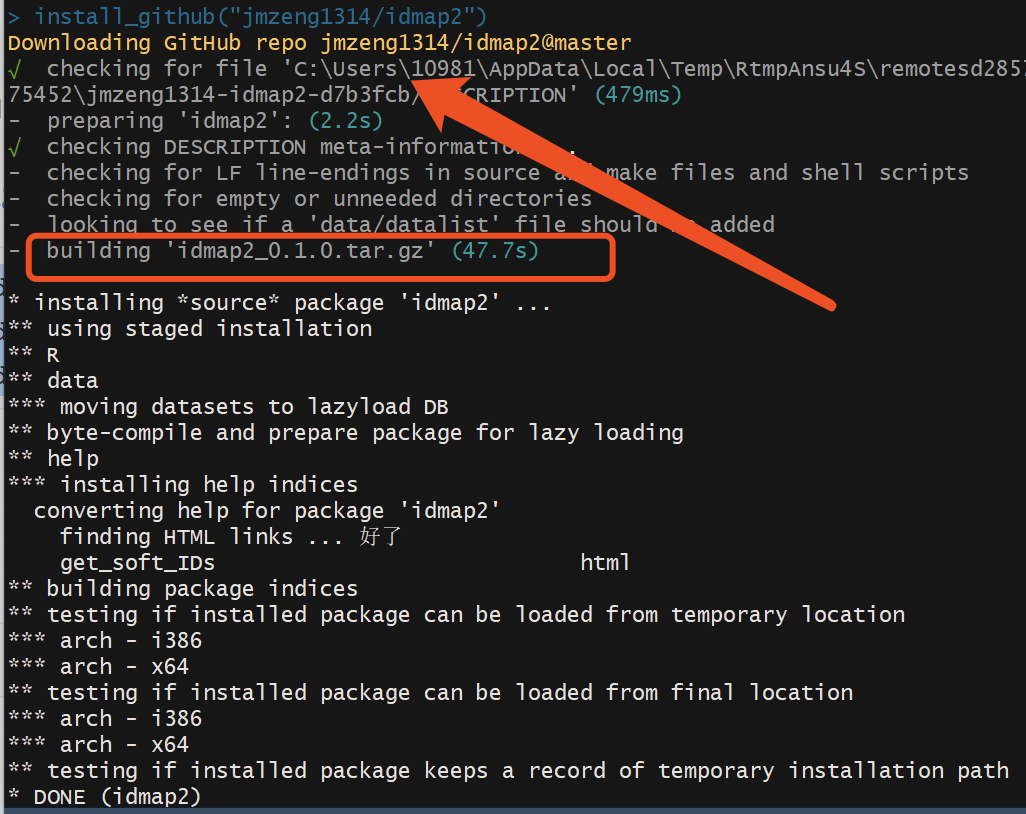
install_github("jmzeng1314/idmap2")
library(idmap2)因为有40M,所以下载安装会比较慢哦,喝杯咖啡吧!也欢迎赞助我一杯咖啡,我们一起喝!
在**大陆,大部分人访问GitHub还是很困难的,如果你成功下载并且安装,结果如下:
如果你确实无法下载, 就需要下载我的微云版本:
然后参考我以前的教程:安装GitHub的R包困难解决方案
也可以迅雷下载GitHub的我的这个项目的文件夹:
https://codeload.github.com/jmzeng1314/idmap2/legacy.tar.gz
同样的获取同样的GPL570 hgu133plus2 [HG-U133_Plus_2] Affymetrix Human Genome U133 Plus 2.0 Array注释信息,一行代码就搞定!
library(idmap1)
ids=getIDs('gpl570')
head(ids)
library(idmap2)
ids=get_soft_IDs('gpl570')
head(ids)可以看到两个包的两个函数非常类似,只需要你提供gpl即可,是不是很方便啦!
当然了,第二个包有一百多个平台,而第一个只有37个,注意哦,比如
idmap1::getIDs('GPL13912') # 失败
idmap2::get_soft_IDs('GPL13912') # 成功
这个 GPL13912 平台,就存在于第二个包,但是不在第一个哈。
你想知道我们支持哪些平台吗,当然是可以看的:
data(gpl_list)
gpl_list[,1:4]
希望你的平台,在我们的列表,能帮助你进行芯片注释。
结合我们发布的 GEO数据库**区镜像横空出世,随时随地方便下载GEO数据集,并且进行ID转换!
library(GEOmirror)
library(idmap1)
library(idmap2)
gset=geoChina('GSE31731')
gset
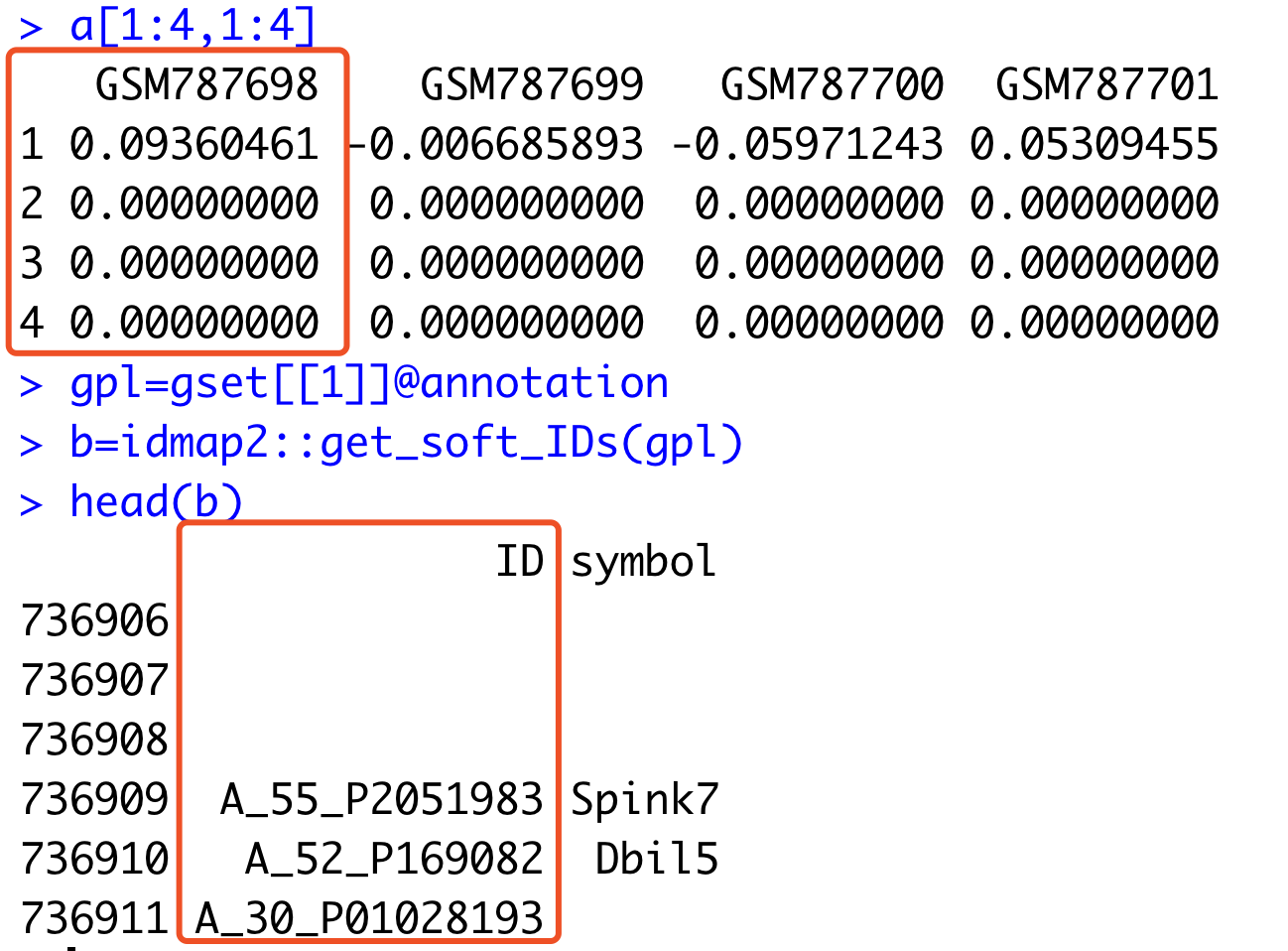
a=exprs(gset[[1]])
a[1:4,1:4]
gpl=gset[[1]]@annotation
b=idmap2::get_soft_IDs(gpl)
head(b)可以看到, 有一些表达矩阵,虽然是芯片做的,但是作者其实并不太懂,上传的时候把探针都漏掉了。不过,还是可以按照行慢慢对应过来。
强烈建议你推荐给身边的博士后以及年轻生物学PI,多一点数据认知,让他们的科研上一个台阶:
- 全国巡讲全球听(买一得五) ,你的生物信息学入门课
- 生信技能树的2019年终总结 ,你的生物信息学成长宝藏
- 2020学习主旋律,B站74小时免费教学视频为你领路