| title | author |
|---|---|
scratch test count |
Kozhin PM |
Скрипт создан для обсчета данных scratch-test`а (что это такое, можно посмотреть тут: https://www.nature.com/articles/nprot.2007.30)
С помощью класса scratch_test_class анализируются фотографии с клеточными культурами. Сегментируются участки с клетками и без. Вычисляются соответствующие площади.
import pandas as pd
import matplotlib.pyplot as plt
import czifile
import scratch_test_class as st
from skimage.segmentation import mark_boundariesST = st.Scratch_test()
ST.setImages('/pics')
ST.setTargets('0d_k')
ST.setTargetThreshold()
ST.count_scratch_area()0 of 3 images
1 of 3 images
2 of 3 images
Done!
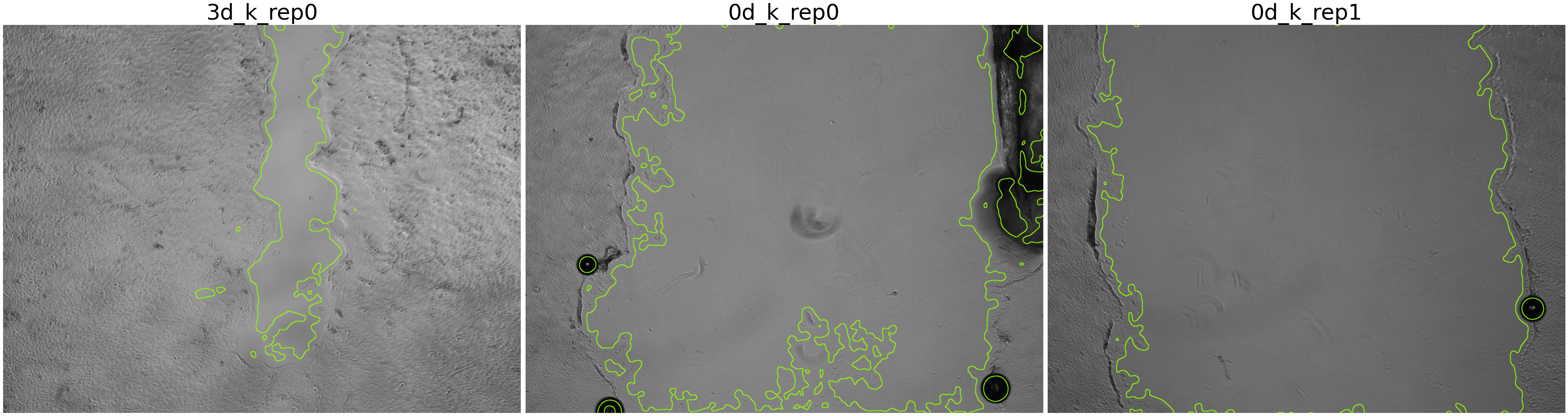
pd.DataFrame({'name': ST.imagenames, 'area': ST.area}) name area
0 3d_k_rep0 426554
1 0d_k_rep0 3200451
2 0d_k_rep1 3669344
Сегментированные площади можно подсветить наложением маски или отобразив границы
ST.display(ncols=3)fig, axes = plt.subplots(ncols=3, nrows=1, figsize=(50, 50))
for ax, b, i, title in zip(axes.flatten(), ST.binaries, ST.images, ST.imagenames):
ax.imshow(czifile.imread(i)[0,:,:,0], cmap='gray')
ax.imshow(b, cmap='Greens_r', alpha=0.5)
ax.axis('off')
ax.set_title(title, fontsize=50)
fig.tight_layout()fig, axes = plt.subplots(ncols=3, nrows=1, figsize=(50, 50))
for ax, b, i, title in zip(axes.flatten(), ST.binaries, ST.images, ST.imagenames):
ax.imshow(mark_boundaries(czifile.imread(i)[0,:,:,0],
b,
color=(0, 1, 0),
outline_color=(1, 1, 0),
mode = 'thick'))
ax.axis('off')
ax.set_title(title, fontsize=50)
fig.tight_layout()