El dataset que exploraremos es un conjunto de datos dedicado al costo del tratamiento de diferentes pacientes. No tenemos datos sobre el diagnóstico de los pacientes, pero tenemos otra información que puede ayudarnos a llegar a diferentes conclusiones utiles, como estimar los estados de salud de los pacientes, o predecir los costos de cuidados medicos.
age: Edad del beneficiario principal.sex: Sexo del contratista del seguro,female,male.bmi: Índice de masa corporal, que proporciona una comprensión del cuerpo, pesos relativamente altos o bajos en relación con la altura, índice objetivo de peso corporal (kg/m^2) utilizando la relación de altura a peso.children: Número de niños cubiertos por seguro de salud | Número de dependientes.smoker: Si es fumador.region: El área residencial del beneficiario en US,northeast,southeast,southwest,northwest.charges: Costos médicos individuales facturados por el seguro de salud.
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as snsdf = pd.read_csv('insurance.csv')rows, columns = df.shape
print( 'Tenemos {} filas y {} columnas'.format(rows, columns) )Tenemos 1338 filas y 7 columnas
df.head().dataframe tbody tr th {
vertical-align: top;
}
.dataframe thead th {
text-align: right;
}
| age | sex | bmi | children | smoker | region | charges | |
|---|---|---|---|---|---|---|---|
| 0 | 19 | female | 27.900 | 0 | yes | southwest | 16884.92400 |
| 1 | 18 | male | 33.770 | 1 | no | southeast | 1725.55230 |
| 2 | 28 | male | 33.000 | 3 | no | southeast | 4449.46200 |
| 3 | 33 | male | 22.705 | 0 | no | northwest | 21984.47061 |
| 4 | 32 | male | 28.880 | 0 | no | northwest | 3866.85520 |
df.dtypesage int64
sex object
bmi float64
children int64
smoker object
region object
charges float64
dtype: object
df.describe().dataframe tbody tr th {
vertical-align: top;
}
.dataframe thead th {
text-align: right;
}
| age | bmi | children | charges | |
|---|---|---|---|---|
| count | 1338.000000 | 1338.000000 | 1338.000000 | 1338.000000 |
| mean | 39.207025 | 30.663397 | 1.094918 | 13270.422265 |
| std | 14.049960 | 6.098187 | 1.205493 | 12110.011237 |
| min | 18.000000 | 15.960000 | 0.000000 | 1121.873900 |
| 25% | 27.000000 | 26.296250 | 0.000000 | 4740.287150 |
| 50% | 39.000000 | 30.400000 | 1.000000 | 9382.033000 |
| 75% | 51.000000 | 34.693750 | 2.000000 | 16639.912515 |
| max | 64.000000 | 53.130000 | 5.000000 | 63770.428010 |
df.mode().dataframe tbody tr th {
vertical-align: top;
}
.dataframe thead th {
text-align: right;
}
| age | sex | bmi | children | smoker | region | charges | |
|---|---|---|---|---|---|---|---|
| 0 | 18 | male | 32.3 | 0 | no | southeast | 1639.5631 |
df.median()age 39.000
bmi 30.400
children 1.000
charges 9382.033
dtype: float64
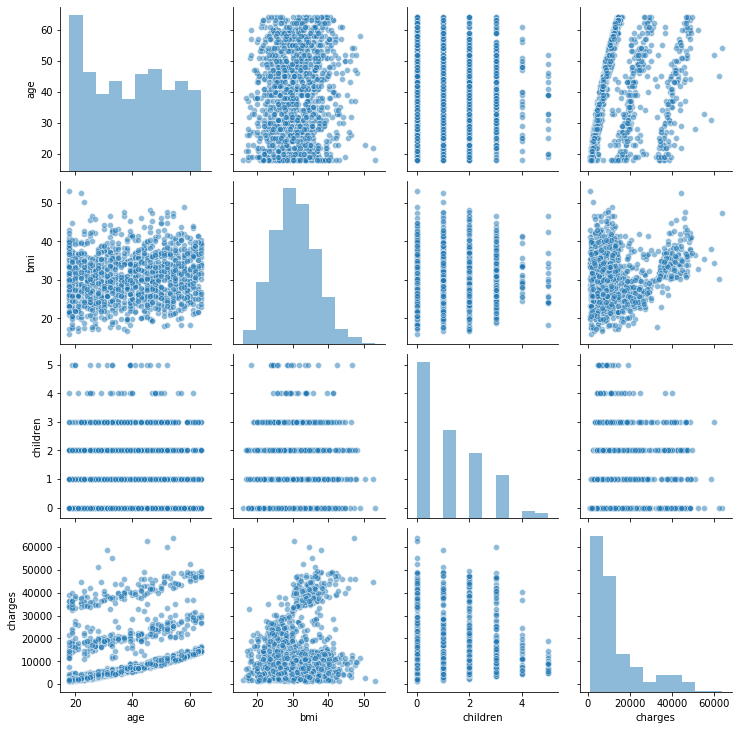
sns.pairplot(data=df,
plot_kws={'alpha': 0.5},
diag_kws={'alpha': 0.5}) <seaborn.axisgrid.PairGrid at 0x12cba6110>
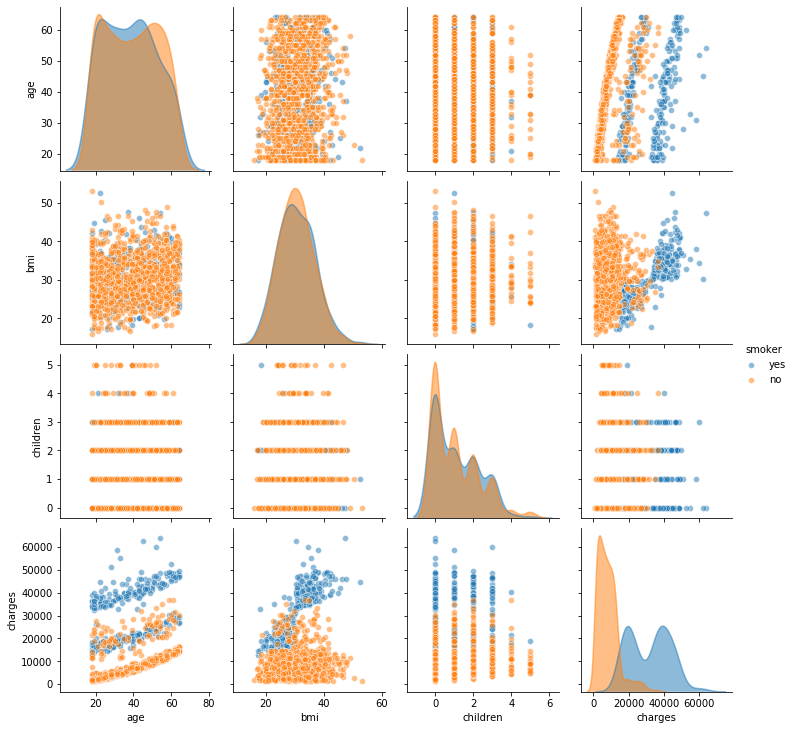
sns.pairplot(data=df,
hue="smoker",
# diag_kind="hist",
plot_kws={'alpha': 0.5},
diag_kws={'alpha': 0.5}) <seaborn.axisgrid.PairGrid at 0x12d6d8dd0>
- Que rango de edades tienen mayor densidad?
- Que rango de edades suelen fumar mas?
- Que rango de edades tienen la mayor cantidad de obesos?
- Que rando de edades tienen la mayor cantidad de hijos?
- Que rango de edades suelen tener mayor gastos medicos?
- La edad afecta los gastos medicos?
- Que genero tiene la mayor densidad?
- Que genero suele fumar mas?
- Que genero tiene la mayor cantidad de obesos?
- Que genero tiene la mayor cantidad de hijos?
- Que genero suele tener mayor gasto medico?
- El genero afecta el gasto medico?
-
Que rango de bmi tiene mayor densidad?
-
El bmi afecta el gasto medico?
-
El promedio del
BMIindica que hay un indice deObesidadalto
- Que cantidad de hijos tiene mayor densidad
- Los padres con hijos fuman?
- Densidad de fumadores
- Los fumadores tienen mayor gastos medicos?
- Que region tiene mayor densidad
- Los gastos medicos aumentan en relacion a la
edad - los gastos medicos se ven grandemente afectados por
smoker
- No tenemos datos o columnas con fechas por tanto no es necesario procesarlas
parse_dates- Se pueden considerar como
dummieslos datos de las columnassex,smoker, de manera que procesandolas pueda ser mas facil analizarlas link- Se pueden agregar mas columnas para representar la columna
BMIcolumnasdummiessegun si pertenece a los diferentes gruposunderweight,normal,overweight,obese,extremely_obeselink- Se pueden mapear los valores de la columna
smokera valoresbooleanoslink- Es posible que se encuentren datos atipicos
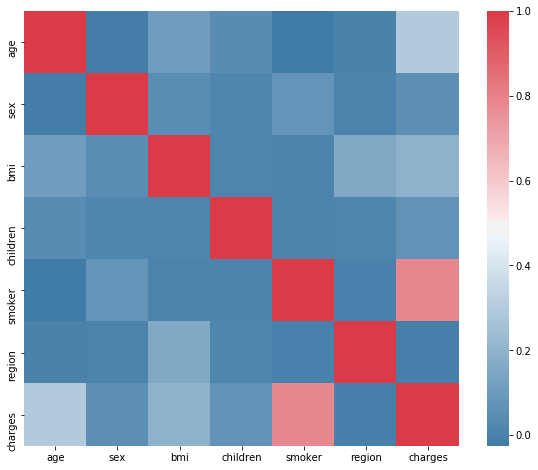
outliersen la columnaBMI- La
correlaciónno toma de manera correcta las columnassexysmokerpero se pueden codificar link- La columna
bmiactualmente es numerica pero puede ser facilmente transportada a su versión categórica solo para facilitar algunas concluciones
df.isna().sum()age 0
sex 0
bmi 0
children 0
smoker 0
region 0
charges 0
dtype: int64
A pesar que nuestro
datasetes pequeño ya esta limpia y sin valoresNaN, por lo cual no necesita que implementemos acciones especificas con los valores faltantes 🍻
Para que la correlacion nos de valores significativos en relacion a las columnas sex y smoker, las cuales podemos considerar como categoricas, las manejaremos como variables dummies y las codificaremos o mapearemos a valores enteros.
from sklearn.preprocessing import LabelEncoderle = LabelEncoder()
# SEX
le.fit(df.sex.drop_duplicates())
df.sex = le.transform(df.sex)
# SMOKER
le.fit(df.smoker.drop_duplicates())
df.smoker = le.transform(df.smoker)
# REGION
le.fit(df.region.drop_duplicates())
df.region = le.transform(df.region)
df.head().dataframe tbody tr th {
vertical-align: top;
}
.dataframe thead th {
text-align: right;
}
| age | sex | bmi | children | smoker | region | charges | |
|---|---|---|---|---|---|---|---|
| 0 | 19 | 0 | 27.900 | 0 | 1 | 3 | 16884.92400 |
| 1 | 18 | 1 | 33.770 | 1 | 0 | 2 | 1725.55230 |
| 2 | 28 | 1 | 33.000 | 3 | 0 | 2 | 4449.46200 |
| 3 | 33 | 1 | 22.705 | 0 | 0 | 1 | 21984.47061 |
| 4 | 32 | 1 | 28.880 | 0 | 0 | 1 | 3866.85520 |
- En principio iba a usar la funcion
.get_dummies()- La funcion
.map()tambien era una opcion, que me ayudaba a mapear valores usando undict, util en el caso desexsmokeryregion
plt.subplots(figsize=(10, 8))
sns.heatmap(df.corr(), cmap=sns.diverging_palette(240, 10, as_cmap=True), square=True)<matplotlib.axes._subplots.AxesSubplot at 0x12dff02d0>