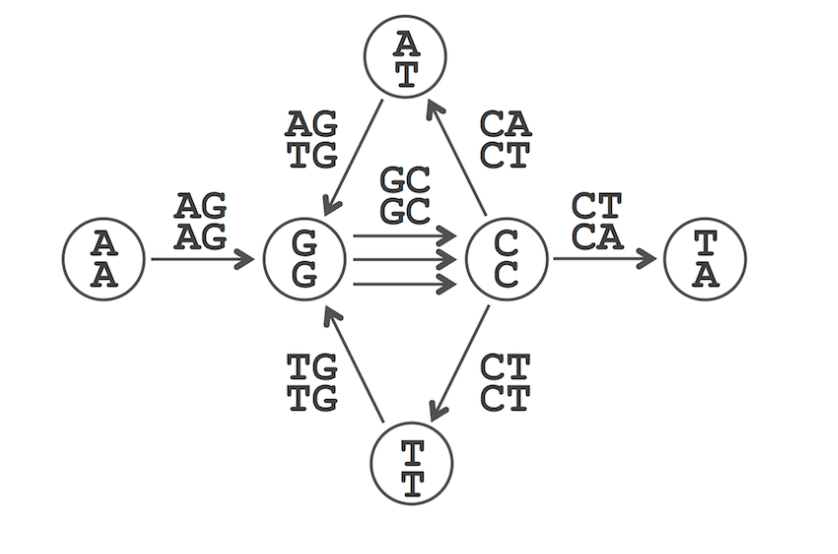
- 读取所有的reads, 按顺序打碎成k-mer,插入DBG中
- DBG每个节点(node)属性:
- kmer: DNA序列,k-mer(string)
- parents: 存储所有父节点的指针(vector)
- children:存储所有子节点的指针(vector)
- length: 从本节点出发,能寻找到的长度最长的DNA序列的长度
- coverage: 此节点经过的次数
- flag : 标记此节点的状态
- 插入节点:
- 维护一个集合(C++中set,使用红黑树实现), 标记每个k-mer是否已经插入到图中。
- 对于新k-mer,建立DBG的新节点, 其前k-mer为其父节点
- 对于插入到图中的kmer,将前k-mer作为其父节点
- 每插入或更新一个节点,递归更新父节点的length属性
- 将两个short reads文件中的所有序列及反向互补序列作为DBG原料
- 所有节点的指针存放在一个向量中维护
- 测序结果的每个碱基会在k-1个连续k-mer中出现,k为k-mer大小
- 每有一个碱基出错,这k-1个连续的碱基会偏离在原来DBG图中出现的路径,形成一小段序列,并最终合并到原序列中。
- 测序结果的每个碱基会在k-1个连续k-mer中出现,k为k-mer大小
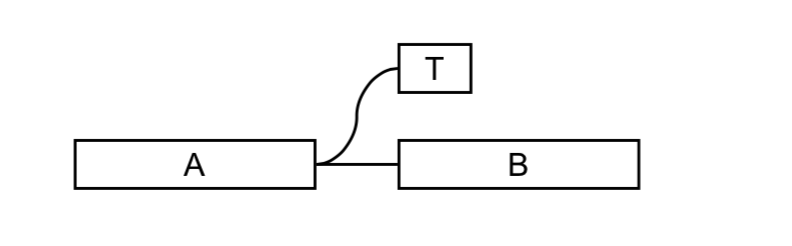
- 每有一个碱基出错,这k-1个连续的碱基会偏离在原来DBG图中出现的路径,形成一小段序列,如果错误出现在某个read的末端,则没有机会合并到原序列中,形成游离的尾端
- 长度不超过k
- 覆盖率很低
- 与对应正常节点相差一个碱基
- 遍历所有节点
- 若节点NODE有两个及以上子节点,则对每对子节点:
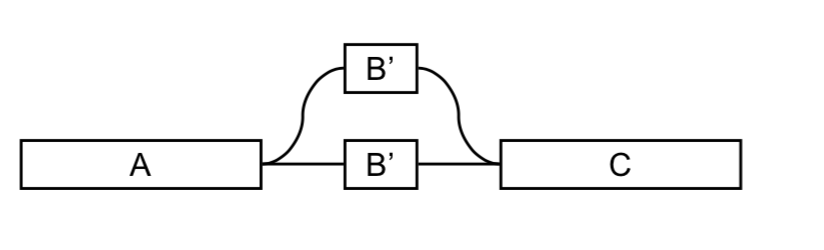
- 判断相似度,若相似度很高,则有一个节点在bubble或tip中
- 判断两个节点的coverage,较小的一个节点在bubble或tips上,将其合并到正常节点中,标记为出错节点
- 合并节点时,将两个节点的coverage相加
- 直到合并为一个节点或者剩余节点相似度很低时,停止合并
- 相似度判断方法:
- 查看两个节点的kmer对应位置不相同碱基个数,不相同碱基个数越多,相似度越低
- 原因:根据tips和bubbles的特点,与其对应的正常节点往往只相差一个碱基
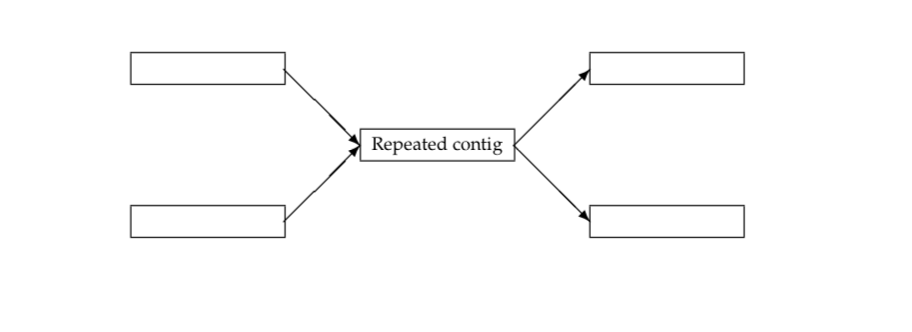
##### 方法A + 查看节点的coverage,若大于阈值COVERAGE(测序重复次数*1.7), 则将此节点拆分 + 拆分方法: - 对每个父节点,复制节点一次得到新节点,新复制的节点同此节点建立唯一的对应关系 - 每个新节点的COVERAGE继承其父节点的COVERAGE。 - 如果有多个子节点,则将父节按coverage排序,将每个子节点与其coverage最接近的分支相连 - 如果只有一个子节点,对子节点进行递归解旋
- 在寻找路径节点时,对于coverage明显高于序列平均覆盖率的节点,只降低coverage,不删除节点。直到coverage与覆盖率差不多的时,经过此节点时,将其删除。
- 由于不能将分支一一对应,所以最终实现后这种方法效果较差,最终采用了方法A
-
寻找所有不为1-入度1-出度的顶点,从这些节点出发,遇到分支或端点便停止搜索。获得一个片段,并将其从图中删除
-
剩余的节点只可能是独立的环,将这些环展开成一个路径,获得一个片段。
-
将所有片段转化成DNA单链输出
MaximalNonBranchingPaths(Graph)
Paths ← empty list
for each node v in Graph
if v is not a 1-in-1-out node
if out(v) > 0
for each outgoing edge (v, w) from v
NonBranchingPath ← the path consisting of the single edge (v, w)
while w is a 1-in-1-out node
extend NonBranchingPath by the outgoing edge (w, u) from w
w ← u
add NonBranchingPath to the set Paths
for each isolated cycle in Graph
add Cycle to Paths
return Paths
###### B 方法二:按深度依次输出路径 + 使用C++数据结构map存放节点,自定义键值比较函数,利用其红黑树的性质,使其按照length排序 + 当map非空: - 从length最大的节点开始寻找一条最长路径 - 将所有经过的路径节点从DBG图中删除 - 每删除一个节点,递归更新其父节点的length,并调整其在map中存储的位置 + 将所有片段转化成DNA单链输出
- 方法一能够输出错误率很低的基因序列片段,但输出基因序列长度较低
- 方法二虽然错误率略高,但能输出较长片段
- 最终提交结果采用了方法二
- Genome_Fraction(%): 99.772
- Duplication ratio: 1.833
- NGA50: 9123.2
- Misassemblies: 1.0
- Mismatches per 100kbp: 94.242
- Genome_Fraction(%): 99.86
- Duplication ratio: 1.8818
- NGA50: 7198.2
- Misassemblies: 1.0
- Mismatches per 100kbp: 0
- Genome_Fraction(%): 98.23
- Duplication ratio: 1.9608
- NGA50: 6406.6
- Misassemblies: 0.0
- Mismatches per 100kbp: 0
- Genome_Fraction(%): 97.017
- Duplication ratio: 1.955
- NGA50: 15153.8
- Misassemblies: 7.0
- Mismatches per 100kbp: 0
- Duplication ratio一直在2.0左右,原因是构建DBG图时,将每个genome和其反向互补序列同时输出。
- 对于data3~data4,拼出的平均长度较短(NGA50较低)
- 后续改进方案:
- 建立节点时,将反向互补序列建立在一个节点,重复率可以大大降低
- 应使用pair-end连接拼接好的序列,提高平均长度
- configs.h: 定义data1~data4会使用到的宏变量
- basic_functions.h: 基本函数,如输入输出,取反向互补序列等
- node.h: 定义DBG图节点及其操作
- dbg.h: 定义DBG图的各种操作函数
- main.cpp: 主函数
说明:由于最开始完成data1时,构建DBG图并没有很好的架构,导致处理测序错误时,程序冗长且很难维护。 因此完成剩余任务时,使用C++重构了代码(A部分)
- basic_functions.py :基本函数,如输入输出,取反向互补序列等
- dbg.py: 定义DBG图及各种操作
- data1.py: 使用以上两个问题,解决data1
- C++: Xcode Version 9.4.1 (9F2000)
- python: python3.6
- 代码测试时,需要将data1~data4放到当前目录
- cousera慕课: Genome Sequencing (Bioinformatics II)第二周
- cousera慕课:DNA序列组装
- cousera慕课:离散数学
- 《一种DNA测序纠错算法》,郑纬民, 张 华, 王小川 (ISSN 1000-9825, CODEN RUXUEW Journal of Software, Vol.17, No.2, February 2006, pp.193−199)
- 基于WGS和CBC测序策略的DNA序列拼接算法研究
- 简单DNA序列组装(非循环子图)
- DNA序列组装(贪婪算法)
- Hybrid error correction and de novo assembly of single-molecule sequencing reads
- 单分子测序reads(PB)的混合纠错和denovo组装
- 每日一生信--序列组装的算法k-mer
- 基因组组装算法
- 感谢hzshang,提供了:
- 处理bubbles与tips时合并节点的实现思路
- De brujin图C语言实现,定义节点的方法