İlk önce fastqc'nin ne olduğuna, daha sonra da ubuntu üzerinden bu farmatı nasıl çalıştırdığımıza bakacağız. Öyleyse işimize fastqc'nin ne olduğuna ve ne işe yaradığını kısaca anlatarak başlayalım.
Fastqc kısaca daha önce elde edilmiş olan dizinin (DNA ya da RNA) kalite kontrolünü yapar. NGS ve Sanger yöntemlerinde sıklıkla kullanılır.
Fastqc'nin temel amacı, veri setlerindeki olası kalite sorunlarını tespit etmektir. Bu; örneğin baz çifti kalitesi, dizi uyumsuzlukları, adaptör kirliliği, düşük kaliteli bazlar ve diğer potansiyel problemleri belirleyerek gerçekleştirilir. Analizin sonuçları, kullanıcıya veri setinde hangi bölgelerin sorunlu olduğunu ve verilerin ne kadar güvenilir olduğunu anlamasına yardımcı olur.
İlk önce Veri_Dizileme adlı bir klasörümüz olsun. Şimdi cd Veri_Dizileme komutu ile bu klasörümüzün içine girelim. Bu klasörün içinde de cutadapt.sh, fastqc.sh, bowtie2.sh gibi dosyalar ve envs, scripts gibi klasörler olsun. Daha sonra dizilime metotları sonucu elde edilen genom dizisi ve referans genom dizisini içeren data klasörünü Veri_Dizileme adlı klasörümüze kopyalayalım. Şimdi;
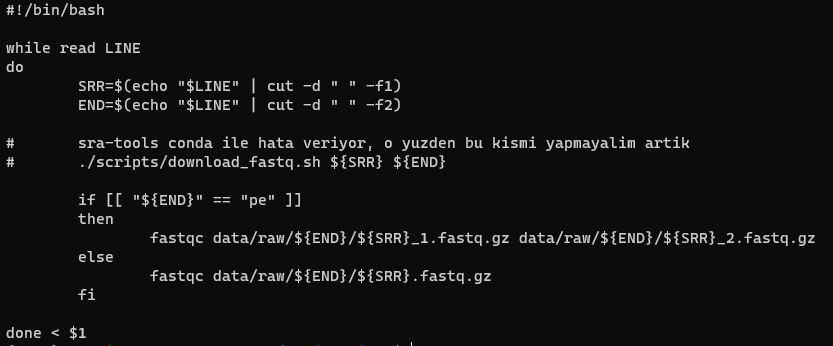
cat fastqc.shkomutu ile fastqc.sh dosyamızı açalım. Bu sayede aşağıdaki görselde bulunan komutlar çıkacaktır:
Bu komutlar sayesinde fastqc formatımız bize uygun bir yol çizerek az sonra anlatacğım şekilde çalışarak okuma kaitesini test etmeye yarayacak.
Şimdi komut satırımıza;
conda activate rnaseqyazalım. Bu sayede rnaseq'i aktifleştirip bununla işlemlerimize devam edeceğiz. Artık dizimizi okumaya başlayabiliriz. Öyleyse komut satırına:
./fastqc.sh data.txtyazalım. Ve komutumuz ile data.txt nin içinde bulunan ERR3079326 pe dizimiz okunacaktır. Sonuç olarak oluşturulan results klasörünün içine html dosyaları şeklinde kaydedilecektir.
Cutadapt, yüksek verimli diziliminizde arama yaparak primerleri, poly-A kuyruklarını, adaptör dizilerini ve diğer istenmeyen dizileri okur ve ortadan kaldırır [martin_cutadapt_2011]. Böylece, sonraki analizlerde daha temiz ve daha güvenilir sonuçlar elde etmek için veri setinin kalitesini artırır. Adaptör kaldırma işlemi, veri setinin işlenmesi sırasında sıralama hatalarını azaltabilir ve hedef dizilerin daha doğru bir şekilde belirlenmesine olanak tanır.
Cutadapt, bu düzeltme işleri için hataya dayanıklı primer dizisi veya adaptör bulma yardımı sağlar. Tek uçlu ve çift uçlu okumalar da çeşitli şekillerde değiştirilebilir ve filtrelenebilir. Cutadapt aynı zamanda okumaların çoğunu da çözebilir [martin_cutadapt_2011]
Hatırlarsanız daha önce Veri_Dizileme adlı bir klasör oluşturmuştuk. ve klasörün içinde cutadapt.sh, fastqc.sh, bowtie2.sh gibi dosyaların olduğunu da söylemişitk. Az önce fasqc ile kalite analizini yaptık. Şimdi de hazırsanız işimize yaramayan adabtörlerimizi kesmeye başlayalım. Şimdi,
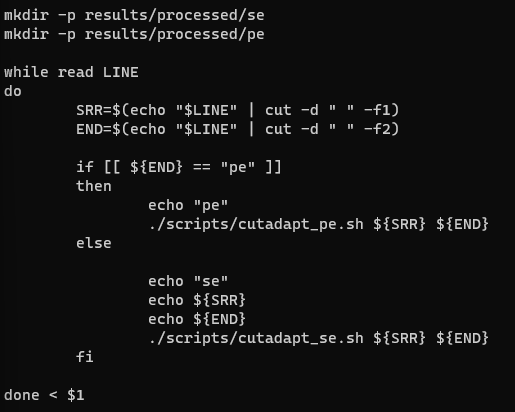
cat cutadapt.shkomutunu komut satırına yazdığımız zaman aşağıdaki görselde bulunan komutlar çıkacaktır.
Peki bu görselde bulunan komutlar bize ne anlatıyor? Bu,aslında adabtörleri kesmek için kullanmış olduğumuz bir pathway. Yani diyoruz ki kesme işlemini yap ve bunları results klasörümüzün içinde bulunan processed klaösrüne kaydet. Öyleyse adaptörlerimizi kesmeye başlayalım. Komut satırına;
./cutadabt.sh data.txtkomutunu yazarsak. komutumuzu vermiş oluruz ve sonuç olarak results kısmında bulunan processed klasörünnde html dosyaları şeklinde kaydedilecektir.
Bowtie2, ultra hızlı bir eşleştirme algoritmasına sahiptir ve genellikle milyonlarca kısa DNA veya RNA dizisini çok hızlı bir şekilde referans genomlara eşleştirmek için kullanılır. Bu, genetik varyantları tespit etmek, transkriptomik analizler yapmak, genomlardaki değişiklikleri belirlemek ve diğer genetik araştırmalarda önemli bir adımdır [noauthor_bowtie_nodate].
Benzer boşluk cezaları ile boşluklu hizalama tamamen Bowtie 2 tarafından desteklenir. Kullanıcı tarafından sağlanan puanlama şeması dışında boşluk sayısı veya boşluk uzunlukları konusunda herhangi bir kısıtlama yoktur. bowtie1 tarafından yalnızca aralıksız hizalamalar algılanır. (With affine gap penalties, gapped alignment is completely supported by Bowtie 2. Apart from the user-supplied scoring scheme, there are no restrictions on the number of gaps or gap lengths. Only ungapped alignments are detected by Bowtie 1.)
Genel olarak Bowtie 2 daha hızlıdır, daha hassastır ve kabaca 50 bp'den daha uzun okumalar için Bowtie 1'e göre daha az bellek gerektirir. Nispeten kısa okumalarda (50 bp'den az), Bowtie 1 bazen daha hassas veya daha hızlı olabilir [langmead_scaling_2019].
Hatırlarsanız daha önce Veri_Dizileme adlı bir klasör oluşturmuştuk. ve klasörün içinde cutadapt.sh, fastqc.sh, bowtie2.sh gibi dosyaların olduğunu da söylemişitk. ilk önce fastqc ile kalite kontrolü yaptık, daha sonra cutadapt ile adabtörleri kestik. Şimdi de bunları hizalamaya başlayacağız. Bunun için komut satırına;
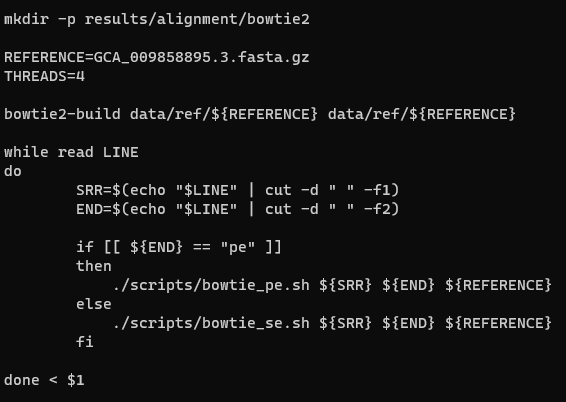
cat bowtie2.shyazalım. Bunun çıktısı olarak aşağıdaki görselde bulunan yolak oluşacaktır.
Tahmin edeceğiniz gibi yukarıdaki görselde anlatmaya çalıştığımız şey bu dizimizi hizala ve hizlama sonucu result klasörümüzün içine alignment adlı bir klasör oluştur ve sonuçlarımızı oraya kaydet. Öyleyse komut satırına;
/.bowtie2.sh data.txtkomutumuzu veriyoruz ve programımızı çalıştırıyoruz. Ve sonuç olarak results klasörümüzü açtığımız zaman hizlama sonucu oluşan dosyalarımız alignment adlı klasörümüzde kaydedilmiş olduğunu göreceğiz.
Üzerinde durmuş olduğumuz konu DNA/RNA dizillimini biyoinformatik bilgisi olarak öğrenmeye çalıştık. Bunun için biyoinformatikte kullanılan bazı metotlar/formatlar üzerinde durduk. Bu formatlar ile bazı işlemleri yerine getirmeye çalıştık. Mesela fastqc formatını komut satırı kullanarak elde edilen genom dizisinin kalite kontrolünü yaptık. Analizin sonuçları, kullanıcıya veri setinde hangi bölgelerin sorunlu olduğunu ve verilerin ne kadar güvenilir olduğunu anlamasına yardımcı olur. Daha sonra cutadapt formatı ile istenmeyen dizileri ortadan kaldırdık. Son olarak bowtie2 formatı ile dizimiz için uygun olan hizalamarı yaptık.
Bu çalışmalar; hastalık araştırmaları ve tanısı, genetik çeşitliliğin anlaşılması, Genomik tıp ve tedavisi gibi insan sağlığını etkileyen alanlarla birlikte Tarımda ve çevresel uygulamarda adeta bir tosetta taşı olmuştur.