使用 paddleOCR 的预训练模型,即使不用fine-tune,也已达到非常好的识别效果。
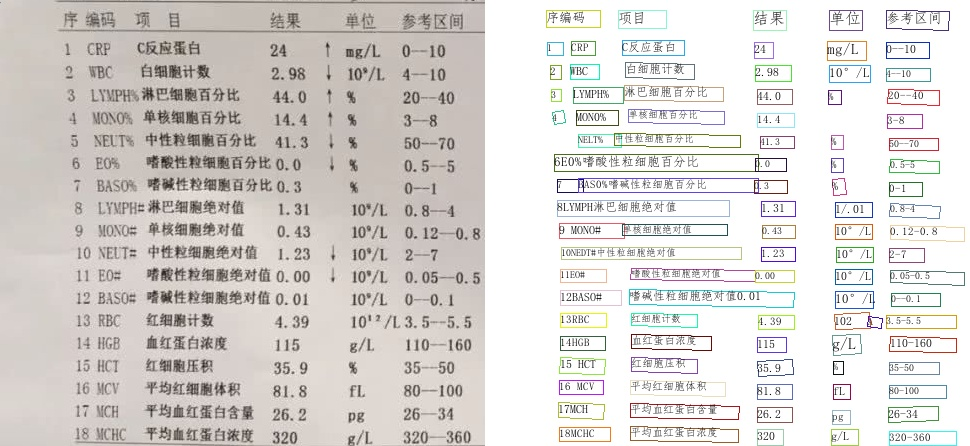
但在实际应用时候,特别是面对一些表格制式的特殊图像(如下图示的医学检验报告,来源于百度图片),我们paddleOCR 并不会按行给出结果,而是按块给出。这需要我们按行合并块,以得到可以阅读的识别文本。
paddleOCR 同样开源了 PP-Structure 用于表格、图片以及列表的识别,效果可自行测试
序编码
项目
结果
单位
参考区间
CRP
C反应蛋白
1
24
mg/L
0--10
WBC
2
白细胞计数
2.98
10°/L
4--10
LYMPH%
3
淋巴细胞百分比...
基于每个表格块的四角坐标做行对应
-
以上图为例,先 sort 每个块的 x 坐标可以得到:
【1】【序编码】【CRP】【项目】【C反应蛋白】【24】【结果】【mg/L】【单位】【0-10】【参考区间】
-
记【1】为当前块,往后遍历,只要满足两个矩形块相交(实际代码中加了更多控制条件),便判断为1行
-
以 y 坐标重新排序每行结果,输出
序编码项目 结果 单位 参考区间
1 CRP C反应蛋白 24 mg/L 0--10
2 WBC 白细胞计数 2.98 10°/L 4--10
3 LYMPH% 淋巴细胞百分比 44.0 % 20--40
4 MON0% 单核细胞百分比 14.4 3-8
NELT%中性粒细胞百分比 41.3 % 50--70
6 E0%嗜酸性粒细胞百分比0.0 % 0.5-5
7 BAS0%嗜碱性粒细胞百分比 0.3 % 0-1
8 LYMPH淋巴细胞绝对值1.31 1/.01 0.8-4
9 MONO#单核细胞绝对值 0.43 10°/L 0.12-0.8
10 NEDT#中性粒细胞绝对值1.23 10°/L 2-7
11 EO#嗜酸性粒细胞绝对值 0.00 10°/L 0.05-0.5
12 BASO#嗜碱性粒细胞绝对值0.01 10°/L 0--0.1
13 RBC红细胞计数 4.39 102 A 3.5-5.5
14 HGB血红蛋白浓度 115 g/L 110-160
15 HCT红细胞压积 35.9 % 35-50
16 MCV平均红细胞体积 81.8 fL 80-100
17 MCH平均血红蛋白含量 26.2 pg 26-34
18 MCHC平均血红蛋白浓度 320 g/L 320-360
- interval==1.0.0
- paddleocr==2.0.6
python ocr_align.py