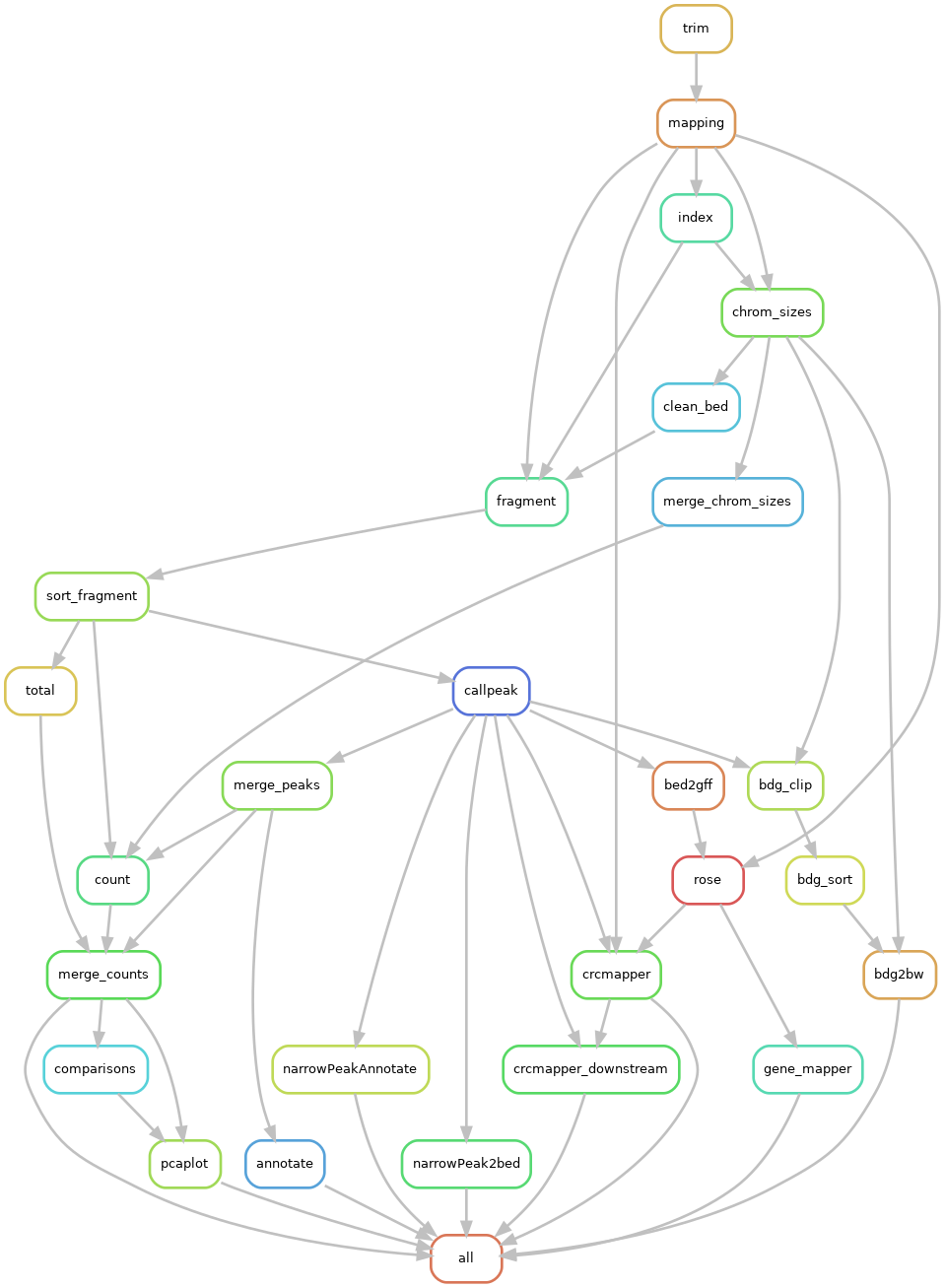
depipe 是一个自动化的 ChIP-Seq 分析流程,在完成配置后,可以自动进行可复现、高并行的 ChIP-Seq 数据分析。
使用 mamba 自动安装所有依赖
mamba env create -f environment.yml
conda activate depipe首先需要创建一个本次分析的配置文件
mkdir project
cd project
cp dapipe/config.yaml .浏览检查配置文件内的所有配置项(所有配置项都是必须的),并按照注释进行更改。然后运行流程
snakemake --snakefile dapipe/Snakefile -j 10 --configfile config.yaml -- all其中的 -j 是允许流程使用的最大进程数。注意最后的位置参数 all,使用这个 rule 代表运行所有的流程,包括富集差异、超级增强子以及核心转录因子。可选的流程有 basic_all 仅进行 peak calling;compare_all 进行富集差异分析;rose_all 分析超级增强子了;crcmapper_all 分析核心转录因子。不同的流程可以相互组合。