该snakemake流程主要是封装了一系列的AWK和Bedtools操作,用于对Gene body区域进化窗口划分.
通过将划分好的窗口与任意特征所在的窗口取交集,就可以绘制特征在基因周围的分布图
例如常见的一些特征:
- 基因甲基化程度CG、CHG和CHH
- DHS染色质开发窗口
- eQTL 区间
- 等等
该pipline基于snakemake v7.25.0以及Bedtools实现,因此运行该pipline时,需要将Bedtools加入到当前环境变量中。
由于该pipline在每个rule中没有加入resource和threads等设置信息,如果脚本出错很有可能是内存不足的原因,可以手动的在每个rule中对相应的内容进行指定
config/config.yaml 文件是包含以下配置信息; 关于该流程的测试数据可以查看test 目录
geneBed: 基因的Bed文件Chrlength: 染色体长度文件,为Samtools faidx输出结果outPutPrefix: 输出文件前缀,输出的主要是gene侧翼和body区域划分好的窗口params: 划分窗口的参数设置geneWindowCount: gene body区域划分为多少个窗口flankLength: 选取多少长度范围的侧翼序列flankWindowCount对应的侧翼序列划分为多少个窗口
将指定基因及其周围序列划分为对应的Bed文件, 划分后的结果文件在config/config.yaml中进行了指定。
#TODO 运行整个流程
cd GWD
snakemake --cores all
将划分好的窗口文件与甲基化特征取交集
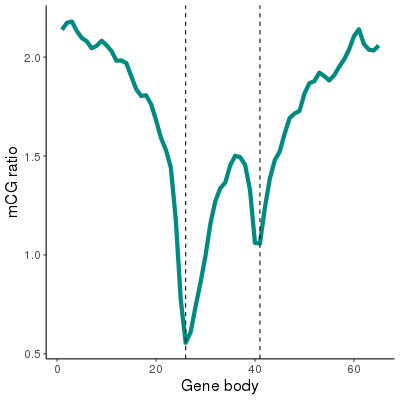
#TODO 获得CG甲基化在Gene body区域的分布
zcat test/CG.txt.gz |intersectBed -a results/test_windows_Id.bed -b - -loj |gzip >results/test.CG.txt.gz 可视化脚本在
plot.ipynb中