项目简介:
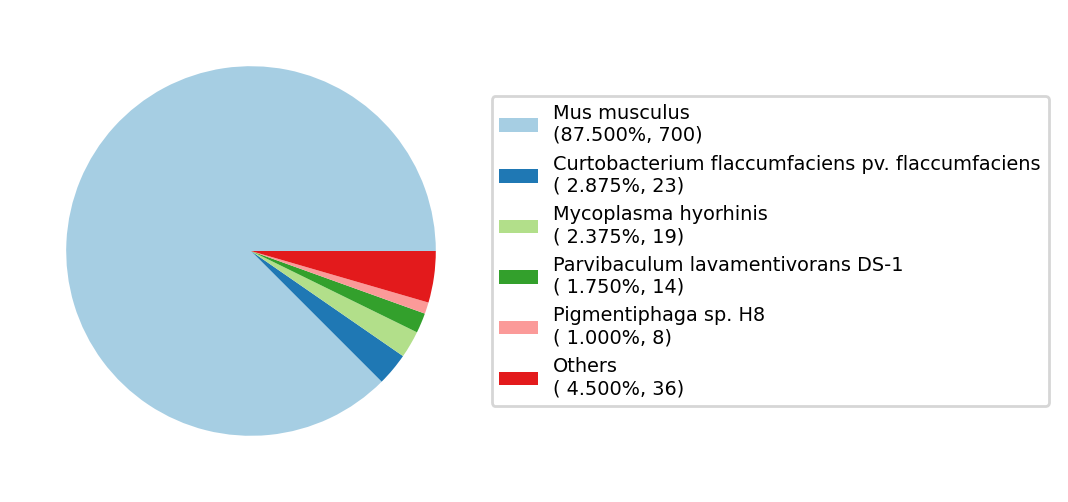
- 建立本地 blast nt 数据库,进行随机抽取1000条reads,进行nt比对统计物种的含量并绘制饼图。
- 该项目主要奉献了脚本用于nt数据库id查找名的优化,为遍历大文件查找提供了一种最有效率的方式。
- 实现**:将大文件先排序,然后分割成各个小文件库,在查找时,按照一定规律按需去导入各个小文件库进行查找,从而大大降低资源消耗。
测试效果:
time ./map_taxid.py ./test/test.blast
# real 0m12.926s
# user 0m9.968s
# sys 0m2.923s
time {
db=/home/chenjun/dataBase/blast_db_FASTA/
cat test.blast | sort -k1,1 -u >test.filter.blast
cat test.filter.blast |cut -f 2 >keys.txt
# 下面这一步占了99%的时间,为1m20s
awk 'NR==FNR{a[$1]=$2;next}NR!=FNR{if($1 in a)print $0}' keys.txt ${db}/db_NCBI_Taxonomy/nucl_gb.accession2taxid.filter.sorted.txt >find.txt
cut -f 2 find.txt >find.txt.col1
awk 'NR==FNR{a[$1]=$0;next}NR!=FNR{if($1 in a)print a[$0]}' ${db}/db_NCBI_Taxonomy/taxdump/names.dmp.db find.txt.col1 >find_res.xls
# \rm test.filter.blast keys.txt find.txt.col1
}
# real 1m22.207s
# user 1m20.908s
# sys 0m0.939s构建代码:
db=$PWD
## 1.环境配置
# blast, numpy, pandas, matplotlib, pickle ... 安装
# 尽量使用官方源,一般具有更新的环境版本不易出现问题。访问缓慢可借助 proxychains4 解决。
conda deactivate; conda deactivate;
conda create -y -n nt -c bioconda blast libgcc numpy pandas matplotlib seqtk
# blast版本地址:<https://ftp.ncbi.nlm.nih.gov/blast/executables/LATEST/>
# blast使用手册《BLAST Command Line Applications User Manual》
## 2.下载所需数据库
# 下载blast的数据库,nt为核酸数据库,nr为蛋白质数据库。以及物种信息对应的数据库,NCBI Taxonomy 数据库下载。
# https://ftp.ncbi.nih.gov/blast/db/FASTA/
wget -c -t 0 https://ftp.ncbi.nih.gov/blast/db/FASTA/nt.gz
wget -c -t 0 https://ftp.ncbi.nlm.nih.gov/pub/taxonomy/accession2taxid/nucl_gb.accession2taxid.gz -O db_NCBI_Taxonomy/
wget -c -t 0 https://ftp.ncbi.nlm.nih.gov/pub/taxonomy/taxdump.tar.gz -O db_NCBI_Taxonomy
# 构建nt数据库
zcat nt.gz >nt
makeblastdb -in nt -parse_seqids -hash_index -dbtype nucl -logfile nt_logfile
# 提取Taxonomy,处理物种对应信息, taxid必须排序
cd db_NCBI_Taxonomy
zcat nucl_gb.accession2taxid.gz | cut -f 1,3|sed 1d|sort -k1 > nucl_gb.accession2taxid.filter.sorted.txt
tar xzvf taxdump.tar.gz
cd -
# cd taxdump
# grep "scientific name" names.dmp >names.dmp.db
## 3. 构建搜索ID用的pydb, map_taxid.py中的pydb路径需要自己指定
conda activate nt
# blast 略,需要格式参数-outfmt 6
sed -i "s#/home/chenjun/dataBase/blast_db_FASTA#$db#" map_taxid.py
python3 map_taxid.py test/test.blast分步执行:
db=/home/chenjun/dataBase/blast_db_FASTA/nt
conda activate nt
fastq=test.fq.gz
num=1000
fasta=test.fa
out=test.blast
# 筛选1000 条 reads 准备进行 blast nt 比对
./get_1000reads.sh
seqtk sample -s 100 $fastq ${num} >${num}reads_${fastq_name}
cat ${num}reads_${fastq_name} | awk '{if(NR%4 == 1){print ">" substr($0, 2)}}{if(NR%4 == 2){print}}' >$fasta
# blast执行
time blastn -query $fasta -out $out -outfmt 6 -db $db -num_threads 10 -evalue 1e-5 -qcov_hsp_perc 50.0 -num_alignments 5
# blast结果的物种查找
time python3 map_taxid.py $out一步执行:
conda activate nt
./fq-nt-check.sh ./test.fq.gz饼图结果:
Taxonomy数据库格式说明: (一般blast比对后的结果为第1/2列所示,我们需要通过1/2列找到对应3列,然后在names.dmp去找到对应物种名并进行统计)
# nucl_gb.accession2taxid.gz
# 第一列Accession : 序列标识码
# 第一列Accession.version : 带版本号的序列标识码
# 第三列: 序列的taxid 号,即物种分类号。如 Homo sapiens 的是9606.
# 第四列:序列的gi号
# accession accession.version taxid gi
# A00001 A00001.1 10641 58418
# A00002 A00002.1 9913 2
# A00003 A00003.1 9913 3
# A00004 A00004.1 32630 57971
# A00005 A00005.1 32630 57972
# A00006 A00006.1 32630 57973
# A00008 A00008.1 32630 57974
# A00009 A00009.1 32630 57975
# A00010 A00010.1 32630 57976
# taxdump.tar.gz 解压后会有7个库,我们用 names.dmp
# names.dmp 中包含物种的taxid号与物种学名。对应的格式如下:
# 第1列为 物种的taxid号。
# 第2列为物种名称。
# 第3列为物种名称及类
# 第4列为该行的信息类型。我们后面会选择 scientific name 信息的行,对应的物种学名。
# 1 | all | | synonym |
# 1 | root | | scientific name |
# 2 | Bacteria | Bacteria <bacteria> | scientific name |
# 2 | bacteria | | blast name |
# 2 | eubacteria | | genbank common name |
# 2 | Monera | Monera <bacteria> | in-part |
# 2 | Procaryotae | Procaryotae <bacteria> | in-part |
# 2 | Prokaryotae | Prokaryotae <bacteria> | in-part |
# 2 | Prokaryota | Prokaryota <bacteria> | in-part |
# 2 | prokaryote | prokaryote <bacteria> | in-part |
blast-nt库的构建参考文章: